Abstract
Introduzione
L’epitelio del tratto gastrointestinale (IG) rappresenta un’ampia superficie per l’interazione ospite-microbo e media l’equilibrio tra la tolleranza degli organismi mutualistici e l’esclusione di potenziali agenti patogeni(Peterson e Artis, 2014). Ciò avviene, in parte, attraverso la formazione di una stretta barriera fisica epiteliale, oltre alla secrezione epiteliale di peptidi antimicrobici e muco(Veereman-Wauters, 1996; Renz et al., 2011). Lo sviluppo e la maturazione della barriera epiteliale coincide con la prima esposizione del tratto gastrointestinale ai microrganismi e la creazione di una comunità microbica all’interno dell’intestino(Palmer et al., 2007; Koenig et al., 2011). Sebbene i microrganismi siano stati a lungo apprezzati come i principali motori dell’espansione postnatale dell’immunità adattativa(Renz et al., 2011; Shaw et al., 2010; Hviid et al., 2011; Abrahamsson et al., 2014; Arrieta et al., 2015), e più recentemente come stimoli chiave nello sviluppo della digestione(Erkosar et al.,2015), del metabolismo(Cho et al., 2012) e della funzione neurocognitiva(Diaz Heijtz et al., 2011; Clarke et al., 2014; Borre et al., 2014; Desbonnet et al., 2014), non è ancora chiaro come la superficie epiteliale umana si adatti alla colonizzazione e all’espansione dei microrganismi all’interno del tratto GI immaturo.
Gli studi sui topi gnotobiotici hanno migliorato la nostra comprensione dell’importanza dei microbi nella normale funzione intestinale, poiché questi topi mostrano profondi difetti di sviluppo nell’intestino(Round and Mazmanian, 2009; Gensollen et al., 2016; Bry et al, 1996; Hooper et al., 1999), tra cui la diminuzione del turnover epiteliale, la ridotta formazione di microvilli(Abrams et al., 1963) e l’alterazione della glicosilazione del muco sulla superficie epiteliale(Bry et al., 1996; Goto et al., 2014; Cash et al., 2006). Tuttavia, l’evidenza suggerisce anche che l’intestino umano immaturo può differire significativamente dall’intestino murino, specialmente nel contesto della malattia(Nguyen et al., 2015). Ad esempio, i neonati prematuri possono sviluppare un’enterocolite necrotizzante (NEC), una malattia infiammatoria di cause sconosciute. Recenti rapporti suggeriscono un’eziologia multifattoriale secondo la quale la funzione di barriera intestinale immaturo predispone il neonato pretermine a lesioni intestinali e infiammazioni in seguito a colonizzazione microbica post-partum(Neu and Walker, 2011; Morrow et al., 2013; Greenwood et al., 2014; Hackam et al., 2013; Afrazi et al., 2014; Fusunyan et al., 2001; Nanthakumar et al., 2011). I modelli di roditori di NEC si sono rivelati dei surrogati inadeguati per lo studio delle malattie umane(Tanner et al., 2015). Pertanto, gli studi diretti delle interazioni medico-microbiche nell’intestino umano immaturo saranno importanti per comprendere le complesse interazioni durante la colonizzazione batterica che portano ad un normale sviluppo intestinale o ad una malattia.
Importanti considerazioni etiche e pratiche hanno limitato la ricerca sull’intestino umano immaturo. Ad esempio, i campioni chirurgici neonatali sono spesso gravemente danneggiati dalla malattia e non sono adatti per gli studi ex vivo. Noi e altri abbiamo già dimostrato in precedenza che gli organi intestinali umani pluripotenti derivati da cellule staminali umane (HIO) assomigliano strettamente al tessuto intestinale immaturo(Spence et al., 2011; Finkbeiner et al., 2015; Watson et al., 2014; Forster et al., 2014; Dedhia et al., 2016; Aurora e Spence, 2016; Chin et al., 2017) e lavori recenti hanno stabilito che gli organoidi gastrointestinali sono un potente modello di patogenesi microbica all’interfaccia mucosa(Leslie et al., 2015; McCracken et al., 2014; Forbester et al., 2015; Hill e Spence, 2017).
Nel lavoro attuale, abbiamo usato gli HIO come epitelio intestinale immaturo modello e un ceppo umano non patogeno di E. coli come colonizzatore intestinale modello per esaminare come le interazioni ospite-microbico influiscono sulla maturazione e la funzione intestinale. Sebbene la composizione del microbioma intestinale neonatale varia da individuo a individuo, gli organismi dei generi Escherichia sono colonizzatori precoci dominanti(Gosalbes et al., 2013; Bäckhed et al., 2015) e l’E. coli non patogeno sono componenti ampiamente prevalenti e molto abbondanti del microbioma delle feci neonatali (Palmer et al.,2007; Koenig et al., 2011; Bäckhed et al., 2015; Morrow et al., 2013). La microiniezione di E. coli nel lume di HIO tridimensionale ha portato alla colonizzazione batterica stabile in vitro e, utilizzando il sequenziamento dell’RNA, abbiamo monitorato i cambiamenti trascrizionali globali in risposta alla colonizzazione. Abbiamo osservato risposte trascrizionali diffuse, dipendenti dal tempo, che sono il risultato sia del contatto batterico che dell’ipossia luminale risultante dalla colonizzazione batterica nell’HIO. L’associazione batterica con l’epitelio immaturo ha aumentato le difese antimicrobiche e ha portato ad un miglioramento della funzione di barriera epiteliale e dell’integrità. Abbiamo osservato che NF-κB è un mediatore centrale a valle dei cambiamenti trascrizionali indotti sia dal contatto batterico che dall’ipossia. Abbiamo ulteriormente sondato il contatto batterico e le risposte epiteliali ipossia-dipendenti utilizzando l’ipossia sperimentale e l’inibizione farmacologica NF-κB, che ci ha permesso di delineare quali delle risposte trascrizionali e funzionali dell’epitelio immaturo erano dipendenti dall’ossigeno e/o NF-κB. Abbiamo scoperto che le interazioni microbico-epiteliali NF-κB-dipendenti sono state benefiche per il miglioramento della funzione di barriera e per la protezione dell’epitelio da danni causati da citochine infiammatorie. Collettivamente, questi studi fanno luce su come il contatto microbico con l’epitelio intestinale umano immaturo può portare ad una funzione modificata.
Risultati
L’epitelio intestinale derivato da cellule staminali pluripotenti trascrizionalmente assomiglia all’epitelio intestinale umano immaturo
Precedenti lavori hanno dimostrato che gli organi intestinali umani derivati da cellule staminali assomigliano a duodeno umano immaturo(Watson et al., 2014; Finkbeiner et al., 2015; Tsai et al., 2017). Inoltre, il trapianto in topi immunocompromessi porta alla maturazione dell’HIO in uno stato simile a quello degli adulti(Watson et al., 2014; Finkbeiner et al., 2015). Queste analisi hanno confrontato gli IO costituiti da epitelio e mesenchima con il tessuto intestinale umano a tutto spessore, che possedeva anche costituenti cellulari privi di IO come neuroni, vasi sanguigni e cellule immunitarie(Finkbeiner et al., 2015). Pertanto, la misura in cui l’epitelio HIO assomiglia all’epitelio intestinale immaturo/fetale è rimasta poco chiara. Per affrontare questa lacuna e caratterizzare ulteriormente l’epitelio HIO rispetto all’epitelio duodenale fetale e adulto, abbiamo isolato e coltivato epitelio da HIO cresciuto interamente in vitro, da duodeno fetale, duodeno adulto, o HIO che era stato trapiantato nella capsula renale di topi immunodeficienti NSG e maturato per 10 settimane. Questi organoidi derivati solo dall’epitelio sono stati espansi in vitro in condizioni di coltura tissutale uniforme per 4-5 passaggi e trattati per il sequenziamento dell’RNA (RNA-seq) (Figura 1-figure supplement 1). Confronto dei trascrittomi globali tra tutti i campioni oltre alle cellule staminali embrionali umane (hESC) utilizzate per generare HIO(Finkbeiner et al., 2015; E-MTAB-3158) ha rivelato una chiara gerarchia in cui sia in vitro cresciuto epitelio HIO cresciuto (p = 5,06 × 10-9) e l’epitelio trapiantato (p = 7,79 × 10-14) condivide un grado sostanzialmente maggiore di somiglianza con l’epitelio del piccolo intestino fetale (Figura 1-figuresupplement 1A).
Mentre il clustering imparziale ha dimostrato che l’epitelio trapiantato assomiglia molto a epitelio fetale, abbiamo notato uno spostamento verso il trascrittoma adulto che ha portato ad un aumento relativo della correlazione tra epitelio HIO trapiantato e l’epitelio adulto derivato dal duodeno cresciuto in vitro(Figura 1-figure supplement 1B, p = 1,17 × 10-4). Analisi dei componenti principali (PCA) di questo multi-dimensionale set di dati di espressione genica(Figura 1-figure supplement 1C) ha corroborato l’analisi di correlazione, e ha indicato che PC1 è stato correlato con lo stadio di sviluppo (PC1, 27,75% varianza cumulativa) e PC2 è stato correlato con lo stato di maturazione dei tessuti (PC2, 21.49% di varianza cumulativa); cumulativamente, PC1 e PC2 rappresentavano il 49,24% della varianza cumulativa tra i campioni, suggerendo che lo stadio di sviluppo e lo stato di maturazione dei tessuti sono le fonti principali della variazione trascrizionale tra i campioni. Epitelio HIO agglomerato con epitelio fetale lungo PC2, mentre l’epitelio HIO trapiantato agglomerato con epitelio adulto.
Abbiamo inoltre utilizzato l’analisi dell’espressione differenziale per dimostrare che l’epitelio HIO cresciuto in vitro è simile all’intestino umano immaturo, mentre l’epitelio HIO trapiantato in vivo è simile all’epitelio adulto. Per fare questo, abbiamo identificato i geni espressi in modo differenziato attraverso due confronti indipendenti: (1) epitelio fetale umano vs. epitelio adulto; (2) epitelio HIO vs. epitelio HIO trapiantato. I geni arricchiti in epitelio HIO trapiantato rispetto all’epitelio HIO sono stati confrontati con i geni arricchiti nel duodeno adulto rispetto al duodeno fetale(Figura 1-figure supplement 1D). C’era una correlazione altamente significativa tra i rapporti di espressione log2-trasformati in cui gli IO trapiantati e l’epitelio adulto condividevano geni arricchiti mentre l’IO e l’epitelio fetale condividevano geni arricchiti (p=2,6 × 10-28). Questa analisi supporta i dati pubblicati in precedenza che indicano che l’epitelio di IO cresciuto in vitro ricapitola la firma di espressione genica del duodeno immaturo e dimostra che l’epitelio IO è in grado di adottare una firma trascrizionale che assomiglia maggiormente al duodeno adulto dopo il trapianto nei topi.
Gli HIO possono essere associati in modo stabile con l’E. coli non patogeno
Dato che l’epitelio HIO ricapitola molte delle caratteristiche dell’epitelio intestinale immaturo, ci siamo proposti di valutare l’effetto della colonizzazione batterica sull’epitelio HIO ingenuo. Studi precedenti hanno stabilito che gli organi intestinali derivati da cellule staminali pluripotenti possono essere iniettati con virus vivi(Finkbeiner et al., 2012) o con agenti patogeni batterici(Leslie et al., 2015; Engevik et al., 2015; Forbester et al. , 2015); tuttavia, non si sapeva se gli HIO potessero essere co-colturati in modo stabile con microrganismi non patogeni. Abbiamo co-coltivato gli IO con il ceppo non mobile di Esherichia coli di derivazione umana ECOR2(Ochman e Selander, 1984). L’intero sequenziamento del genoma e l’analisi filogenetica hanno dimostrato che l ‘E. col i str. ECOR2 è strettamente correlato ad altri E. coli umani non patogeni e solo lontanamente correlati con E. col i patogeni e isolati di Shigella(Figura 1-figure supplement 3). Abbiamo sviluppato una tecnica di microiniezione per introdurre E. col i vivo nel lume HIO in modo da evitare la contaminazione dei mezzi circostanti(Figura 1-figure supplement 2). Gli HIO microiniettati con105 E. col i vivi che esprimono in modo costitutivo le GFP mostrano una robusta fluorescenza verde entro 3 ore dalla microiniezione(Figura 1A e Video 1). Numerosi E. col i localizzati nello spazio luminale a 48 ore dopo la microiniezione e sono presenti adiacenti all’epitelio HIO, con alcuni apparentemente residenti in stretta opposizione alla superficie epiteliale apicale(Figura 1B).
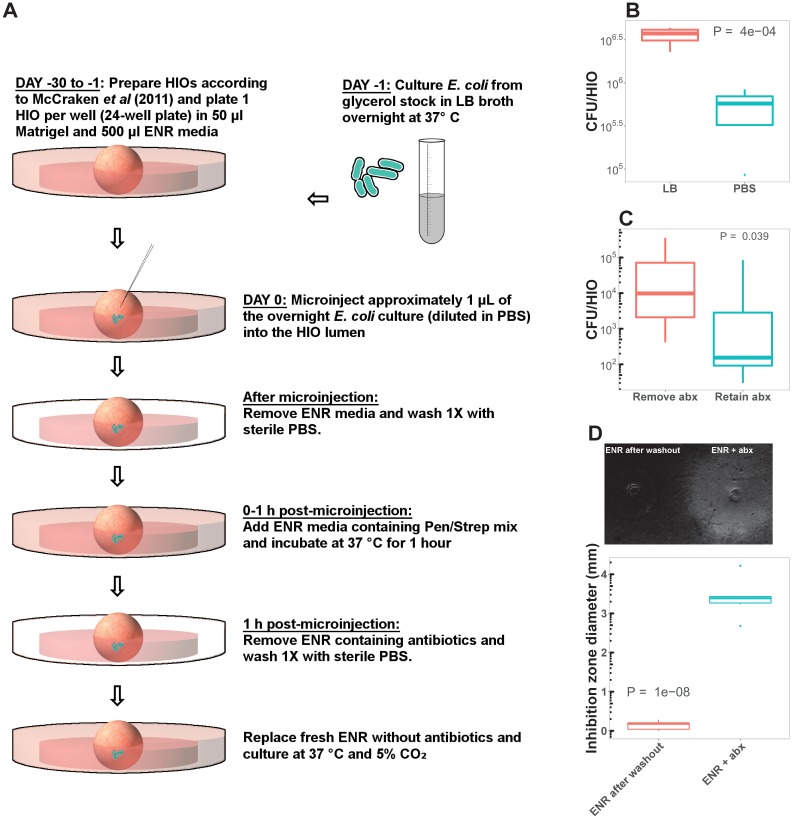
Figura 1-figura supplemento 3.Figura 1. HIO può essere stabilmente associato con non patogeno E. coli.Pluripotent staminali cellule staminali di origine intestinale epitelio trascrizionalmente assomiglia l’immaturo umano epitelio intestinale.albero filogenetico basato sulla massima somiglianza distanza genomica tra E.coli str.HIO protocollo di colonizzazione.(A) Intensità fluorescente media di un organoide intestinale umano (HIO) contenente GFP+E. coli str.HIO vivo. ECOR2. I pannelli inferiori mostrano immagini rappresentative delle serie temporali. Rappresentante di tre esperimenti indipendenti. Il video 1 è un’animazione corrispondente a questo dataset.(B) Micrografia confocale dell’epitelio HIO (E-caderina) in associazione diretta con GFP+ E. colia 48 ore dopo la microiniezione con 104 E. coli indiretta. Ingrandimento 60X.C) CFU luminosi per HIO E. coli a 24 ore dopo la microiniezione rispetto alla concentrazione iniettata di 5 × 10-1 a 5 × 105 CFU per HIO all’inizio dell’esperimento. N= 10 repliche biologiche per dose di E. coli. Il valore r2e P mostrato in figura rappresentano i risultati di un’analisi di regressione lineare della relazione tra il cambiamento di 24 ore in CFU/HIO e il numero iniziale di CFU iniettate.(D) CFU luminosi per HIO a 0-72 ore dopo la microiniezione con 10 CFU E. coli per HIO. N= 13-17 replicare gli HIO per punto temporale. Il valore p rappresenta i risultati del t-test di uno Studente a due code che confronta le due condizioni indicate.(E) Percentuale giornaliera di colture HIO senza E. coli coltivabile nei media esterni a seguito di microiniezione di E. coli (N= 48) o microiniezione di PBS (N = 8).(A) Matrice di correlazione di Pearson con il clustering erarchico per il gene RNA-seq normalizzato per il trascrittoma intero conta RNA-seq da epitelio isolato dai tessuti indicati sugli assi.(B) Pearson coefficiente di correlazione per il confronto di tutto il trascrittoma normalizzato RNA-seq conteggi gene RNA-seq tra ciascuno dei tipi di campione elencati sull’asse x e adulti epitelio intestinale piccolo intestinale. p-Valore indica i risultati di uno studente non accoppiato su due lati t-test.(C) L’analisi dei componenti di principio di tutto il complesso del trascrittoma RNA-seq normalizzato conta dei geni. Cumulativo spiegato varianza per PC1 e PC2 è indicato come una percentuale sugli assi x e y, rispettivamente.(D) Tracciato della densità del Log2 -trasformato Fold cambiamento di densità nell’espressione genica in epitelio da HIO trapiantato su epitelio da HIO coltivato in vitro tracciata contro il Log 2 -trasformato Fold cambiamento di densità nell’espressione genica in epitelio dell’intestino tenue adulto su epitelio fetale dell’intestino tenue. L’intensità del colore blu indica la densità dei punti nello spazio bidimensionale. SI, intestino tenue; tx, tessuto trapiantato; hPSC, cellula staminale umana pluripotente; HIO, organoide intestinale umano.ECOR2(Ochman e Selander, 1984), il ceppo utilizzato negli esperimenti di colonizzazione HIO, isolati di E. coli strettamente correlati disponibili sul database PATRIC(Wattam et al., 2017), e ceppi di tipo patogeno dai generi Esherichia, Shigella e Salmonella. Un numero di riferimento del genoma PATRIC segue il nome di ciascun taxa.(A) Rappresentazione schematica della microiniezione di IO con E. coli vivi. Vedere Materiali e metodi per ulteriori dettagli.B) Un confronto tra CFU/HIO a 24 ore di microiniezione di HIO microiniezione di HIO con E. coli vivo 10 × 103 CFU diluito in PBS sterile o brodo LB fresco. N = 5 HIO per condizione. Tutti gli esperimenti presentati nel documento principale rappresentano E. col i diluito in PBS.(C) Per testare l’effetto del carryover antibiotico della crescita di E. coli nel lume dell’HIO, abbiamo confrontato CFU/HIO in HIO coltivati in mezzi esenti da antibiotici (‘Remove abx’) o mezzi contenenti penicillina e streptomicina a 24 ore dopo la microiniezione con 10 3 CFU (unità formanti colonie) di E. coli vivi. Tutti gli esperimenti presentati nell’articolo principale rappresentano gli HIO coltivati in mezzi privi di antibiotici a causa dell’apparente effetto degli antibiotici nel sopprimere la crescita dell’E. coli all’interno del lume dell’HIO. N = 5 IO per condizione.(D) In diversi esperimenti, la traslocazione batterica è stata misurata mediante samplificazione dei terreni di coltura HIO esterni(Figura 1 e 8). Per valutare la potenziale influenza del carryover antibiotico nelle nostre stime di traslocazione batterica, abbiamo misurato l’inibizione della crescita in colture di E. coli placcate come un prato di agar LB e trattate con 1 μl di campioni di media HIO raccolti durante la fase di lavaggio antibiotico di 1 ora o dopo il lavaggio della coltura HIO con PBS e la sostituzione con media freschi privi di antibiotici (vedi pannello A). N = 6 repliche per condizione di coltura. Tutti i valori p rappresentano i risultati dei confronti del t-test dello studente con una coda non accoppiata.

Video 1.2. Animazione di singole immagini microscopiche epifluorescenti da un organoide intestinale umano (HIO) contenente GFP+ E.coli str.ECOR2. Le immagini sono state catturate a intervalli di 10 minuti nel corso di 18 ore e coalizzate in ordine sequenziale. Rappresentante di tre esperimenti indipendenti. Vedi Figura 1A.
Al fine di determinare il numero minimo di unità di formazione della colonia (CFU) di E. col i necessari per stabilire la colonizzazione a breve termine (24 ore), abbiamo microiniettato un numero crescente di E. coli vivo sospeso in PBS in HIOs singolo e raccolto e determinato il numero di batteri nel contenuto luminale a 24 ore post-microiniezione(Figura 1C). I singoli HIO possono essere colonizzati in modo stabile da un minimo di 5 CFU E. coli per HIO con il 77,8% di successo (cultura luminale positiva e media culture esterne negative a 24 ore dopo l’iniezione) e il 100% di successo a ≥100 CFU per HIO (Figura 1C). L’aumento del numero di CFU E. coli microiniettato in ogni HIO a t= 0 ha portato ad un significativo aumento del CFU luminale medio per HIO a 24 ore dopo la microiniezione a qualsiasi dose (ANOVA p=0,37; Figura 1C) . Così, il tasso di crescita di 24 ore di E. coli all’interno del lume HIO (CFU×HIOt = 24-1CFU×HIOt = 0-1) è stato correlato negativamente con il CFU iniettato (r2 = 0,625, p = 3,1 × 10-12; Figura 1C).
Successivamente, abbiamo esaminato la stabilità delle co-colture di HIO e di E. coli nel tempo in vitro. Gli HIO sono stati microiniettati con 10 CFU E. col i e mantenuti per 24-72 ore (Figura 1D). Rapida espansione di E. coli densità all’interno del lume HIO è stata osservata nelle prime 24 ore, con densità batterica relativamente stabile a 48-72 ore. Un aumento di 6,25 volte la densità batterica è stato osservato tra 24 e 72 ore dopo la microiniezione (p = 0,036). È importante notare che i campioni prelevati dai terreni di coltura HIO esterni erano negativi per la crescita dell’E. coli.
Infine, abbiamo esaminato la stabilità delle colture HIO dopo la microiniezione di E. col i(Figura 1E). Un totale di 48 singoli HIO sono stati sottoposti a microiniezione con 104 CFU E. col i ciascuno. I controlli sono stati microiniettati con il solo PBS sterile. Abbiamo scoperto che i mezzi di coltura esterni erano sterili nel 100% degli HIO di controllo durante l’intero esperimento, e nel 100% degli HIO iniettati con E . col i nei giorni 0-2 post-microiniezione. Nei giorni 3-9 post-microiniezione alcuni terreni di coltura erano positivi per la crescita dell’E. coli; tuttavia, il 77,08% degli IIO iniettati con E. coli erano negativi per l’E. coli nei terreni di coltura esterni durante tutto l’arco di tempo. Sono stati condotti ulteriori esperimenti di controllo per determinare se i mezzi di coltura HIO hanno avuto un effetto sulla crescita dell’E. coli. I terreni di coltura HIO inoculati con E. coli hanno dimostrato che i terreni stessi hanno permesso una robusta crescita batterica e quindi l’assenza di crescita di E. coli in terreni esterni provenienti da colture HIO non poteva essere attribuita alla sola composizione dei terreni(Figura 1-figure supplement 3). Pertanto, la grande maggioranza degli HIO colonizzati da E. col i rimane stabile per un periodo prolungato quando viene coltivata in vitro e senza antibiotici.

Figura 1-figure supplement 3.Figura 1—figura supplemento 3. Gli IO possono essere associati in modo stabile con l’E. coli non patogeno. L’epitelio intestinale derivato da cellule staminali Pluripotenti trascrizionalmente assomiglia all’epitelio intestinale umano immaturo.l’albero filogenetico basato sulla massima distanza genomica di somiglianza tra il protocollo di colonizzazione di E. coli str.HIO.(A) Intensità media di fluorescenza di un organoide intestinale umano (HIO) contenente GFP+E. coli str.HIO vivo. ECOR2. I pannelli inferiori mostrano immagini rappresentative delle serie temporali. Rappresentante di tre esperimenti indipendenti. Il video 1 è un’animazione corrispondente a questo dataset.(B) Micrografia confocale dell’epitelio HIO (E-caderina) in associazione diretta con GFP+ E. colia 48 ore dopo la microiniezione con 104 E. coli indiretta. Ingrandimento 60X.C) CFU luminosi per HIO E. coli a 24 ore dopo la microiniezione rispetto alla concentrazione iniettata di 5 × 10-1 a 5 × 105 CFU per HIO all’inizio dell’esperimento. N= 10 repliche biologiche per dose di E. coli. Il valore r2e P mostrato in figura rappresentano i risultati di un’analisi di regressione lineare della relazione tra il cambiamento di 24 ore in CFU/HIO e il numero iniziale di CFU iniettate.(D) CFU luminosi per HIO a 0-72 ore dopo la microiniezione con 10 CFU E. coli per HIO. N= 13-17 replicare gli HIO per punto temporale. Il valore p rappresenta i risultati del t-test di uno Studente a due code che confronta le due condizioni indicate.(E) Percentuale giornaliera di colture HIO senza E. coli coltivabile nei media esterni a seguito di microiniezione di E. coli (N= 48) o microiniezione di PBS (N = 8).(A) Matrice di correlazione di Pearson con il clustering erarchico per il gene RNA-seq normalizzato per il trascrittoma intero conta RNA-seq da epitelio isolato dai tessuti indicati sugli assi.(B) Pearson coefficiente di correlazione per il confronto di tutto il trascrittoma normalizzato RNA-seq conteggi gene RNA-seq tra ciascuno dei tipi di campione elencati sull’asse x e adulti epitelio intestinale piccolo intestinale. p-Valore indica i risultati di uno studente non accoppiato su due lati t-test.(C) L’analisi dei componenti di principio di tutto il complesso del trascrittoma RNA-seq normalizzato conta dei geni. Cumulativo spiegato varianza per PC1 e PC2 è indicato come una percentuale sugli assi x e y, rispettivamente.(D) Tracciato della densità del Log2 -trasformato Fold cambiamento di densità nell’espressione genica in epitelio da HIO trapiantato su epitelio da HIO coltivato in vitro tracciata contro il Log 2 -trasformato Fold cambiamento di densità nell’espressione genica in epitelio dell’intestino tenue adulto su epitelio fetale dell’intestino tenue. L’intensità del colore blu indica la densità dei punti nello spazio bidimensionale. SI, intestino tenue; tx, tessuto trapiantato; hPSC, cellula staminale umana pluripotente; HIO, organoide intestinale umano.ECOR2(Ochman e Selander, 1984), il ceppo utilizzato negli esperimenti di colonizzazione HIO, isolati di E. coli strettamente correlati disponibili sul database PATRIC(Wattam et al., 2017), e ceppi di tipo patogeno dai generi Esherichia, Shigella e Salmonella. Un numero di riferimento del genoma PATRIC segue il nome di ciascun taxa.(A) Rappresentazione schematica della microiniezione di IO con E. coli vivi. Vedere Materiali e metodi per ulteriori dettagli.B) Un confronto tra CFU/HIO a 24 ore di microiniezione di HIO microiniezione di HIO con E. coli vivo 10 × 103 CFU diluito in PBS sterile o brodo LB fresco. N = 5 HIO per condizione. Tutti gli esperimenti presentati nel documento principale rappresentano E. col i diluito in PBS.(C) Per testare l’effetto del carryover antibiotico della crescita di E. coli nel lume dell’HIO, abbiamo confrontato CFU/HIO in HIO coltivati in mezzi esenti da antibiotici (‘Remove abx’) o mezzi contenenti penicillina e streptomicina a 24 ore dopo la microiniezione con 10 3 CFU (unità formanti colonie) di E. coli vivi. Tutti gli esperimenti presentati nell’articolo principale rappresentano gli HIO coltivati in mezzi privi di antibiotici a causa dell’apparente effetto degli antibiotici nel sopprimere la crescita dell’E. coli all’interno del lume dell’HIO. N = 5 IO per condizione.(D) In diversi esperimenti, la traslocazione batterica è stata misurata mediante samplificazione dei terreni di coltura HIO esterni(Figura 1 e 8). Per valutare la potenziale influenza del carryover antibiotico nelle nostre stime di traslocazione batterica, abbiamo misurato l’inibizione della crescita in colture di E. coli placcate come un prato di agar LB e trattate con 1 μl di campioni di media HIO raccolti durante la fase di lavaggio antibiotico di 1 ora o dopo il lavaggio della coltura HIO con PBS e la sostituzione con media freschi privi di antibiotici (vedi pannello A). N = 6 repliche per condizione di coltura. Tutti i valori p rappresentano i risultati dei confronti del t-test dello studente con una coda non accoppiata.
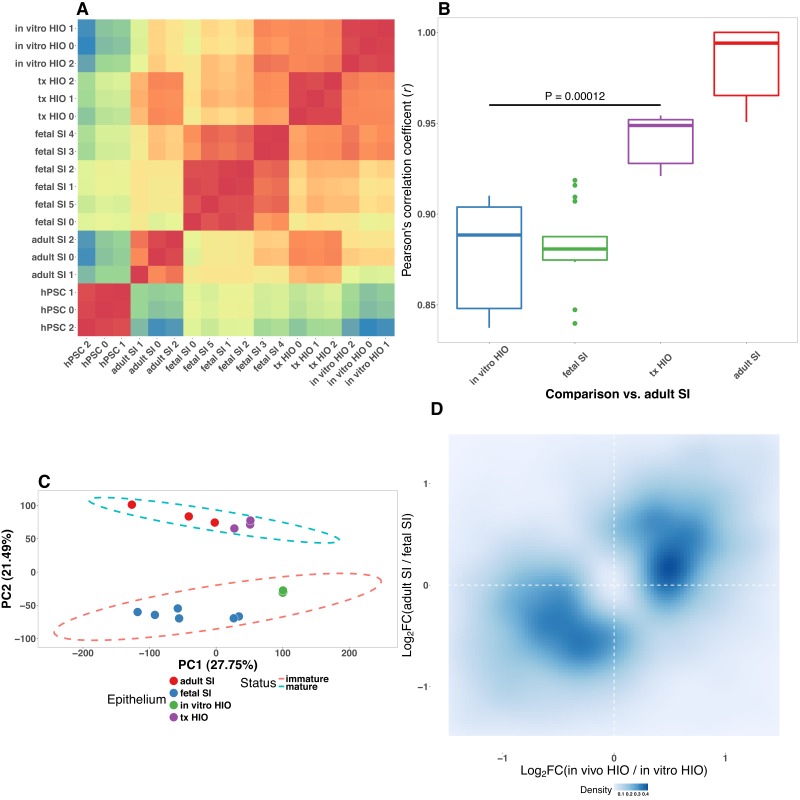
Figura 1-figure supplemento 1.Figura 1—figura 1. L’epitelio intestinale derivato da cellule staminali pluripotenti trascrizionalmente assomiglia all’epitelio intestinale umano immaturo.(A) Matrice di correlazione di Pearson con il clustering erarchico per il gene RNA-transcriptomo intero normalizzato trascriptome conta RNA-seq da epitelio isolato dai tessuti indicati sugli assi.(B) Pearson coefficiente di correlazione per il confronto di tutto il trascrittoma normalizzato RNA-seq conteggi gene RNA-seq tra ciascuno dei tipi di campione elencati sull’asse x e adulti epitelio intestinale piccolo intestinale. p-Valore indica i risultati di uno studente non accoppiato su due lati t-test.(C) L’analisi dei componenti di principio di tutto il complesso del trascrittoma RNA-seq normalizzato conta dei geni. Cumulativo spiegato varianza per PC1 e PC2 è indicato come una percentuale sugli assi x e y, rispettivamente.(D) Tracciato della densità del Log2 -trasformato Fold cambiamento di densità nell’espressione genica in epitelio da HIO trapiantato su epitelio da HIO coltivato in vitro tracciata contro il Log 2 -trasformato Fold cambiamento di densità nell’espressione genica in epitelio dell’intestino tenue adulto su epitelio fetale dell’intestino tenue. L’intensità del colore blu indica la densità dei punti nello spazio bidimensionale. SI, intestino tenue; tx, tessuto trapiantato; hPSC, cellula staminale umana pluripotente; HIO, organoide intestinale umano.
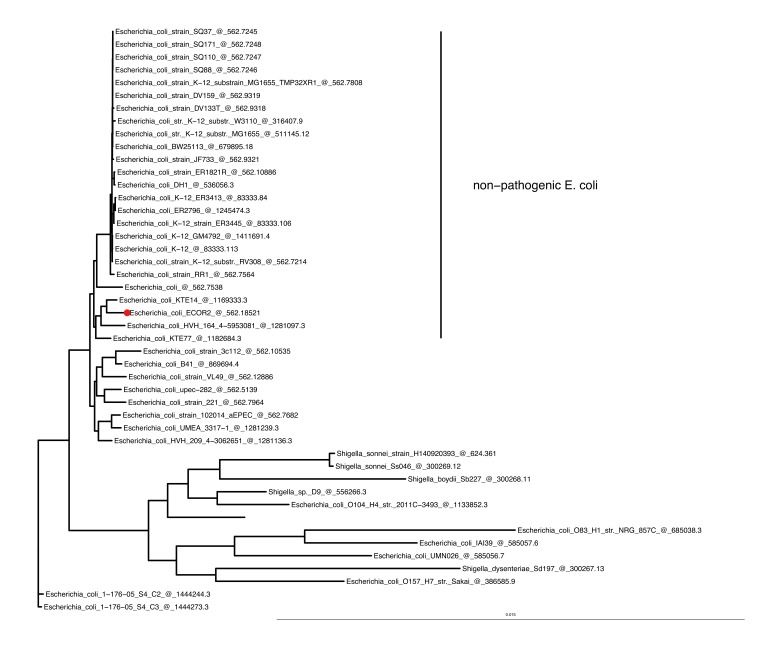
Figura 1-figure supplemento 2.Albero filogenetico basato sulla distanza genomica di massima somiglianza tra E.coli str.ECOR2(Ochman e Selander, 1984), il ceppo utilizzato negli esperimenti di colonizzazione HIO, isolati di E . coli strettamente correlati disponibili sul database PATRIC(Wattam et al., 2017), e ceppi di tipo patogeno dai generi Esherichia, Shigella e Salmonella. Un numero di riferimento del genoma PATRIC segue il nome di ciascun taxa.

Figura 1-figure supplement 3.Protocollo di colonizzazione HIO.(A) Rappresentazione schematica della microiniezione di HIO con E. coli vivo. Vedere Materiali e metodi per ulteriori dettagli.(B) Un confronto di CFU/HIO a 24 hpost-microiniezione di HIO microiniezione di HIO con 10 × 103 CFU coli vivi di E. coli diluito in PBS sterile o brodo LB fresco. N = 5 HIO per condizione. Tutti gli esperimenti presentati nel documento principale rappresentano E. col i diluito in PBS.(C) Per testare l’effetto del carryover antibiotico della crescita di E. coli nel lume dell’HIO, abbiamo confrontato CFU/HIO in HIO coltivati in mezzi esenti da antibiotici (‘Remove abx’) o mezzi contenenti penicillina e streptomicina a 24 ore dopo la microiniezione con 10 3 CFU (unità formanti colonie) di E. coli vivi. Tutti gli esperimenti presentati nell’articolo principale rappresentano gli HIO coltivati in mezzi privi di antibiotici a causa dell’apparente effetto degli antibiotici nel sopprimere la crescita dell’E. coli all’interno del lume dell’HIO. N = 5 IO per condizione.(D) In diversi esperimenti, la traslocazione batterica è stata misurata mediante samplificazione dei terreni di coltura HIO esterni(Figura 1 e 8). Per valutare la potenziale influenza del carryover antibiotico nelle nostre stime di traslocazione batterica, abbiamo misurato l’inibizione della crescita in colture di E. coli placcate come un prato di agar LB e trattate con 1 μl di campioni di media HIO raccolti durante la fase di lavaggio antibiotico di 1 ora o dopo il lavaggio della coltura HIO con PBS e la sostituzione con media freschi privi di antibiotici (vedi pannello A). N = 6 repliche per condizione di coltura. Tutti i valori p rappresentano i risultati dei confronti del t-test dello studente con una coda non accoppiata.

Video 1.2. Animazione di singole immagini microscopiche epifluorescenti da un organoide intestinale umano (HIO) contenente GFP+ E.coli str.ECOR2. Le immagini sono state catturate a intervalli di 10 minuti nel corso di 18 ore e coalizzate in ordine sequenziale. Rappresentante di tre esperimenti indipendenti. Vedi Figura 1A.
La colonizzazione batterica provoca una risposta trascrizionale su larga scala, dipendente dal tempo
La colonizzazione dell’intestino immaturo da parte dei microbi è associata alla maturazione funzionale in entrambi i sistemi modello (Kremeret al., 2013; Sommer et al., 2015; Broderick et al., 2014; Erkosar et al., 2015) e nei neonati umani (Renz et al.,2011). Per valutare se l’esposizione degli HIO all’E. coli ha portato alla maturazione dell’interfaccia epiteliale, abbiamo valutato gli eventi trascrizionali che seguono la microiniezione di E. coli vivo nel lume dell’HIO. HIO iniettati con PBS (controlli) e HIO co-colturati con E. coli sono stati raccolti per l’analisi trascrizionale dopo 24, 48 e 96 ore(Figura 2). A 24 ore dopo la microiniezione, un totale di 2018 geni sono stati espressi in modo differenziato (regolato-FDR < 0,05), e il numero totale di geni espressi in modo differenziato è stato ulteriormente aumentato a 48 e 96 ore dopo la microiniezione rispetto ai controlli PBS-iniezione (Figura 2A). L’analisi dei componenti di principio ha dimostrato che l’attività trascrizionale globale negli IO è significativamente alterata dall’esposizione all’E. coli, con il grado di cambiamento trascrizionale rispetto agli IO di controllo che aumenta nel tempo(Figura 2B).
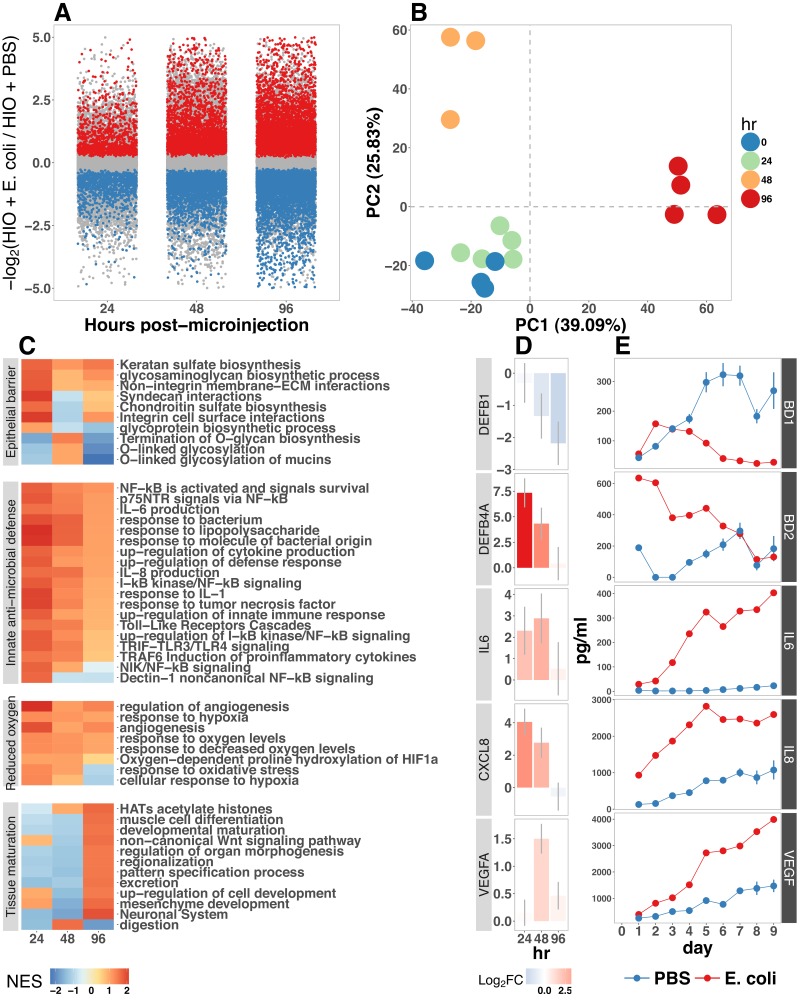
Figura 2.(A) Log2-transformato cambiamento di piega nel conteggio normalizzato RNA-seq gene RNA-seq in E. coli colonizzato HIOs a 24, 48, e 96 ore post-microiniezione con 104vivo E. coli rispetto a PBS-iniezione HIOs. I geni espressi in modo differenziale (FDR-adjusted p-value < 0,05) sono indicati in rosso (up-regulated) o blu (down-regulated). I risultati tracciate sono il cambiamento medio di piega per gene per ogni gruppo.(B) Principio componente grafico dei componenti di HIO a 0-96 ore dopo la microiniezione derrivati da interi conteggi di RNA-trascriptome RNA-seq gene normalizzato. La varianza cumulativa spiegata per PC1 e PC2 è indicata come percentuale sugli assi x e y, rispettivamente.(C) Mappa termica dei punteggi di arricchimento normalizzati (NES) da GSEA di dati di espressione RNA-seq normalizzati utilizzando i database GO e REACTOME. Un valore positivo di NES indica l’attivazione di un determinato set di geni e un valore negativo suggerisce la soppressione relativa di un set di geni. Tutti i punteggi di NES sono calcolati in relazione ai controlli PBS-microiniezione.(D) Cambiamento medio log2 fold change in conteggi normalizzati di RNA-seq gene normalizzati a 24-96 ore dopo la microiniezione rispetto ai controlli HIOs PBS-injected.(E) Secrezione di proteine a 0-9 giorni dopo la microiniezione con PBS o E. coli misurata con ELISA nel supernatante di colture HIO. I geni indicati in D corrispondono alle proteine misurate in E. N= 4 (0 ore), 5 (24 ore), 3 (48 ore) e 4 (96 ore) repliche biologiche composte da 5-6 HIO riuniti per replicato per i pannelli A-D. N= 48 IIO iniettati con coli-iniezione di E. e N = 8 IIO iniettati con PBS per il pannello E.
Gene set enrichment analysis (GSEA)(Subramanian et al., 2005) utilizzando i database GO(Ashburner et al., 2000; Gene Ontology Consortium, 2015) e REACTOME(Croft et al., 2014; Fabregat et al., 2016) per valutare i dati di espressione dell’RNA-seq hanno rivelato cambiamenti coordinati nell’espressione genica relativi alla difesa antimicrobica innata, alla produzione di barriere epiteliali, all’adattamento a basso ossigeno e alla maturazione dei tessuti(Figura 2C). Percorsi di difesa antimicrobica innata, compresi i geni correlati alla segnalazione NF-κB, la produzione di citochine, e la segnalazione del recettore Toll-like receptor (TLR) sono stati fortemente up-regolati a 24 ore dopo la microiniezione e in generale hanno mostrato una diminuzione dell’attivazione in punti successivi. GSEA ha anche rivelato cambiamenti nell’espressione genica coerente con i livelli di ossigeno ridotti o ipossia, compresa l’induzione di segnali pro-angiogenesi. Un certo numero di percorsi relativi alla sintesi e modificazione delle glicoproteine, tra cui mucine O-linked, glicosaminoglicani e proteoglicani, sono stati up-regolati nelle fasi iniziali della risposta trascrizionale (Syndecans, integrine), ha mostrato un inizio un po ‘ritardato (O-linked mucine), o ha mostrato l’attivazione coerente in tutti i punti di tempo post-microiniezione (cheratano solfato e glicosaminoglicano biosintesi). Infine, gli insiemi di geni associati a una serie di processi coinvolti nella maturazione e nello sviluppo dei tessuti hanno seguito un distinto modello di espressione tardivo. Questo includeva termini di ontologia genica ampia per la morfogenesi d’organo, maturazione dello sviluppo, e la regionalizzazione, nonché processi più specifici come la differenziazione delle cellule mesenchimali e muscolari, e processi associati con il sistema nervoso(Figura 2C).
Abbiamo anche fatto correlazioni tra i geni upregulated nei dati RNA-seq(Figura 2D) e fattori proteici presenti nei mezzi di coltura organica a seguito di microiniezione di E. coli(Figura 2E). β-defensina 1 (DEFB1( gene); BD-1 (proteina)) e β-defensina 2 (DEFB4A(gene); BD-2 (proteina)) hanno mostrato modelli distinti di espressione, con sia DEFB1 e il suo prodotto proteico BD-1 stabile a 24 ore dopo la microiniezione di E. coli, ma relativamente soppressa in punti di tempo successivi, e DEFB4A e BD-2 fortemente indotta in punti di tempo precoce e cedendo nel tempo rispetto ai controlli PBS-iniezione. Al contrario, i regolatori infiammatori IL-6 e IL-8 e il fattore pro-angiogenesi VEGF sono stati fortemente indotti a livello trascrizionale entro 24-48 ore dalla microiniezione di E. coli. La secrezione di IL-6, IL-8 e VEGF è aumentata nel tempo, raggiungendo un picco di 5-9 giorni dopo l’associazione dell’E. coli rispetto ai controlli iniettati con PBS (Figura 2E). Presi insieme, questi dati dimostrano una risposta trascrizionale su larga scala e dipendente dal tempo all’associazione di E. col i con distinti modelli di espressione genica e secrezione di proteine in fase precoce e tardiva.

Figura 2.(A) Log2-transformato cambiamento di piega nel conteggio normalizzato RNA-seq gene RNA-seq in E. coli colonizzato HIOs a 24, 48, e 96 ore post-microiniezione con 104 E. colivivo rispetto a PBS-iniezione HIOs. I geni espressi in modo differenziale (FDR-adjusted p-value < 0,05) sono indicati in rosso (up-regulated) o blu (down-regulated). I risultati tracciate sono il cambiamento medio di piega per gene per ogni gruppo.(B) Principio componente grafico dei componenti di HIO a 0-96 ore dopo la microiniezione derrivati da interi conteggi di RNA-trascriptome RNA-seq gene normalizzato. La varianza cumulativa spiegata per PC1 e PC2 è indicata come percentuale sugli assi x e y, rispettivamente.(C) Mappa termica dei punteggi di arricchimento normalizzati (NES) da GSEA di dati di espressione RNA-seq normalizzati utilizzando i database GO e REACTOME. Un valore positivo di NES indica l’attivazione di un determinato set di geni e un valore negativo suggerisce la soppressione relativa di un set di geni. Tutti i punteggi di NES sono calcolati in relazione ai controlli PBS-microiniezione.(D) Cambiamento medio log2 fold change in conteggi normalizzati di RNA-seq gene normalizzati a 24-96 ore dopo la microiniezione rispetto ai controlli HIOs PBS-injected.(E) Secrezione di proteine a 0-9 giorni dopo la microiniezione con PBS o E. coli misurata con ELISA nel supernatante di colture HIO. I geni indicati in D corrispondono alle proteine misurate in E. N= 4 (0 ore), 5 (24 ore), 3 (48 ore) e 4 (96 ore) repliche biologiche composte da 5-6 HIO riuniti per replicato per i pannelli A-D. N= 48 IIO iniettati con coli-iniezione di E. e N = 8 IIO iniettati con PBS per il pannello E.
La colonizzazione batterica determina un aumento transitorio della proliferazione epiteliale e della maturazione degli enterociti
Mentre l’analisi trascrizionale ha dimostrato forti cambiamenti dipendenti dal tempo nelle cellule che compongono l’HIO a seguito della colonizzazione di E. coli, abbiamo ipotizzato che l’esposizione ai batteri può anche alterare il comportamento cellulare e / o la composizione dell’HIO. Studi precedenti hanno dimostrato che la colonizzazione batterica promuove la proliferazione epiteliale in organismi modello(Bates et al., 2006; Cheesman et al., 2011; Neal et al., 2013; Kremer et al., 2013; Ijssennagger et al., 2015). Abbiamo esaminato la proliferazione epiteliale negli IO su un arco di tempo di 96 ore trattando gli IO con una singola esposizione di 2 ore di 10 μM EdU aggiunti ai terreni di coltura da 22 a 24 ore dopo la microiniezione con 104 CFU E. coli o PBS da soli. HIOs sono stati successivamente raccolti per immunoistochimica a 24, 48, e 96 ore dopo la microiniezione(Figura 3). Il numero di cellule epiteliali proliferanti (Edu + + e E-caderina +) è stato elevato di ben tre volte in E. coli colonizzato HIO rispetto ai PBS trattati con PBS a 24 ore(Figura 3A-B). Tuttavia, a 48 ore dopo la microiniezione, la proporzione di cellule epiteliali EdU + è stata significativamente diminuita in E. coli colonizzato HIOs rispetto al controllo HIOs trattati trattati. Questa osservazione è stata supportata da un altro marcatore di proliferazione, KI67 (Gerdes et al., 1984)(Figura 3B), così come RNA-seq dati che dimostrano una soppressione complessiva dei geni del ciclo cellulare in E. col i colonizzato HIOs HIOs relativi a PBS-iniezione HIOs a 48 ore post-microiniezione(Figura 3-figure supplemento 1). Da 96 ore dopo la microiniezione la proporzione di cellule epiteliali EdU + era quasi identica in E. coli e PBS-IOs trattati con PBS (Figura 3B). Collettivamente, questi risultati suggeriscono che la colonizzazione di E. col i è associata ad una rapida esplosione di proliferazione epiteliale, ma che relativamente poche delle cellule figlie risultanti sono conservati successivamente all’interno dell’epitelio.
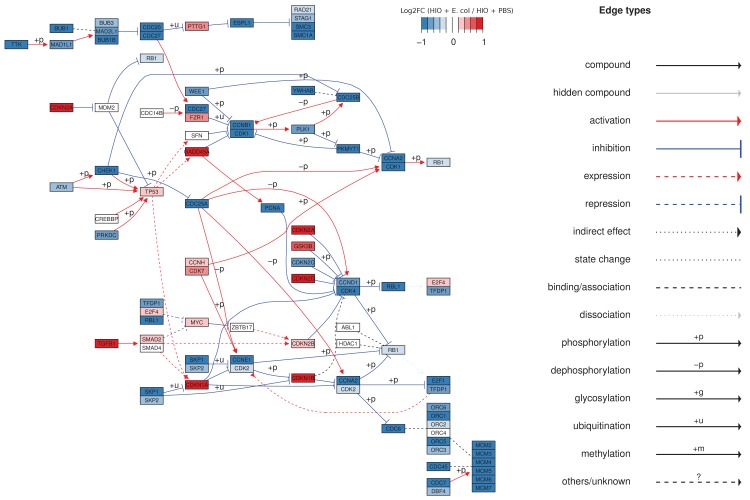
Figura 3-figure supplemento 1.La colonizzazione batterica provoca un aumento transitorio della proliferazione epiteliale e la maturazione degli enterociti.Pathview(Luo e Brouwer, 2013) trama del percorso KEGG(Kanehisa e Goto, 2000) che mostra il ciclo mitotico cellulare (‘HSA 04110’) sovrapposto con RNA-seq dati di espressione corrispondenti al log2 -trasformati cambiamento piega in espressione di trascrizioni del ciclo cellulare da HIOs microiniezione con E. coli rispetto a PBS-iniezione HIOs a 48 h post-microiniezione.(A) Micrografie confocali rappresentative di IO iniettati con PBS o104 CFU E. coli str. ECOR2 a 24-96 ore dopo la microiniezione e colorati con indicatori fluorescenti per EdU+DNA, E-caderina o nuclei (DAPI) come indicato nelle etichette della figura . Tutti gli HIO sono stati esposti a 10 μM EdU a 22 ore dopo la microiniezione e l’EdU è stato rimosso a 24 ore. I pannelli sono rappresentativi di 4 IOO per punto temporale per condizione di trattamento.(B) Quantificazione del numero di cellule epiteliali EdU-positive e Ki67-positive (cellule E-caderina + cellule) per 10X campo di microscopia confocale. Un campo di microscopia confocale 10X composto da 200-1000 cellule epiteliali è stato raccolto da ciascuno dei 4 HIO per punto temporale per gruppo di trattamento. Le barre di errore rappresentano l’errore standard della media e i valori p riflettono i risultati di un t-test di uno Studente non accoppiato a due code che confronta gli IO iniettati con gli IO iniettati con PBS in quel punto temporale.(C) Micrografie confocali rappresentative degli IO iniettati con PBS o104 CFU E. coli str. ECOR2 a 24-96 ore dopo la microiniezione e colorato con anticorpi fluorescenti per Sox9, E-caderina o nuclei (DAPI) come indicato nelle etichette della figura. I pannelli sono rappresentativi di 4 HIO per punto temporale per condizione di trattamento.Media di 4 repliche biologiche, ognuna delle quali rappresenta 5-6 HIO raggruppati.
Il fattore di trascrizione Sox9 è espresso da cellule progenitrici nell’epitelio intestinale murino (Bastideet al., 2007; Mori-Akiyama et al., 2007), e diversi sottotipi epiteliali sono derivati da una popolazione di progenitori Sox9-espressiva nell’epitelio intestinale maturo (Bastide et al., 2007; Furuyama et al., 2011). Abbiamo esaminato l’espressione SOX9 in HIOs a seguito di microiniezione con E. coli o PBS da sola su un corso di 96 ore(Figura 3C). Negli HIO trattati con PBS, la maggior parte delle cellule epiteliali ha mostrato una robusta espressione nucleare SOX9 in tutti i punti temporali esaminati. Tuttavia, l’espressione SOX9 è stata drasticamente ridotta in E. coli colonizzato HIOs a 48-96 ore dopo la microiniezione ed è stato notevolmente distribuito in nuclei più lontano dal lume e adiacente al mesenchima sottostante, rispecchiando la distribuzione alterata di EdU + nuclei visto in Figura 3B. Questa osservazione suggerisce che vi è una riduzione del numero di cellule progenitrici nell’epitelio HIO a seguito della colonizzazione di E. coli e implica che altri tipi di epitelio possono rappresentare una maggiore proporzione dell’epitelio HIO in punti successivi post-colonizzazione. Non abbiamo visto alcuna colorazione apprezzabile per le cellule epiteliali che esprimono il calice, Paneth, o marcatori cellulari enteroendocrini (MUC2, DEFA5, e CHGA, rispettivamente; dati negativi non mostrati). Tuttavia, l’espressione del piccolo intestinale pennello confine enzima dipeptidyl peptidase-4 (DPPIV) è stato trovato per essere robustly espresso nel E. coli colonizzato HIOs a 48 e 96 ore post-microiniezione(Figura 3D). La DPPIV non è stata rilevata in nessuno degli IIO iniettati con PBS in nessun punto temporale. Il lisozima (LYZ), un enzima antimicrobico espresso dai progenitori di Paneth-like nelle cripte intestinali piccole Bevins e Salzman (2011), è stato ampiamente distribuito nell’epitelio degli IO trattati con PBS come abbiamo descritto in precedenza (Spenceet al., 2011) (Figura 3D). Tuttavia, negli IIO con colonizzazione di E. coli, l’espressione LYZ era limitata a distinti cluster di cellule epiteliali e, in particolare, non si è mai sovrapposta alla colorazione DPPIV(Figura 3D). Dato che i marcatori Paneth Cell bona f ide (cioè DEFA5) non sono stati osservati in nessun HIO, è probabile che l’espressione LYZ stia segnando una popolazione di cellule simile a quella dei progenitori. Presi insieme, questi esperimenti indicano che la colonizzazione di E. coli induce un sostanziale ma transitorio aumento del tasso di proliferazione epiteliale seguito da una riduzione e ridistribuzione dei progenitori epiteliali proliferanti e la differenziazione di una popolazione di cellule che esprimono gli enzimi del boarder degli enterociti del piccolo intestino per un periodo di 2-4 giorni.

Figura 3-figure supplemento 1.Figura 3—figura 1. La colonizzazione batterica determina un aumento transitorio della proliferazione epiteliale e della maturazione degli enterociti.2. Pathview(Luo e Brouwer, 2013) trama del percorso KEGG(Kanehisa e Goto, 2000) che mostra il ciclo mitotico delle cellule (‘HSA 04110’) sovrapposto con i dati di espressione RNA-seq corrispondenti al log2 -trasformati cambiamento piega nell’espressione dei trascritti del ciclo cellulare da HIOs microiniezione con E. coli rispetto a PBS-iniezione HIOs a 48 h post-microiniezione . (A) Micrografie confocali rappresentative di IO iniettati con PBS o104 CFU E. coli str. ECOR2 a 24-96 ore dopo la microiniezione e colorati con indicatori fluorescenti per EdU+DNA, E-caderina o nuclei (DAPI) come indicato nelle etichette della figura . Tutti gli HIO sono stati esposti a 10 μM EdU a 22 ore dopo la microiniezione e l’EdU è stato rimosso a 24 ore. I pannelli sono rappresentativi di 4 IOO per punto temporale per condizione di trattamento.(B) Quantificazione del numero di cellule epiteliali EdU-positive e Ki67-positive (cellule E-caderina + cellule) per 10X campo di microscopia confocale. Un campo di microscopia confocale 10X composto da 200-1000 cellule epiteliali è stato raccolto da ciascuno dei 4 HIO per punto temporale per gruppo di trattamento. Le barre di errore rappresentano l’errore standard della media e i valori p riflettono i risultati di un t-test di uno Studente non accoppiato a due code che confronta gli IO iniettati con gli IO iniettati con PBS in quel punto temporale.(C) Micrografie confocali rappresentative degli IO iniettati con PBS o104 CFU E. coli str. ECOR2 a 24-96 ore dopo la microiniezione e colorato con anticorpi fluorescenti per Sox9, E-caderina o nuclei (DAPI) come indicato nelle etichette della figura. I pannelli sono rappresentativi di 4 HIO per punto temporale per condizione di trattamento.Media di 4 repliche biologiche, ognuna delle quali rappresenta 5-6 HIO raggruppati.

Figura 3-figure supplement 1.Pathview(Luo e Brouwer, 2013) trama del percorso KEGG(Kanehisa e Goto, 2000) che mostra il ciclo mitotico delle cellule (‘HSA 04110’) sovrapposto con i dati di espressione RNA-seq corrispondenti al log2 -trasformazione del cambiamento di piega nell’espressione dei trascritti del ciclo cellulare da HIO microiniettato con E. coli rispetto a HIO iniettati con PBS a 48 ore dopo la microiniezione . Media di 4 repliche biologiche, ognuna delle quali rappresenta 5-6 HIO in pool.
La colonizzazione di E. coli è associata ad una riduzione di O2 luminale
L’epitelio intestinale maturo è caratterizzato da un forte gradiente di ossigeno, che va dall’8% di ossigeno all’interno della parete intestinale a <2% di ossigeno nel lume dell’intestino tenue (Fisheret al., 2013). La riduzione del contenuto di ossigeno nel lume intestinale si verifica durante il periodo perinatale immediato(Gruette et al., 1965), con conseguenti cambiamenti nella fisiologia epiteliale(Glover et al., 2016; Kelly et al., 2015; Colgan et al., 2013; Zeitouni et al., 2016) che contribuisce a plasmare la successiva composizione del microbiota(Schmidt e Kao, 2014; Espey, 2013; Albenberg et al., 2014; Palmer et al., 2007; Koenig et al., 2011). L’analisi della risposta globale trascrizionale all’associazione dell’E. coli nel tessuto intestinale immaturo ha rivelato cambiamenti pronunciati e coordinati nell’espressione genica coerenti con l’insorgenza dell’ipossia(Figura 2C-E). Abbiamo quindi misurato la concentrazione di ossigeno nel lume di controllo HIOs e dopo microiniezione di E. col i vivo utilizzando un optodo di fibra ottica di 50 micron di diametro (Figura 4A-B). Concentrazione di ossigeno di base nel lume dell’organoide era 8,04 ± 0,48%, che è stato significativamente ridotto rispetto ai mezzi di coltura esterni (18,86 ± 0,37%, p = 3,6 × 10-11). A 24 e 48 ore dopo la microiniezione, la concentrazione di ossigeno luminale è stata significativamente ridotta in E. coli-injected HIOs rispetto a PBS-iniezione HIOs (p = 0,04 e p = 5,2 × 10-05, rispettivamente) raggiungendo concentrazioni fino a 1,67 ± 0,62% a 48 ore (Figura 4A). E. coli HIO iniettati sono stati raccolti e CFU sono stati enumerati dal contenuto luminale a 24 e 48 ore dopo la microiniezione. Abbiamo osservato una correlazione negativa altamente significativa tra CFU luminale e la concentrazione di ossigeno luminale dove una maggiore densità di batteri luminali è stata correlata con concentrazioni di ossigeno più basse (r2 = 0,842, p = 6,86 × 10-5; Figura 4B). Infine, al fine di valutare l’ossigenazione relativa nell’epitelio stesso, abbiamo utilizzato una piccola molecola pimonidazolo (PMDZ), che forma coniugati covalenti con gruppi tiolici su proteine citoplasmatiche solo in condizioni di basso ossigeno(Arteel et al., 1998). L’immunochimica fluorescente ha dimostrato un maggiore assorbimento di PMDZ nell’epitelio HIO associato all’E. coli, e negli HIO cresciuti nell’1% di O2 come controllo positivo rispetto agli HIO iniettati con PBS, o HIO iniettati con calore ucciso E. coli a 48 ore dopo la microiniezione(Figura 4C). Così, l’ossigeno luminale ed epiteliale è ridotto a seguito di microiniezione di E. col i nell’HIO, coerente con i dati nei topi che mostrano che l’epitelio in vivo è in un simile stato di basso ossigeno in condizioni fisiologiche normali(Schmidt e Kao, 2014; Kelly et al., 2015; Kim et al., 2017).
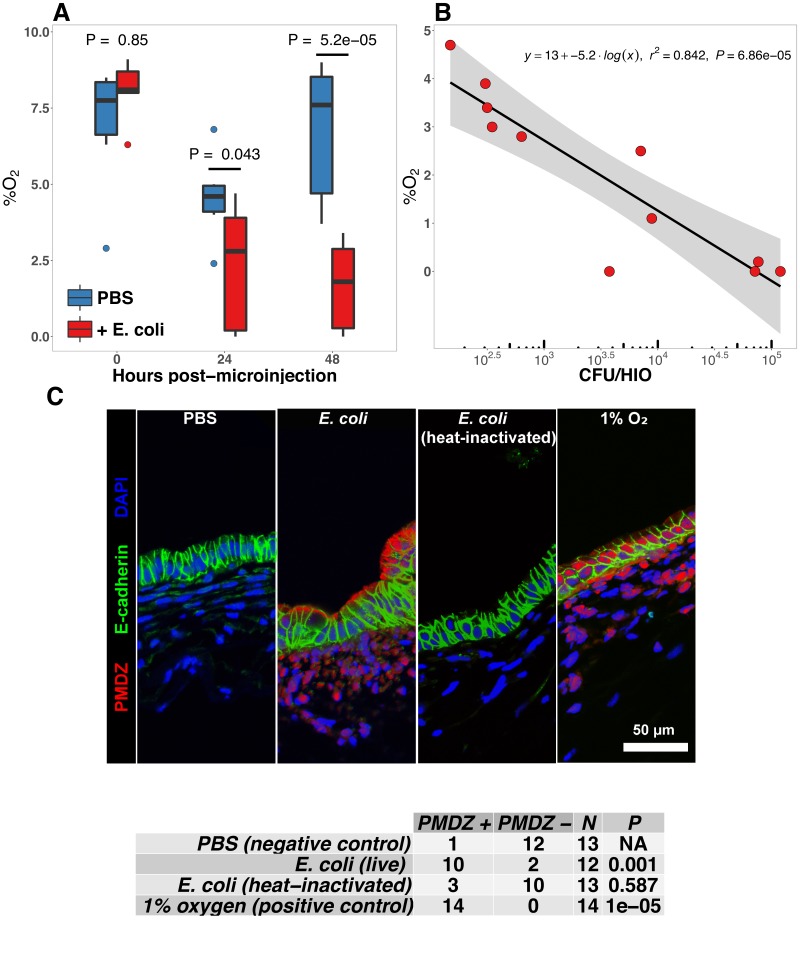
Figura 4.Figura 4. La colonizzazione di E. coli è associata ad una riduzione della concentrazione di ossigeno luminale.(A) Concentrazione di ossigeno luminale in organuli intestinali umani a 0-48 ore dopo la microiniezione con 104 CFU live E. coli. p I valoririflettono i risultati dei test t-test degli studenti non accoppiati con una sola coda per i confronti indicati. N= 6-11 replicare gli IO per gruppo di trattamento per punto temporale.(B) Analisi di regressione lineare della regressione lineare di CFU E. coli luminale per organoide e concentrazione di ossigeno luminale nello stesso organoide 24 ore dopo la microiniezione.(C) Micrografie confocali dell’epitelio dell’HIO in PBS- ed E. coli iniettato in HIO a 48 ore dopo la microiniezione. Le immagini sono rappresentative delle repliche dettagliate nella tabella, con 12-14 repliche di HIO per gruppo di trattamento messe in comune da due esperimenti separati. I singoli HIO sono stati valutati come PMDZ+ o PMDZ- in base alla presenza o assenza, rispettivamente, di coniugati PMDZ come rilevato dalla microscopia immunofluorescente. p-Valori rappresentano i risultati di χ2 contingency test che confrontano la distribuzione di PMDZ+ e PMDZ- HIO nel gruppo trattato con PBS a ciascuna delle altre condizioni.

Figura 4.Figura 4. La colonizzazione di E. coli è associata ad una riduzione della concentrazione di ossigeno luminale.(A) Concentrazione di ossigeno luminale in organuli intestinali umani a 0-48 ore dopo la microiniezione con 104 CFU live E. coli. p I valoririflettono i risultati di non accoppiato a una coda Studenti t-test per i confronti indicati. N= 6-11 replicare gli IO per gruppo di trattamento per punto temporale.(B) Analisi di regressione lineare della regressione lineare di CFU E. coli luminale per organoide e concentrazione di ossigeno luminale nello stesso organoide 24 ore dopo la microiniezione.(C) Micrografie confocali dell’epitelio dell’HIO in PBS- ed E. coli iniettato in HIO a 48 ore dopo la microiniezione. Le immagini sono rappresentative delle repliche dettagliate nella tabella, con 12-14 repliche di HIO per gruppo di trattamento messe in comune da due esperimenti separati. I singoli HIO sono stati valutati come PMDZ+ o PMDZ- in base alla presenza o assenza, rispettivamente, di coniugati PMDZ come rilevato dalla microscopia immunofluorescente. p-Valori rappresentano i risultati di χ2 contingency test che confrontano la distribuzione di PMDZ+ e PMDZ- HIO nel gruppo trattato con PBS a ciascuna delle altre condizioni.
NF-κB integra stimoli microbici complessi e ipossici
La colonizzazione diE. coli provoca una robusta risposta trascrizionale nel tessuto intestinale immaturo(Figura 2) che è associata all’insorgenza dell’esaurimento dell’ossigeno luminale e della relativa ipossia tissutale(Figura 4). Ci siamo posti l’obiettivo di determinare se possiamo assegnare porzioni della risposta trascrizionale all’interazione diretta con i microbi o al successivo esaurimento dell’ossigeno luminale. Nell’analisi RNA-seq(Figura 2), la segnalazione NF-κB è emersa come una delle principali vie coinvolte in questa complessa interazione ospite-microbo, e NF-κB ha dimostrato da altri di agire come mediatore trascrizionale sia del contatto microbico che della risposta all’ipossia tissutale (Rius et al., 2008; Gilmore, 2006; Wullaert et al., 2011). L’ontologia genica e l’analisi del percorso di REACTOME hanno mostrato che i componenti di segnalazione NF-κB sono anche altamente up-regolati a seguito di microiniezione di E. coli in HIO (Figura 2C e Figura 5-figure supplement 1A). Così, abbiamo valutato il ruolo della segnalazione NF-κB nella risposta trascrizionale associata al contatto microbico e la risposta associata all’ipossia utilizzando l’inibitore altamente selettivo dell’IKKβ SC-514 (Kishoreet al., 2003; Litvak et al., 2009) per inibire la fosforilazione e l’attivazione del fattore di trascrizione p65 (Figura 5-figuresupplement 1B). Un’altra serie di HIO è stata contemporaneamente trasferita in una camera ipossica e coltivata in 1% di O2 con e senza SC-514. A 24 ore dopo il trattamento, gli HIO sono stati raccolti per l’isolamento dell’RNA e l’RNA-seq. Abbiamo ideato uno schema sperimentale che ci ha permesso di analizzare i contributi relativi di contatto microbico e ipossia luminale microbica associata a ipossia nella risposta trascrizionale all’associazione con E. coli vivo(Figura 5A e Figura 5-figure supplement 1C). In primo luogo, abbiamo identificato un insieme di geni significativamente up-regolato (log2FC > 0 e FDR-adjusted p-value < 0,05) per microiniezione di E. coli vivo o di calore inattivato E. coli (geni dipendenti da contatto ). Da questo set di geni, abbiamo identificato un sottoinsieme che è stato soppresso dalla presenza dell’inibitore NF-κB SC-514 durante l’associazione con E. coli vivo o termoinattivato (log2FC < 0 e FDR-adjusted p-value < 0.05; Gene Set I, Figura 5B). Quindi, Gene Set I rappresenta la risposta trascrizionale dipendente da NF-κB alla E. coli viva o morta. I geni indotti da E. col i vivi o inattivati dal calore ma non soppressi da SC-514 sono stati considerati indipendenti da NF-κB (Gene Set III, Figura 5B). Allo stesso modo, abbiamo confrontato i geni comunemente up-regolati per associazione con E. coli vivo e quelli up-regolati in condizioni di cultura 1% O2. Un sottoinsieme di geni indotti da colture di E. col i vivo o di O2 all’1% ma soppressi dalla presenza dell’inibitore NF-κB è stato identificato come risposta trascrizionale NF-κB-dipendente dall’ipossia associata all’ipossia (Gene Set II, Figura 5B) . I geni indotti dall’E. coli vivo o dall’ipossia ma non inibiti dalla presenza dell’inibitore NF-κB sono stati considerati risposte trascrizionali indipendenti da NF-κB all’ipossia associata al microbo (Gene Set IV). Gli elenchi dei geni per ogni set di geni si trovano nel file supplementare 1.
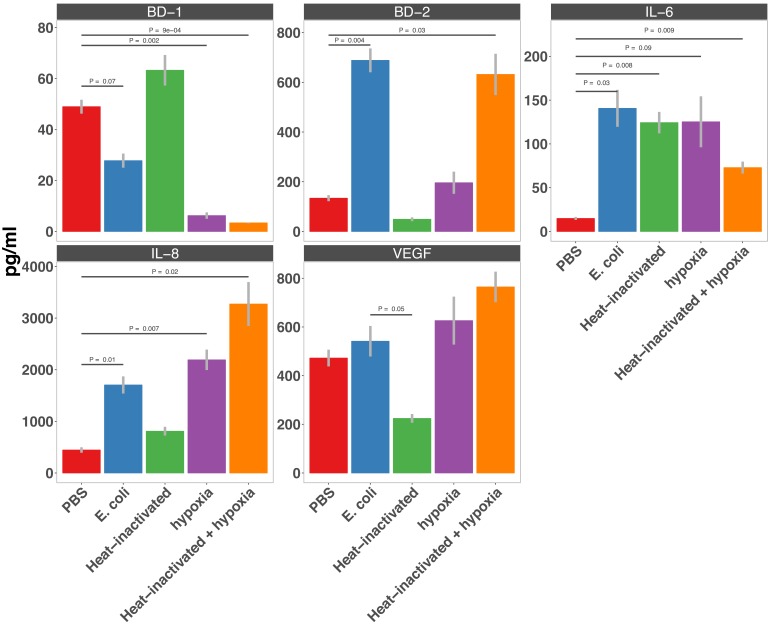
Figura 5-figure supplemento 2.NF-κB integra gli stimoli microbici complessi e gli stimoli ipossici.NF-κB segnale percorso di acticvazione in HIOs.Secretion di AMPs (BD-1 & BD-2), citochine (IL-6 & IL-8), e il pro-angiogenesi fattore di crescita VEGF in HIOs microiniezione con PBS, 10 4 CFU E.(A) Schema di analisi per l’identificazione di insiemi di geni che rappresentano i componenti della risposta trascrizionale all’E. coli vivo che potrebbero essere ricapitolati con E. coli inattivato a caldo (indotta da contatto) o ipossia (ipossia microbica indotta da ipossia) così come i sottoinsiemi di geni indotti attraverso la segnalazione dipendente da NF-κB. Gli HIO sono stati microiniettati con PBS,104 CFU E. col i o una concentrazione equivalente di E. coli inattivati dal calore e coltivati in condizioni standard di coltura cellulare o ipossiche (1% O2, 5%CO2, 94% N2) con e senza 10 μM SC-514.(B) Spargimento trame con sovrapposizione di densità che indica i geni che soddisfano i criteri a priori identificati nel pannello A con un FDR-adjusted p-value di < 0,05 per i confronti elencati sugli assi del grafico.(C) Bar plot della proporzione di geni nella mappatura degli insiemi di geni in ingresso a ciascun percorso dai valori p di arricchimento p dei database GO e REACTOME per ciascuno degli insiemi di geni identificati nel pannello A. I percorsi con i valori p di arricchimento > 0,01 sono stati esclusi dal plot. I risultati rappresentano N= 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO raggruppati e trattati in modo identico.(A) Rappresentazione in Heatmap dell’espressione normalizzata di RNA-seqgene normalizzato dipendente dal tempo per i componenti del percorso NF-κB in HIO a 0-96 ore dopo la microiniezione.(B) Western blot di p65 fosforilato p65 e p65 totale in lisati cellulari da HIOs microiniettato con PBS o vivo E. coli e trattati con IKKβ inibitore SC-514 (1 μM) come indicato in figura.(C) log2-transformato fold-change in espressione genica relativa ai controlli PBS-iniezione per tutte le sette condizioni sperimentali esaminati in questa serie di esperimenti: vivo E. col i + / – SC-514, calore ucciso E. coli + / – SC-514, e la cultura ipossica + / – SC-514, e PBS + SC-514. I geni espressi in modo differenziale (FDR regolato < 0,05) sono indicati in rosso (up-regolato) o blu (down-regolato). Media di tre a cinque repliche per condizione sperimentale.(D) Top 10 percentile da FDR-adjusted p-valore p di gruppi di geni sovrarappresentati dai database GO, KEGG, e REACTOME in geni che sono stati significativamente up- o down-regolato dal trattamento con SC-514 solo rispetto al PBS-iniezione HIO di controllo HIOs.(E) (Copiato dalla Figura 5 per facilità di riferimento) Diagramma di Venn che mostra il numero di geni condivisi tra Gene Set I, Gene Set II, e l’insieme di geni che sono significativamente down-regolati in HIOs PBS-injected trattati con SC-514 rispetto a HIOs di controllo PBS-injected.(F) Top 10 percentile da FDR-adjusted p-value dei set di geni sovrarappresentati dai database GO, KEGG, e REACTOME nei geni che sono stati condivisi tra il Gene Set I o II e l’insieme di geni che sono significativamente down-regolati in HIO trattati con SC-514 relativi agli HIO di controllo PBS-injected.coli, o concentrazione anequivalente di E. coli inattivato a caldo e coltivato in condizioni standard di coltura cellulare o in condizioni ipossiche (1% O2 , 5%CO2, 94% N 2) per 24 ore, come misurato da ELISA. N = 7-8 HIO per condizione.
In seguito all’identificazione di questi quattro insiemi di geni, abbiamo poi applicato l’analisi della sovrarappresentazione utilizzando i database dei percorsi GO e REACTOME per identificare i percorsi arricchiti per ciascuno dei quattro insiemi di geni, ottenendo quattro modelli chiaramente distinguibili di arricchimento dei percorsi genici(Figura 5C). Il contatto con l’E. coli vivo o attivato dal calore è sufficiente a promuovere l’espressione dei geni coinvolti nel mantenimento dell’integrità della barriera epiteliale e nella produzione di mucina, un effetto che viene soppresso in presenza dell’inibitore NF-κB. Inoltre, i principali percorsi di sviluppo, tra cui la morfogenesi epiteliale, lo sviluppo del tratto digestivo e l’espressione degli enzimi digestivi, sembrano essere guidati principalmente dall’associazione batterica e sono in gran parte dipendenti da NF-κB. Una robusta difesa innata e adattiva richiede sia il contatto batterico che l’ipossia, con alcuni geni associati all’elaborazione dell’antigene e alla segnalazione delle citochine che sono dipendenti da NF-κB (Gene Set II) e altri associati a set di geni NF-κB-independenti (Gene Set III e IV). I geni associati ai peptidi antimicrobici defensinici sono stati arricchiti solo nel set di geni NF-κB-dipendenti dall’ipossia (Gene Set IV), suggerendo che i peptidi antimicrobici sono regolati da meccanismi che sono distinti da altri aspetti dell’integrità della barriera epiteliale come le mucine e le giunzioni epiteliali (Gene Set I). I componenti di segnalazione TLR sono stati ampiamente potenziati dall’E. coli vivo e associati sia al contatto microbico che all’ipossia erano in gran parte indipendenti da NF-κB (Gene Set III e IV). C’era una notevole firma trascrizionale che suggeriva un adattamento metabolico e mitocondriale ai batteri che era indipendente da NF-κB e principalmente guidato dal contatto batterico piuttosto che dall’ipossia (Gene Set III).
Per interrogare i cambiamenti trascrizionali influenzati dall’esposizione a SC-514, abbiamo esaminato i set di geni sovrarappresentati dai database GO e REACTOME in geni che erano significativamente up- o down-regolati dal trattamento con SC-514 da solo(Figura 5-figure supplement 1C e D) . In particolare, SC-514 da solo non sembra avere un forte effetto sui percorsi identificati nella Figura 5C come risposte chiave NF-κB-dipendenti dal contatto batterico e/o ipossia. In Figura 5-figure supplement 1E, abbiamo esaminato il grado di sovrapposizione tra Gene Set I, Gene Set II, e l’insieme dei geni che sono significativamente down-regolati in PBS-injected HIOs trattati con SC-514. Questa analisi dimostra che la maggior parte dei geni del Set I e del Set II non sono significativamente down-regolati negli IO iniettati in PBS trattati con SC-514. Gli effetti più significativi di SC-514 solo tra i geni del Set I e del Set II sono legati al metabolismo, allo stato redox e alla dinamica ribosomica(Figura 5-figure supplement 1F). Quindi, l’effetto di SC-514 da solo non può spiegare i cambiamenti NF-κB-dipendenti nella difesa innata e adattiva, l’integrità della barriera epiteliale, l’angiogenesi e la segnalazione dell’ipossia, o lo sviluppo intestinale a seguito di contatto batterico e/o ipossia durante la colonizzazione.
Infine, abbiamo anche esaminato il ruolo del contatto microbico e ipossia indotta da colonizzazione cambiamenti indotti in AMP, citochina, e la secrezione del fattore di crescita utilizzando ELISA(Figura 5-figure supplemento 2). Coerentemente con i risultati dei dati RNA-seq, questi risultati indicano che ci sono diverse risposte al contatto batterico e ipossia. Abbiamo osservato casi in cui le citochine sono state indotte da contatto microbico o ipossia da sola (IL-6), altri casi in cui l’ipossia sembrava essere lo stimolo dominante (BD-1), e un terzo paradigma normativo in cui la risposta all’E. coli vivo risulta evidentemente dall’influenza cumulativa del contatto batterico e dell’ipossia (BD-2, IL-8, VEGF). Presi insieme, questa analisi dimostra che l’associazione dell’epitelio intestinale immaturo con l’E. coli vivo si traduce in un’interazione complessa tra contatto microbico e ipossia microbica associata all’espressione genica indotta da ipossia e alla secrezione di proteine.

Figura 5-figure supplemento 2.NF-κB integra gli stimoli microbici complessi e gli stimoli ipossici.NF-κB segnale percorso di acticvazione in HIOs.Secrezione di AMPs (BD-1 & BD-2), citochine (IL-6 & IL-8), e il pro-angiogenesi fattore di crescita VEGF in HIOs microiniezione con PBS, 10 4 CFU E.(A) Schema di analisi per l’identificazione di insiemi di geni che rappresentano i componenti della risposta trascrizionale all’E. coli vivo che potrebbero essere ricapitolati con E. coli inattivato a caldo (indotta da contatto) o ipossia (ipossia microbica indotta da ipossia) così come i sottoinsiemi di geni indotti attraverso la segnalazione dipendente da NF-κB. Gli HIO sono stati microiniettati con PBS,104 CFU E. col i o una concentrazione equivalente di E. coli inattivati dal calore e coltivati in condizioni standard di coltura cellulare o ipossiche (1% O2, 5%CO2, 94% N2) con e senza 10 μM SC-514.(B) Spargimento trame con sovrapposizione di densità che indica i geni che soddisfano i criteri a priori identificati nel pannello A con un FDR-adjusted p-value di < 0,05 per i confronti elencati sugli assi del grafico.(C) Bar plot della proporzione di geni nella mappatura degli insiemi di geni in ingresso a ciascun percorso dai valori p di arricchimento p dei database GO e REACTOME per ciascuno degli insiemi di geni identificati nel pannello A. I percorsi con i valori p di arricchimento > 0,01 sono stati esclusi dal plot. I risultati rappresentano N= 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO raggruppati e trattati in modo identico.(A) Rappresentazione in Heatmap dell’espressione normalizzata di RNA-seqgene normalizzato dipendente dal tempo per i componenti del percorso NF-κB in HIO a 0-96 ore dopo la microiniezione.(B) Western blot di p65 fosforilato p65 e p65 totale in lisati cellulari da HIOs microiniettato con PBS o vivo E. coli e trattati con IKKβ inibitore SC-514 (1 μM) come indicato in figura.(C) log2-transformato fold-change in espressione genica relativa ai controlli PBS-iniezione per tutte le sette condizioni sperimentali esaminati in questa serie di esperimenti: vivo E. col i + / – SC-514, calore ucciso E. coli + / – SC-514, e la cultura ipossica + / – SC-514, e PBS + SC-514. I geni espressi in modo differenziale (FDR regolato < 0,05) sono indicati in rosso (up-regolato) o blu (down-regolato). Media di tre a cinque repliche per condizione sperimentale.(D) Top 10 percentile da FDR-adjusted p-valore p di gruppi di geni sovrarappresentati dai database GO, KEGG, e REACTOME in geni che sono stati significativamente up- o down-regolato dal trattamento con SC-514 solo rispetto al PBS-iniezione HIO di controllo HIOs.(E) (Copiato dalla Figura 5 per facilità di riferimento) Diagramma di Venn che mostra il numero di geni condivisi tra Gene Set I, Gene Set II, e l’insieme di geni che sono significativamente down-regolati in HIOs PBS-injected trattati con SC-514 rispetto a HIOs di controllo PBS-injected.(F) Top 10 percentile da FDR-adjusted p-value dei set di geni sovrarappresentati dai database GO, KEGG, e REACTOME nei geni che sono stati condivisi tra il Gene Set I o II e l’insieme di geni che sono significativamente down-regolati in HIO trattati con SC-514 relativi agli HIO di controllo PBS-injected.coli, o concentrazione anequivalente di E. coli inattivato a caldo e coltivato in condizioni standard di coltura cellulare o in condizioni ipossiche (1% O2 , 5%CO2, 94% N 2) per 24 ore, come misurato da ELISA. N = 7-8 HIO per condizione.
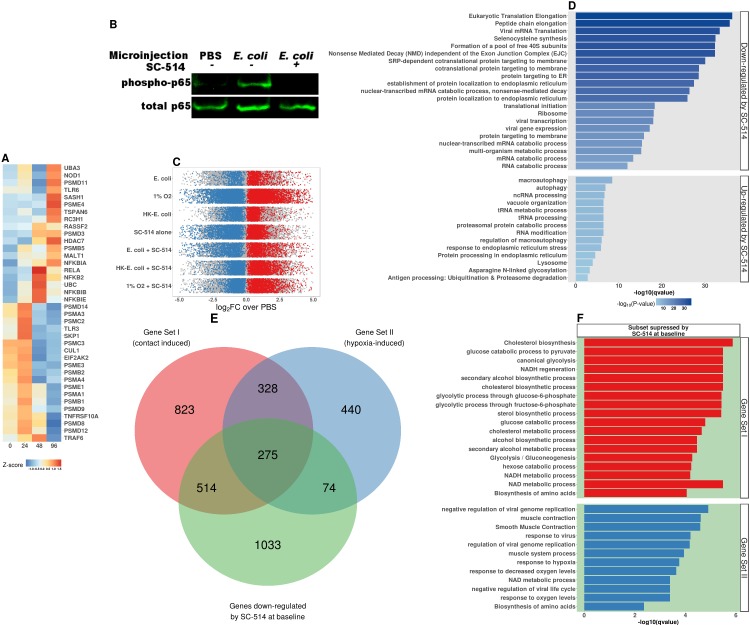
Figura 5-figure supplement 1.NF-κB percorso di segnalazione acticvazione in HIOs.(A) Rappresentazione della mappa termica dell’espressione normalizzata di RNA-seqgene normalizzato dipendente dal tempo per i componenti della via NF-κB in HIO a 0-96 ore dopo la microiniezione.(B) Western blot di p65 fosforilato p65 e p65 totale in lisati cellulari da HIOs microiniettato con PBS o vivo E. coli e trattati con IKKβ inibitore SC-514 (1 μM) come indicato in figura.(C) log2-transformato fold-change in espressione genica relativa ai controlli PBS-iniezione per tutte le sette condizioni sperimentali esaminati in questa serie di esperimenti: vivo E. col i + / – SC-514, calore ucciso E. coli + / – SC-514, e la cultura ipossica + / – SC-514, e PBS + SC-514. I geni espressi in modo differenziale (FDR regolato < 0,05) sono indicati in rosso (up-regolato) o blu (down-regolato). Media di tre a cinque repliche per condizione sperimentale.(D) Top 10 percentile da FDR-adjusted p-valore p di gruppi di geni sovrarappresentati dai database GO, KEGG, e REACTOME in geni che sono stati significativamente up- o down-regolato dal trattamento con SC-514 solo rispetto al PBS-iniezione HIO di controllo HIOs.(E) (Copiato dalla Figura 5 per facilità di riferimento) Diagramma di Venn che mostra il numero di geni condivisi tra Gene Set I, Gene Set II, e l’insieme di geni che sono significativamente down-regolati in HIOs PBS-injected trattati con SC-514 rispetto a HIOs di controllo PBS-injected.(F) Top 10 percentile da FDR-adjusted p-value dei set di geni sovrarappresentati dai database GO, KEGG, e REACTOME nei geni che sono stati condivisi tra il Gene Set I o II e l’insieme di geni che sono significativamente down-regolati in HIO trattati con SC-514 relativi agli HIO di controllo PBS-injected.

Figura 5-figure supplement 2.Figura 5—supplemento di figura 2. Secrezione di AMP (BD-1 & BD-2), citochine (IL-6 & IL-8), e il fattore di crescita pro-angiogenesi VEGF negli HIO microiniettati con PBS, 10 4 CFU E.coli, o concentrazione anequivalente di E. coli inattivato a caldo e coltivato in condizioni standard di coltura cellulare o in condizioni ipossiche (1% O2 , 5%CO2, 94% N 2) per 24 ore, come misurato da ELISA. N = 7-8 HIO per condizione.
La colonizzazione batterica favorisce la secrezione di peptidi antimicrobici
I peptidi antimicrobici (AMP) sono effetti chiave per la difesa innata delle superfici epiteliali(Muniz et al., 2012) e agiscono per inibire la crescita microbica attraverso la lisi diretta della parete cellulare batterica e la modulazione del metabolismo batterico(Ganz, 2003; Bevins e Salzman, 2011; O’Neil e O’Neil, 2003; Vora et al., 2004; Brogden, 2005). L’espressione del gene Defensin è altamente up-regolata a seguito di microiniezione di E. coli in HIO(Figure 2D-E e e 4C). Utilizzando un database di AMP noti(Wang et al., 2016) per interrogare i nostri set di dati RNA-seq, abbiamo scoperto che diversi AMP sono up-regolati nell’epitelio intestinale immaturo dopo l’associazione di E. coli (Figura 6A). Tra questi, DEFB4A e DEFB4B, geni duplicati che codificano il peptide umano β-defensina 2 (Harderet al., 1997), sono stati i più altamente regolati; altri AMP indotti dall’associazione di E. coli includono i peptidi multifunzionali CCL20, CXCL2, CXCL1, CXCL6, CXCL3, REG3A (Cashet al., 2006), e LTF (Figura 6A). L’analisi dei dati RNA-seq da HIOs microiniettato con E.coli vivo o ucciso a caldo con e senza inibitore NF-κB o coltura di HIOs in condizioni di ipossia aveva indicato che i geni della defensina erano stati arricchiti tra l’insieme dei geni NF-κB-indipendenti indotti dall’ipossia (Figura 5C). Abbiamo esaminato l’espressione DEFB4A in particolare(Figura 6B) e abbiamo trovato che rispetto al trattamento di controllo, microiniezione di E. coli vivo ha portato ad un aumento di 7,38 volte l’espressione DEFB4A normalizzata. Coerentemente con l’idea che l’espressione del DEFB4A è indotta da ipossia e non dipende dalla segnalazione di NF-κB, gli IO trattati con inibitore di NF-κB iniettati con E. coli mostravano ancora un aumento di ~ 8 volte nell’espressione genica e gli IO coltivati per ipossia mostravano un’induzione di ~ 5,5 volte (Figura 6B). D’altra parte, microiniezione con calore inattivato E. coli ha portato a induzione DEFB4A che era significativamente inferiore rispetto alla microiniezione con E. coli vivo (p = 0,007. Un modello simile di espressione è stato osservato per il DEFB4B(Figura 6-figure supplement 1).
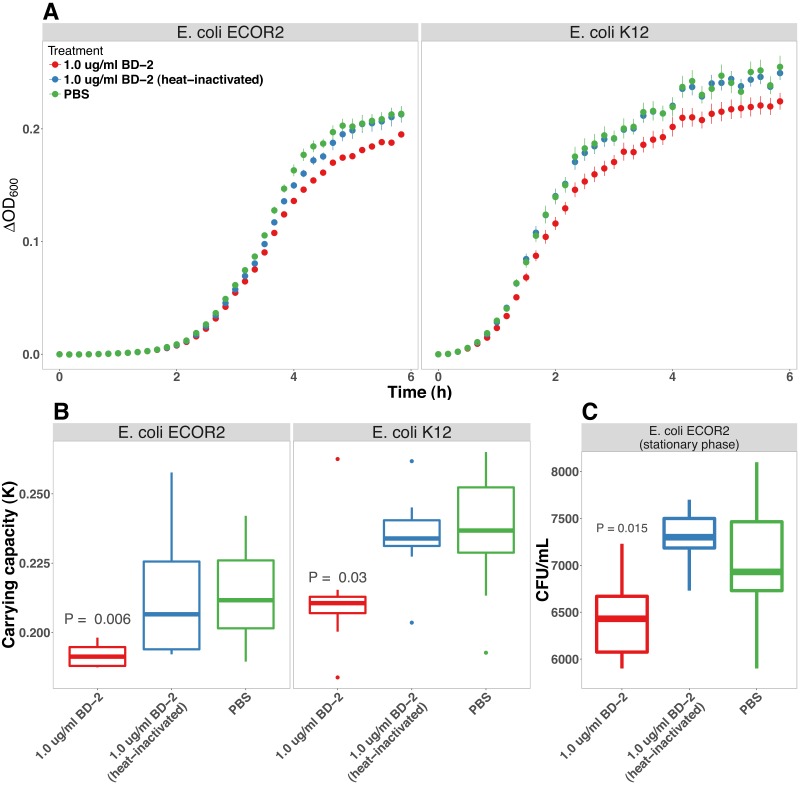
Figura 6-figure supplemento 2.La colonizzazione batterica promuove la secrezione di peptidi antimicrobici.il cambiamento di piega normalizzato nell’espressione del DEFB4B, un gene duplicato che codifica il peptide umano β-defensina 2 (BD-2), in ciascuna delle condizioni indicate in relazione al trattamento di controllo della PBS.il BD-2 inibisce la crescita dell’E. coli in vitro.(A) Cambiamento normalizzato della piega nell’espressione del gene del peptide antimicrobico (AMP) in HIO associati a E. coli a 24 ore rispetto al trattamento di controllo del PBS.(B) Cambiamento di piega normalizzata in espressione di DEFB4A, il gene che codifica il peptide umano β-defensina 2 (BD-2), in ciascuna delle condizioni indicate in relazione al trattamento di controllo PBS. I risultati nei pannelli A e B rappresentano N= 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO in pool e trattati in modo identico.(C) Concentrazione del peptide BD-2 nel supernatante in coltura a 24 ore, misurata con ELISA in colture HIO trattate come indicato. N= 10-14 HIO trattati individualmente per condizione di trattamento con dati combinati da tre esperimenti di replicazione indipendenti.(D) Densità ottica (600 nm) di colture in sospensione di E. coli integrata con PBS o BD-2 a intervalli di 10 minuti su un periodo di 18 ore a 37.(E) Capacità di trasporto (K) di media integrati con varie concentrazioni di BD-2 derrivati dalle curve di crescita presentate nel pannello D. N= 8 repliche biologiche per gruppo di trattamento per i pannelli D ed E. p-Valori rappresenta i risultati di uno Studente a due code t-test per i confronti indicati.Concentrazione di peptide BD-2 in coltura supernatante a 24 ore come misurato da ELISA in colture HIO trattate come indicato. p-Valori p rappresenta i risultati di uno Studente a due code t-test per il confronto indicato.(A) Densità ottica (600 nm) di E. coli str. ECOR2 e E. col i str. K12suspensione colture integrate con PBS, BD-2 (1 μg/ml), o BD-2 inattivato a calore a intervalli di 10 minuti per un periodo di 6 ore a 37◦C.(B) Capacità di trasporto(K) di colture di E. coli incubate con PBS, BD-2 (1 μg/ml), o BD-2 inattivato a calore durante la crescita in fase di log (pannello A). p-Valori rappresenta i risultati di un t-test di uno studente a una coda rispetto al trattamento con PBS. Per i pannelli A e B, N = 8 per condizione sperimentale per ogni ceppo di E. coli.(C) CFU/ml di fase stazionaria tardiva E. col i str. Colture ECOR2 diluite in PBS e integrate con BD-2 (1 μg/ml) o BD-2 inattivato a caldo per 6 ore a 37◦C.N = 8 (BD-2 e BD-2 inattivato a caldo) o 16 (PBS).
Abbiamo anche esaminato la secrezione di β-defensina 2 peptide umano (BD-2) nel supernatante di E. coli-associati HIOs (Figura 2E e Figura 6C). BD-2 secrezione è stata aumentata 3,4 volte a 24 ore dopo la microiniezione di E. coli (p = 2,7 × 10-8). Tuttavia, l’inattivazione termica dell’E. coli o l’aggiunta di inibitore NF-κB ha portato alla soppressione della secrezione di BD-2 rispetto all’E. coli vivo (p=0,00051 e 1,6 × 10-6, rispettivamente).
Per determinare se i livelli di BD-2 prodotto da HIO e secreto nei media erano sufficienti a ritardare la crescita batterica, abbiamo testato l’effetto del BD-2 a concentrazioni che ricapitolano lo stato di base nel HIO (~ 0,1 μg/ml) e dopo microiniezione con E.coli (~ 1 μg/ml ) sulla crescita in vitro di E. coli oltre 18 ore (Figura 6D). Anche se c’era poco effetto sulla densità di E. coli durante la crescita iniziale log-fase, BD-2 ha ridotto la quantità di tempo i batteri spesi in fase di crescita log-fase, e la densità di E. coli è stata significativamente diminuita nel tempo in mezzi di crescita batterica integrato BD-2 (p = 0,001), con conseguente significativa diminuzione della capacità effettiva di carico in vitro, o densità massima della popolazione(Figura 6E, p = 8 × 10-4). Inoltre, le concentrazioni di BD-2 coerente con quelli trovati in HIO / E. coli supernatante (1 mg / ml) è stato significativamente più inibitorio di bassa concentrazione BD-2 (0,1 mg / ml) nel nostro test di crescita in vitro (p = 0,013). Ulteriori dati suggeriscono che l’attività inibitoria del BD-2 in vitro non è specifica per l ‘E. coli str. ECOR2 e dipende dal mantenimento della struttura della proteina BD-2, poiché il BD-2 inibisce in modo simile la crescita di E. coli str. K12, e il BD-2 inattivato dal calore ha perso questi effetti inibitori(Figura 6-figure supplement 2). Da questa serie di esperimenti, si conclude che la colonizzazione dell’E. coli promuove una migliore espressione delle AMP, compreso il BD-2, a concentrazioni sufficienti a sopprimere la crescita microbica.

Figura 6-figure supplement 2.La colonizzazione batterica promuove la secrezione di peptidi antimicrobici.il cambiamento di piega normalizzato nell’espressione del DEFB4B, un gene duplicato che codifica il peptide umano β-defensina 2 (BD-2), in ciascuna delle condizioni indicate in relazione al trattamento di controllo PBS.il BD-2 inibisce la crescita dell’E. coli in vitro.(A) Cambiamento normalizzato della piega nell’espressione del gene del peptide antimicrobico (AMP) in HIO associati a E. coli a 24 ore rispetto al trattamento di controllo del PBS.(B) Cambiamento di piega normalizzata in espressione di DEFB4A, il gene che codifica il peptide umano β-defensina 2 (BD-2), in ciascuna delle condizioni indicate in relazione al trattamento di controllo PBS. I risultati nei pannelli A e B rappresentano N= 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO in pool e trattati in modo identico.(C) Concentrazione del peptide BD-2 nel supernatante in coltura a 24 ore, misurata con ELISA in colture HIO trattate come indicato. N= 10-14 HIO trattati individualmente per condizione di trattamento con dati combinati da tre esperimenti di replicazione indipendenti.(D) Densità ottica (600 nm) di colture in sospensione di E. coli integrata con PBS o BD-2 a intervalli di 10 minuti su un periodo di 18 ore a 37.(E) Capacità di trasporto (K) di media integrati con varie concentrazioni di BD-2 derrivati dalle curve di crescita presentate nel pannello D. N= 8 repliche biologiche per gruppo di trattamento per i pannelli D ed E. p-Valori rappresenta i risultati di uno Studente a due code t-test per i confronti indicati.Concentrazione di peptide BD-2 in coltura supernatante a 24 ore come misurato da ELISA in colture HIO trattate come indicato. p-Valori p rappresenta i risultati di uno Studente a due code t-test per il confronto indicato.(A) Densità ottica (600 nm) di E. coli str. ECOR2 e E. col i str. K12suspensione colture integrate con PBS, BD-2 (1 μg/ml), o BD-2 inattivato a calore a intervalli di 10 minuti per un periodo di 6 ore a 37◦C.(B) Capacità di trasporto(K) di colture di E. coli incubate con PBS, BD-2 (1 μg/ml), o BD-2 inattivato a calore durante la crescita in fase di log (pannello A). p-Valori rappresenta i risultati di un t-test di uno studente a una coda rispetto al trattamento con PBS. Per i pannelli A e B, N = 8 per condizione sperimentale per ogni ceppo di E. coli.(C) CFU/ml di fase stazionaria tardiva E. col i str. Colture ECOR2 diluite in PBS e integrate con BD-2 (1 μg/ml) o BD-2 inattivato a caldo per 6 ore a 37◦C.N = 8 (BD-2 e BD-2 inattivato a caldo) o 16 (PBS).
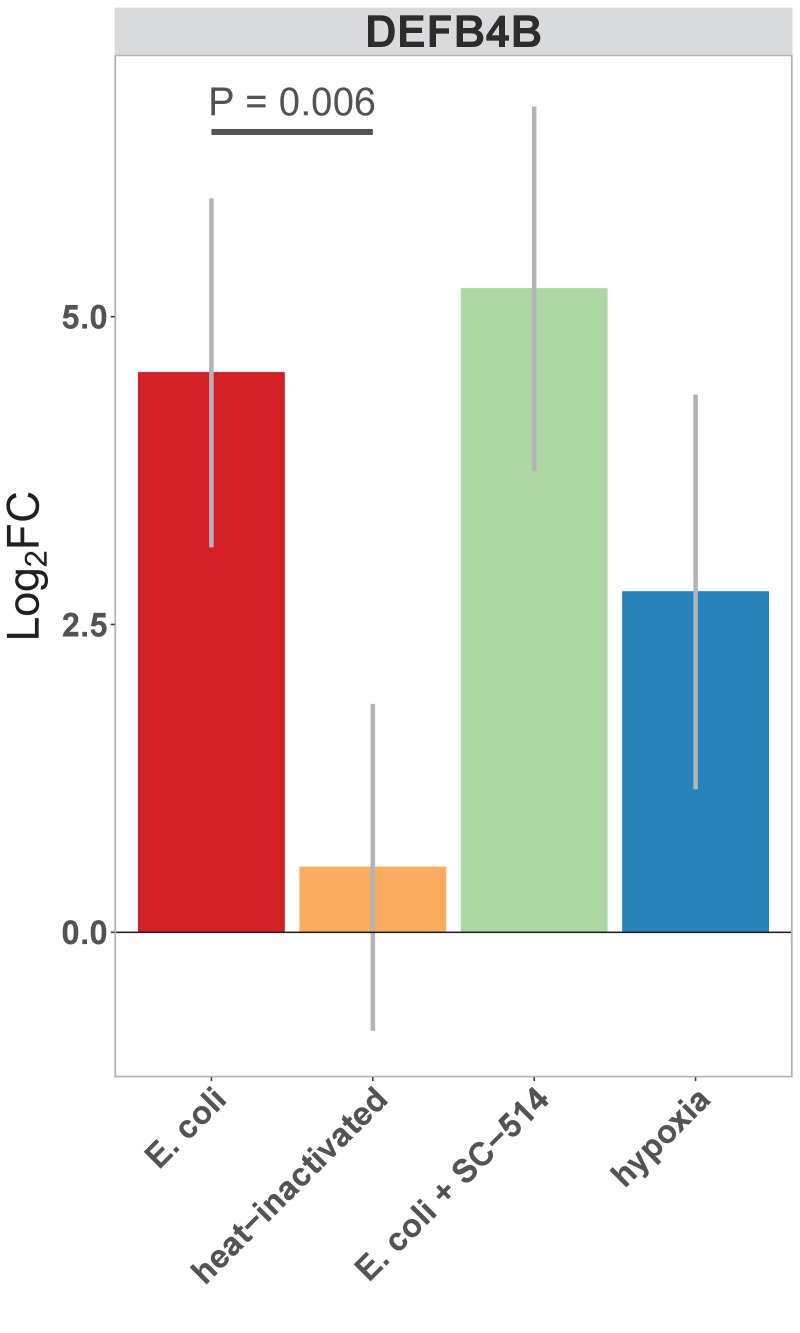
Figura 6-figure supplement 1.Cambiamento di piega normalizzato nell’espressione del DEFB4B, un gene duplicato che codifica il peptide umano β-defensina 2 (BD-2), in ciascuna delle condizioni indicate in relazione al trattamento di controllo del PBS.Concentrazione del peptide BD-2 in coltura supernatante a 24 ore come misurato da ELISA in colture HIO trattate come indicato. p-Valori rappresenta i risultati di uno studente a due code t-test per il confronto indicato.

Figura 6-figure supplemento 2.BD-2 inibisce la crescita di E. coli in vitro.(A) Densità ottica (600 nm) di E. col i str. ECOR2 e E. coli str. K12suspensione colture integrate con PBS, BD-2 (1 μg/ml), o BD-2 inattivato a calore a intervalli di 10 minuti per un periodo di 6 ore a 37◦C.(B) Capacità di trasporto(K) di colture di E. coli incubate con PBS, BD-2 (1 μg/ml), o BD-2 inattivato a calore durante la crescita in fase di log (pannello A). p-Valori rappresenta i risultati di un t-test di uno studente a una coda rispetto al trattamento con PBS. Per i pannelli A e B, N = 8 per condizione sperimentale per ogni ceppo di E. coli.(C) CFU/ml di fase stazionaria tardiva E. col i str. Colture ECOR2 diluite in PBS e integrate con BD-2 (1 μg/ml) o BD-2 inattivato a caldo per 6 ore a 37◦C.N = 8 (BD-2 e BD-2 inattivato a caldo) o 16 (PBS).
La colonizzazione batterica favorisce l’espressione delle mucine epiteliali e delle glicotransferasi
Le mucine sono una componente essenziale dell’integrità epiteliale, fungendo da formidabile barriera all’invasione microbica e da deposito per gli AMP secreti(Bergstrom e Xia, 2013; Cornick et al., 2015; Johansson e Hansson, 2016; Kim e Ho, 2010). La sintesi di mucina richiede una serie complessa di modifiche post-traslazionali che aggiungono catene laterali di carboidrati ad alto peso molecolare alla proteina della mucina del nucleo(Varki, 2017). I nostri dati RNA-seq suggeriscono che l’espressione del gene della mucina dipende sia dal contatto batterico che dalla segnalazione NF-κB (Figura 5C). Pertanto, abbiamo esaminato l’espressione dei geni nel controllo e E. coli HIO microiniettato che codificano le proteine del nucleo della mucina così come le glicotransferasi che generano l’ampia varietà di modifiche della mucina post-traslazionale(Figura 7A). Sebbene alcune glicotransferasi siano state aumentate a 24 ore dopo la microiniezione di E. coli, l’espressione delle proteine del nucleo della mucina e molte glicotransferasi hanno raggiunto i livelli di picco a 48 ore dopo l’introduzione di E. coli nel lume dell’HIO(Figura 7A). Periodico Acido-Schiff e colorazione blu alciana (PAS/AB) di sezioni prese da HIO a 48 ore dopo la microiniezione di E. coli rivelano la formazione di un robusto strato di mucina sulla superficie epiteliale apicale composto sia acida (AB-positivo) e neutro (PAS-positivo) componenti glicoproteine, suggerendo una ricca matrice di O-linked mucine, glicosaminoglicani e proteoglicani(Figura 7B-C). È interessante notare che l’associazione di E. coli ha causato un’induzione iniziale di MUC5AC a 48 ore che è stata ridotta di 96 ore(Figura 7A). MUC5AC è più altamente espresso all’interno della mucosa gastrica, ma è stato riportato anche nell’epitelio duodenale(Buisine et al., 1998, 2001; Rodríguez-Piñeiro et al., 2013). D’altra parte, MUC2 è più comunemente associato con il duodeno, ed è aumentato più lentamente, mostrando l’espressione di picco dopo 96 ore di associazione con E. coli(Figura 7A). La co-colorazione degli IO di controllo e degli IO microiniettati con E. col i ha dimostrato la colocalizzazione con l’Ulex europaeus agglutinina I (UEA1), una lectina ad alta specificità per la frazione terminale del fucosio Fucα1-2Gal-R (Figura 7D). Ciò suggerisce che in seguito all’associazione di E. coli, gli IO producono mucine con modificazioni dei carboidrati associate alla colonizzazione batterica in vivo(Cash et al., 2006; Hooper et al., 1999; Goto et al., 2014).
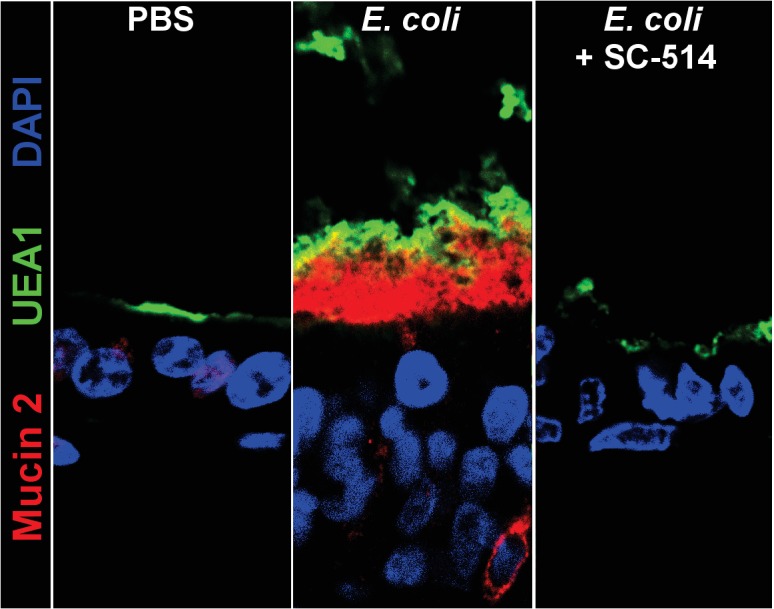
Figura 7-figure supplement 1.Figura 7—figura 1. La colonizzazione batterica promuove l’espressione delle mucine epiteliali e delle glicotransferasi.micrografie confocali rappresentative degli IO trattati come indicato.(A) Heatmap di RNA-seq normalizzato RNA-seq glicotransferasi e mucina conteggi genetici di HIOs associati con E.coli a 0-96 ore post-microiniezione. N= 4 (0 ore), 5 (24 ore), 3 (48 ore) e 4 (96 ore) repliche biologiche costituite da 5-6 HIO raggruppati per ogni replicato.(B) Colorazione periodica acido-Schiff e Alcian Blue (PAS-AB) di HIO di controllo o HIO microiniettato con E. coli e coltivato per 48 ore con ingrandimento 10X.C) Epitelio di HIO da HIO di controllo o HIO microiniettato con E. col i e coltivato per 48 ore colorato con H e E, AB, PAS, o PAS-AB e ripreso al microscopio a luce 100X.D) Micrografia confocale di epitelio HIO da un HIO di controllo o da un HIO microiniettato con E. coli e coltivato per 48 ore. I nuclei sono colorati di blu con DAPI, e le proteine fluorescenti antibody-labeled E-cadhereina e Mucin 5 AC sono pseudocolorati in bianco o rosso, rispettivamente. UEA1 lectina è usato per etichettare il carboidrato moiety Fucα1-2Gal-R, che è pseudo colorato in verde. 60X ingrandimento ottico.(E) Heatmap di RNA-seq normalizzato glicotransferasi e mucina glicotransferasi conteggi genetici di HIOs associati con vivo o calore inattivato E. coli, E. coli+ NF-κB inibitore (SC-514) o HIOs coltivate in condizioni di ipossia per 24 ore. I risultati rappresentano la media di N= 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO riuniti e trattati in modo identico.(F) Colorazione PAS-AB degli IO trattati come indicato nelle etichette della figura per un ingrandimento di 24 ore 10X. Le immagini istologiche e immunofluorescenti nei pannelli B-D e F sono rappresentative di tre o più esperimenti indipendenti, ciascuno costituito da 5-10 IO per gruppo di trattamento.Immuno-colorazione fluorescente pseudo-colorazione applicata come indicato nella legenda della figura. Ingrandimento ottico 40X con zoom digitale 3X.
RNA-seq dati suggeriscono che le mucine O-linked mucine erano altamente arricchito tra il sottoinsieme dei geni indotti da contatto batterico in un sottoinsieme di geni indotta da contatto batterico in un NF-κB-dipendente modo (Figura 5). Abbiamo esaminato questo fenomeno a livello di singoli geni glicosiltransferasi e mucina(Figura 7E). La trascrizione indotta da E. coli di mucine e glicosiltransferasi(Figura 7E) e la secrezione di mucina(Figura 7-figure supplement 1) è stata soppressa in presenza dell’inibitore NF-κB SC-514. Inoltre, la coltura di HIO in condizioni di ipossia non è stata sufficiente a promuovere la trascrizione dei geni coinvolti nella sintesi della mucina(Figura 7E). Questo risultato è stato confermato con la colorazione PAS/AB di HIOs microiniettato con PBS, E. coli vivo o termoinattivato, o coltivato in condizioni di ipossia per 24 ore, dove il contatto batterico ha favorito la formazione di uno strato di muco, mentre la microiniezione di PBS o la coltura in condizioni di ipossia non lo ha fatto(Figura 7F). Presi nel loro insieme, questi risultati indicano che l’associazione dell’epitelio intestinale immaturo con l’E. coli promuove una robusta secrezione di muco attraverso un meccanismo dipendente da NF-κB e che l’ipossia da sola non è sufficiente a ricapitolare la produzione di muco indottadall’E. coli.

Figura 7-figure supplement 1.Figura 7—figura 1. Colonizzazione batterica promuove l’espressione di mucine epiteliali e glicotransferasi.micrografie confocali rappresentative di HIO trattati come indicato.(A) Heatmap di RNA-seq normalizzato RNA-seq glicotransferasi e mucina conteggi genetici di HIOs associati con E.coli a 0-96 ore post-microiniezione. N= 4 (0 ore), 5 (24 ore), 3 (48 ore) e 4 (96 ore) repliche biologiche costituite da 5-6 HIO raggruppati per ogni replicato.(B) Colorazione periodica acido-Schiff e Alcian Blue (PAS-AB) di HIO di controllo o HIO microiniettato con E. coli e coltivato per 48 ore con ingrandimento 10X.C) Epitelio di HIO da HIO di controllo o HIO microiniettato con E. col i e coltivato per 48 ore colorato con H e E, AB, PAS, o PAS-AB e ripreso al microscopio a luce 100X.D) Micrografia confocale di epitelio HIO da un HIO di controllo o da un HIO microiniettato con E. coli e coltivato per 48 ore. I nuclei sono colorati di blu con DAPI, e le proteine fluorescenti antibody-labeled E-cadhereina e Mucin 5 AC sono pseudocolorati in bianco o rosso, rispettivamente. UEA1 lectina è usato per etichettare il carboidrato moiety Fucα1-2Gal-R, che è pseudo colorato in verde. 60X ingrandimento ottico.(E) Heatmap di RNA-seq normalizzato glicotransferasi e mucina glicotransferasi conteggi genetici di HIOs associati con vivo o calore inattivato E. coli, E. coli+ NF-κB inibitore (SC-514) o HIOs coltivate in condizioni di ipossia per 24 ore. I risultati rappresentano la media di N= 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO riuniti e trattati in modo identico.(F) Colorazione PAS-AB degli IO trattati come indicato nelle etichette della figura per un ingrandimento di 24 ore 10X. Le immagini istologiche e immunofluorescenti nei pannelli B-D e F sono rappresentative di tre o più esperimenti indipendenti, ciascuno costituito da 5-10 IO per gruppo di trattamento.Immuno-colorazione fluorescente pseudo-colorazione applicata come indicato nella legenda della figura. Ingrandimento ottico 40X con zoom digitale 3X.

Figura 7-figure supplemento 1.Micrografie confocali rappresentative di HIO trattati come indicato.2. Immunostaining fluorescente pseudo-colorazione applicata come indicato nella legenda figura. 40X ingrandimento ottico con zoom digitale 3X.
La segnalazione NF-κB è necessaria per il mantenimento dell’integrità della barriera a seguito della colonizzazione batterica
Avendo stabilito che l’epitelio intestinale immaturo in HIO(Figura 1-figure supplement 1) può essere stabilmente associato con E. coli non patogeno ( Figura1), con conseguenti ampi cambiamenti nell’attività trascrizionale (Figura2) e portando ad un’elevata produzione di AMP (Figura6) e alla secrezione di muco epiteliale (Figura7), abbiamo ipotizzato che questi cambiamenti nell’espressione genica e proteica avrebbero conseguenze funzionali per la barriera epiteliale immaturo. L’analisi RNA-seq ha dimostrato un’ampia up-regolazione della trascrizione nei geni coinvolti nella formazione della giunzione aderente e altre interazioni cellula-cellula in HIO dopo la microiniezione con E. coli vivo che è stato inibito in presenza di NF-κB inibitore SC-514 (Figura 8A). Abbiamo utilizzato un test di permeabilità FITC-dextran modificato(Leslie et al., 2015) e l’imaging in tempo reale di culture HIO vivo per misurare la funzione di barriera epiteliale in HIO microiniettato con PBS, E. coli vivo, o vivo E. coli+SC-514 a 24 ore dopo la microiniezione (Figura 8B). Mentre gli HIO microiniettati con PBS o E. coli hanno mantenuto il 94,1 0,3% della fluorescenza FITC-dextran durante il periodo di saggio di 20 ore, gli HIO microiniettati con E. coli in coltura in presenza di SC-514 hanno mantenuto solo il 45,5 ± 26,3% del segnale fluorescente (p=0,02; Figura 8B). Abbiamo anche misurato il tasso di traslocazione batterica attraverso l’epitelio HIO, che ha portato a mezzi di coltura contaminati(Figura 8C). HIO microiniettato con E. coli e trattato con SC-514 sono stati confrontati con E. coli microiniettato HIO trattati con veicolo (controlli DMSO) e PBS microiniettato controlli in 7 giorni in coltura. Gli IO associati con E. coli +SC-514 hanno mostrato una rapida insorgenza di traslocazione batterica entro 2-3 giorni, con la traslocazione batterica rilevata nel 96% degli IO trattati con SC-514 entro il giorno 7 rispetto al 23% degli IO microiniettati con E. coli e trattati con DMSO (P=<2 × 10-16; Figura 8C). Pertanto, bloccando la segnalazione NF-κB inibito la maturazione barriera epiteliale barriera con conseguente aumento della traslocazione batterica durante l’associazione E. coli con l’epitelio immaturo.
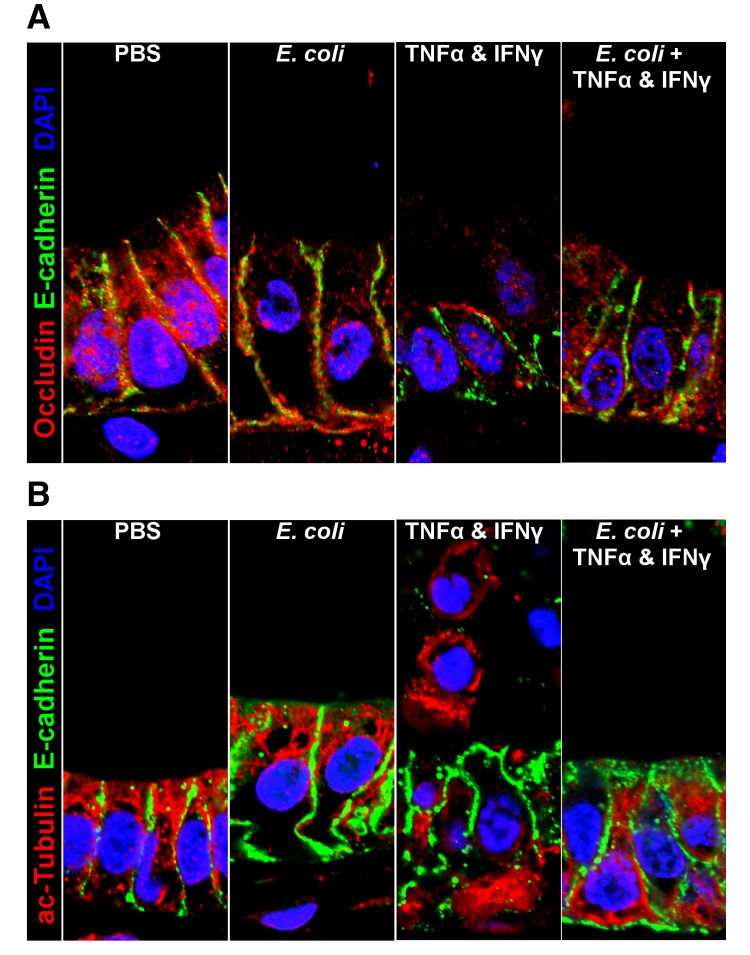
Figura 8-figure supplemento 1.Figura 8— figura 1. La colonizzazione batterica promuove la resilienza della barriera epiteliale tramite NF-κB.Representative micrografie confocali di IO trattati come indicato.(A) Heatmap dei dati RNA-seq che indicano l’espressione relativa dei geni associati alla giunzione Adherens o all’assemblaggio di giunzione cellula-cellula basato sull’annotazione nel database REACTOME. I risultati rappresentano la media di N = 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO raggruppati e trattati in modo identico.(B) Intensità di fluoresscenza relativa nel tempo in HIO microiniettato con 4 kDa FITC-dextran e ripreso a intervalli di 10 minuti. Gli IO sono stati pretrattati mediante microiniezione con104 CFU E. coli in PBS o PBS da solo e sono stati messi in coltura per 24 ore prima del trattamento con mezzi contenenti 10 μM SC-514 o PBS da solo e l’iniezione di 2 mg/ml di FITC-destrano (4 kDa) all’inizio dell’imaging. La linea rappresenta il miglior adattamento ai valori medi di intensità fluorescente in ogni condizione con la regione grigia che indica S.E. per la linea di adattamento. N= 7-9 HIO per gruppo.(C) Tasso di traslocazione batterica nel tempo negli HIO trattati come indicato nella legenda delle figure, come rilevato dalla raccolta giornaliera di mezzi HIO esterni e dall’arricchimento nel brodo di crescita batterica. N= 24 (E. coli +SC-514), N = 48 (E. coli), e N = 12 (PBS edE. coli + veicolo).(D) Intensità di fluorescenza relativa nel tempo in HIO microiniettato con FITC-dextran e ripreso a intervalli di 10 minuti. HIOs sono stati pretrattati per microiniezione con104 CFU E. coli in PBS o PBS solo e coltivato per 24 ore prima del trattamento con mezzi contenenti 500 ng/ml TNF-α e 500 ng/ml IFN-γ o PBS solo e l’iniezione di 2 mg/ml FITC-dextran (4 kDa) all’inizio di imaging. La linea rappresenta il miglior adattamento ai valori medi di intensità fluorescente in ogni condizione con la regione grigia che indica S.E. per la linea di adattamento. N= 8-9 HIO per gruppo.(E) Micrografie confocali rappresentative degli IO trattati come indicato in D. Pseudocolorizzazione con immunocolorazione fluorescente applicata come indicato nella legenda della figura. Ingrandimento ottico 60X con zoom digitale 2X. SC-514, inibitore delle piccole molecole di NF-κB; HK, inattivato a caldo; TNF, fattore di necrosi tumorale-α; IFN, interferone-γ.Fluorescente immunocolorazione pseudocolorante applicata come indicato nelle leggende figura. Ingrandimento ottico 60X con zoom digitale 2X.

Figura 8-figure supplemento 1.Figura 8. Colonizzazione batterica promuove la resilienza della barriera epiteliale tramite NF-κB.micrografie confocali rappresentative di HIO trattati come indicato.(A) Heatmap dei dati RNA-seq che indicano l’espressione relativa dei geni associati alla giunzione Adherens o all’assemblaggio di giunzione cellula-cellula basato sull’annotazione nel database REACTOME. I risultati rappresentano la media di N = 4-5 repliche biologiche per condizione di trattamento, con ogni replicato costituito da 5-6 HIO raggruppati e trattati in modo identico.(B) Intensità di fluoresscenza relativa nel tempo in HIO microiniettato con 4 kDa FITC-dextran e ripreso a intervalli di 10 minuti. Gli IO sono stati pretrattati mediante microiniezione con104 CFU E. coli in PBS o PBS da solo e sono stati messi in coltura per 24 ore prima del trattamento con mezzi contenenti 10 μM SC-514 o PBS da solo e l’iniezione di 2 mg/ml di FITC-destrano (4 kDa) all’inizio dell’imaging. La linea rappresenta il miglior adattamento ai valori medi di intensità fluorescente in ogni condizione con la regione grigia che indica S.E. per la linea di adattamento. N= 7-9 HIO per gruppo.(C) Tasso di traslocazione batterica nel tempo negli HIO trattati come indicato nella legenda delle figure, come rilevato dalla raccolta giornaliera di mezzi HIO esterni e dall’arricchimento nel brodo di crescita batterica. N= 24 (E. coli +SC-514), N = 48 (E. coli), e N = 12 (PBS edE. coli + veicolo).(D) Intensità di fluorescenza relativa nel tempo in HIO microiniettato con FITC-dextran e ripreso a intervalli di 10 minuti. HIOs sono stati pretrattati per microiniezione con104 CFU E. coli in PBS o PBS solo e coltivato per 24 ore prima del trattamento con mezzi contenenti 500 ng/ml TNF-α e 500 ng/ml IFN-γ o PBS solo e l’iniezione di 2 mg/ml FITC-dextran (4 kDa) all’inizio di imaging. La linea rappresenta il miglior adattamento ai valori medi di intensità fluorescente in ogni condizione con la regione grigia che indica S.E. per la linea di adattamento. N= 8-9 HIO per gruppo.(E) Micrografie confocali rappresentative degli IO trattati come indicato in D. Pseudocolorizzazione con immunocolorazione fluorescente applicata come indicato nella legenda della figura. Ingrandimento ottico 60X con zoom digitale 2X. SC-514, inibitore delle piccole molecole di NF-κB; HK, inattivato a caldo; TNF, fattore di necrosi tumorale-α; IFN, interferone-γ.Fluorescente immunocolorazione pseudocolorante applicata come indicato nelle leggende figura. Ingrandimento ottico 60X con zoom digitale 2X.

Figura 8-figure supplemento 1.Rappresentante micrografie confocali di HIO trattati come indicato.2. Immunostaining fluorescente pseudo-colorazione applicata come indicato nelle leggende figura. 60X ingrandimento ottico con zoom digitale 2X.
La colonizzazione batterica promuove la resilienza della barriera epiteliale durante la sfida citochina
Infine, abbiamo saggiato la funzione di barriera epiteliale in circostanze che ricapitolano l’infiammazione fisiologica. TNFα e IFNγ sono citochine chiave che mediano l’attività innata e adattativa delle cellule immunitarie nell’intestino (Turner,2009) durante l’infezione batterica (Rhee et al.,2005; Emami et al., 2012) e in enterocolite necrotizzante (Tan et al.,1993; Ford et al., 1996, 1997; Halpern et al., 2003; Upperman et al., 2005). La combinazione di TNFα e IFNγ è stata precedentemente dimostrata per indurre la permeabilità di barriera in modo dose-dipendente nelle colture epiteliali Transwell (Wang et al., 2005; Wang et al., 2006). Così, gli HIO sono stati microiniettati con PBS o E. coli vivo e coltivato per 24 ore e sono stati successivamente microiniettato con FITC-destrano e trattati con PBS o un cocktail di TNFα e IFNγ aggiunto ai mezzi esterni per esporre l’epitelio basolaterale (Figura 8D). Perdita di FITC-destrano fluorescenza è stata osservata utilizzando live-imaging e ha indicato che il trattamento con TNFα e IFNγ da solo ha portato ad una rapida e sostenuta diminuzione della fluorescenza luminale rispetto al PBS o E. coli iniettato HIOs (p = 5 × 10-4, Figura 8D). Tuttavia, HIOs associati con E. coli prima dell’aggiunta del TNFα e IFNγ cocktail mantenuto segnale fluorescente significativamente più rispetto al trattamento con TNFα e IFNγ solo (p = 0,042, Figura 8D). Abbiamo esaminato l’espressione e la distribuzione della proteina di giunzione stretta ZO-1, e la proteina basale-laterale E-caderina (ECAD) nelle sezioni istologiche prese da PBS e E. coli-associati HIOs sottoposti a trattamento TNFα e IFNγ (Figura 8E). Rispetto ai controlli, lo strato epiteliale è altamente disorganizzato in HIO trattati con TNFα e IFNγ, con colorazione citoplasmatica ZO-1 e ECAD disorganizzato. Al contrario, HIOs associati con E. coli prima del trattamento TNFα e IFNγ mantenere e organizzato epitelio colonnare epitelio con robusta apicale ZO-1 e correttamente localizzato colorazione ECAD (Figura 8E). Allo stesso modo, la corretta localizzazione di ulteriori marcatori aggiuntivi di barriera epiteliale integrità occludina (OCLN) e acetilato-tubulina sono conservati in HIO associati con E. coli durante il trattamento TNFα e IFNγ rispetto ai HIO trattati con TNFα e IFNγ solo (Figura 8-figuresupplemento 1).Questi risultati suggeriscono che la colonizzazione dell’epitelio immaturo con E. coli risultati in un epitelio che è più robusto per sfidare da citochine infiammatorie potenzialmente dannose.
Discussione
Il lavoro qui presentato dimostra che gli IO rappresentano un robusto sistema modello per studiare le interazioni iniziali tra l’epitelio gastrointestinale e i microbi colonizzatori che si verificano nell’immediato periodo postnatale. I microrganismi introdotti nel tratto digestivo alla nascita stabiliscono una relazione intima e mutualistica con l’ospite nel tempo(Costello et al., 2012; Palmer et al., 2007; Koenig et al., 2011; Bäckhed et al., 2015; Wopereis et al., 2014). Tuttavia, l’espansione delle popolazioni batteriche nell’intestino rappresenta anche una sfida importante per l’omeostasi intestinale attraverso l’esposizione a MAMP potenzialmente infiammatori(Tanner et al., 2015; Renz et al., 2011), il consumo di ossigeno dei tessuti(Glover et al.,2016; Espey, 2013; Albenberg et al., 2014), la digestione della barriera mucosa(Marcobal et al., 2013; Desai et al., 2016) e la competizione per i substrati metabolici(Rivera-Chávez et al., 2016; Kaiko et al., 2016 ). L’epitelio intestinale maturo funge da barriera cruciale per i microbi che abitano le superfici del lume e delle mucose(Artis, 2008; Turner, 2009; Desai et al., 2016; Kelly et al., 2015; Cornick et al., 2015; Peterson e Artis, 2014; Hackam et al., 2013; Turner, 2009). La funzione specifica dell’epitelio nell’adattarsi alla colonizzazione microbica iniziale, indipendente dal sistema immunitario innato e adattivo, rimane poco chiara a causa della mancanza di sistemi modello appropriati che ricapitolino il mutualismo microbico ospite. Chiarire il ruolo dell’epitelio nella colonizzazione del tratto digestivo da parte dei microrganismi è essenziale per comprendere la base molecolare del mutualismo microbico ospite stabile nell’intestino maturo.
Per esaminare l’istituzione del mutualismo microbico ospite, abbiamo scelto di esaminare l’interazione tra l’epitelio immaturo degli HIO e un ceppo non patogeno di E. coli. Le enterobatteriacee, tra cui l ‘E. coli, sono abbondanti nell’intestino neonato(Palmer et al., 2007; Koenig et al., 2011; Bäckhed et al., 2015; DIABIMMUNE Study Group et al., 2016). Diverse indagini su larga scala sulla composizione microbica hanno dimostrato che l’E. coli è tra gli organismi più diffusi e abbondanti nei campioni di feci dei neonati(Bäckhed et al., 2015; Koenig et al., 2011) e nel meconio (Gosalbes et al.,2013). I ceppi non patogeni di E. coli possono rappresentare organismi modello ideali per esaminare l’impatto della colonizzazione batterica dell’epitelio immaturo a causa della loro prevalenza nella popolazione neonatale e della loro rilevanza per la colonizzazione naturale, la caratterizzazione estesa e la facilità di manipolazione in laboratorio . Microiniezione di E. coli non patogeni nel lume di HIOs ha portato a stabili, co-colture a lungo termine(Figura 1). E. coli cresce rapidamente all’interno del lume HIO(Figura 1), raggiungendo densità approssimativamente paragonabili alle popolazioni presenti nell’intestino tenue umano(Donaldson et al., 2016) entro 24 ore. Inoltre, l’HIO è in grado di sostenere questa popolazione microbica interna per diversi giorni mantenendo l’integrità della barriera epiteliale(Figura 1). Questa osservazione è implicita nella conclusione che l’epitelio immaturo, insieme ad uno strato mesenchimale poco strutturato, è intrinsecamente in grado di adattarsi alle sfide imposte dalla colonizzazione con batteri intestinali non patogeni.
Per esaminare più da vicino questi adattamenti epiteliali della colonizzazione microbica, abbiamo eseguito un’analisi trascrizionale di questa risposta. Gli IO colonizzati da E. coli mostrano una diffusa attivazione trascrizionale di vie di riconoscimento batterico innate, comprese le cascate di segnalazione TLR e i mediatori a valle come NF-κB (Figura 2). La composizione cellulare dell’epitelio HIO è raffinato dopo la colonizzazione di E. col i, con un rapido ma transitorio aumento della proliferazione epiteliale che precede una riduzione generale del numero di cellule progenitrici epiteliali immature e l’emergere di enterocyte maturi che esprimono enzimi digestivi bordo pennello bordo digestivo(Figura 3). Insieme, questi risultati suggeriscono che gli stimoli batterici esercitano un’ampia influenza sulla composizione molecolare e cellulare dell’epitelio immaturo.
Stimoli indiretti derivanti dall’attività microbica possono anche modellare la funzione epiteliale(Buffie e Pamer, 2013), e il trascrittoma di E. coli colonizzato HIO riflette una risposta cellulare alla ridotta disponibilità di ossigeno(Figura 2). La riduzione della concentrazione di O2 luminale si verifica nell’intestino neonatale(Gruette et al., 1965; Fisher et al., 2013; Zheng et al., 2015), forse come risultato del consumo di O2 disciolto dai batteri anaerobi anaerobi anaerobici e facoltativi che predominano nel microbioma intestinale nei primi anni di vita(Espey, 2013; Fanaro et al., 2003; Favier et al., 2002; Palmer et al., 2007), e l’epitelio intestinale maturo è ipossico rispetto alla lamina propria sottostante a causa della stretta vicinanza al contenuto anaerobico luminoso(Glover et al., 2016; Kelly et al., 2015; Zheng et al., 2015). Abbiamo misurato il contenuto di ossigeno luminale e l’ipossia epiteliale in HIO microiniettato con E. coli vivo, trovando che la concentrazione di ossigeno luminale è ridotta più di 10 volte rispetto ai media circostanti. Questo stato di ipossia relativa si estende nell’epitelio stesso ed è correlato con l’aumento della densità microbica(Figura 4). Così, anche se gli IO non hanno la rete di capillari che svolgono un ruolo essenziale nella fornitura di ossigeno dei tessuti nell’intestino, gli IO di E. coli-colonizzati ricapitolano in vitro il gradiente di ossigeno presente nell’interfaccia epiteliale.
La colonizzazione dell’HIO da parte di E. coli comprende quindi due ampi stimoli: l’esposizione immediata a segnali mediati da contatto come i MAMP, e l’insorgenza di ossigeno luminale limitante e ipossia epiteliale. Sebbene il potenziale significato dell’esposizione a prodotti microbici nel contesto dell’ipossia tissutale sia ampiamente riconosciuto nell’ambito dell’enterocolite necrotizzante(Tanner et al., 2015; Afrazi et al., 2014; Hackam et al., 2013; Neu e Walker, 2011; Upperman et al., 2005; Nanthakumar et al., 2011), questo paradigma di segnalazione dei due fattori non è stato ben studiato come componente della normale colonizzazione e sviluppo intestinale. Utilizzando il sistema di modello HIO, è stato possibile progettare esperimenti che esaminano separatamente l’impatto relativo dei segnali microbici mediati per contatto da segnali ipossici associati a microbici(Figura 5 e Figura 5-figure supplement 2). Questo approccio rivela che la risposta trascrizionale completa generata dall’HIO dopo la colonizzazione di E. coli è il prodotto sia di segnali dipendenti dal contatto che di segnali dipendenti dall’ipossia, con E. coli o ipossia inattivati dal calore che ricapitolano da soli sottoinsiemi distinti dei cambiamenti nell’espressione genica osservati negli HIO colonizzati con E. col i vivi(Figura 5). Studi futuri potranno esaminare il ruolo di ulteriori stimoli microbici associati all’ipossia indipendenti dall’ipossia, come i prodotti metabolici(Kaiko et al., 2016) e i MAMP associati alla viabilità(Sander et al., 2011), nel mediare la risposta epiteliale alla colonizzazione batterica iniziale.
La segnalazione di NF-κB è stata implicata nella risposta a valle sia ai segnali microbici mediati dal contatto (Zhange Ghosh, 2001; Xiao e Ghosh, 2005; Kawai e Akira, 2007) sia all’ipossia dei tessuti (Koong et al.,1994; Rius et al., 2008; Arias-Loste et al., 2015; Oliver et al., 2009; Zeitouni et al., 2016; Colgan et al., 2013; Grenz et al., 2012). L’inibizione farmacologica di NF-κB ha portato alla soppressione di entrambe le espressioni geniche microbiche associate al contatto e all’ipossia negli IO, inibendo sia le vie di difesa della barriera epiteliale mediata dal contatto che l’attivazione immunitaria associata all’ipossia (Figura 5). NF-κB sembra svolgere un ruolo chiave nell’integrazione degli stimoli complessi derivanti dall’esposizione a prodotti microbici e dall’insorgenza di ipossia localizzata nell’epitelio intestinale immaturo durante la colonizzazione batterica.
La maturazione molecolare e cellulare dell’intestino che si verifica durante l’infanzia si traduce in ultima analisi in una maggiore capacità funzionale(Lebenthal e Lebenthal, 1999; Sanderson e Walker, 2000; Neu, 2007). La colonizzazione batterica è associata a una maggiore funzione di barriera epiteliale negli animali gnotobiotici, compresi i cambiamenti nella produzione di peptidi antimicrobici e muco(Vaishnava et al., 2008; Cash et al., 2006; Goto et al., 2014; García-Lafuente et al., 2001; Malago , 2015; Ménard et al., 2008). Le difese prodotte nell’epitelio intestinale sono mediatori critici della densità e della composizione delle popolazioni microbiche nell’intestino e proteggono l’epitelio dall’invasione microbica(Kisich et al., 2001; Ostaff et al., 2013; Cullen et al., 2015; Salzman et al., 2003; Salzman et al., 2010). La produzione di BD-2 è drammaticamente aumentata negli IO subito dopo la colonizzazione dell’E. coli(Figura 2, Figura 5-figure supplement 2 e Figura 6), raggiungendo concentrazioni sufficienti a limitare la crescita eccessiva di E. coli (Figura 6 e Figura 6-figure supplement 2) senza precludere completamente la colonizzazione batterica potenzialmente benefica (Figura 1). Le mucine secrete e le mucine associate alla superficie cellulare formano una barriera fisica ai microbi nell’intestino, agiscono come serbatoi locali di peptidi antimicrobici e servono come substrati per la crescita di microrganismi benefici(Desai et al., 2016; Johansson e Hansson, 2016; Cornick et al., 2015; Hansson, 2012; Li et al., 2015; Dupont et al., 2014; Bergstrom e Xia, 2013). L’epitelio HIO immaturo produce un robusto strato di muco costituito da oligosaccaridi neutri e acidi con modificazioni terminali dei carboidrati a seguito della colonizzazione con E. coli(Figura 7). È importante notare che l’ipossia da sola non provoca la produzione di muco, mentre l’introduzione di E. col i inattivata dal calore induce la secrezione di muco in corrispondenza dell’epitelio apicale(Figura 7), suggerendo che il contatto microbico è lo stimolo principale che provoca la secrezione di muco negli HIO.
La permeabilità della barriera epiteliale è un importante parametro della funzione intestinale che riflette il grado di selettività nel trasferimento di nutrienti attraverso lo strato epiteliale e l’esclusione di batteri e altri materiali potenzialmente dannosi(Bischoff et al., 2014). Gli aumenti della permeabilità della barriera epiteliale si verificano nell’ambito dell’infiammazione(Ahmad et al., 2017; Michielan e D’Incà, 2015) e delle malattie infettive (Shawkie McCole ,2017). Colonizzazione di HIOs con E. coli risulta in una maggiore trascrizione dei geni associati alla formazione della giunzione aderente e altre interazioni cellula-cellula nell’epitelio(Figura 8). Tuttavia, l’inibizione della segnalazione di NF-κB aumenta drasticamente sia la permeabilità della barriera epiteliale che il tasso di traslocazione batterica (Figura 8), suggerendo che la segnalazione di NF-κB è fondamentale per mantenere l’integrità della barriera epiteliale dopo la colonizzazione. L’espressione dei geni coinvolti nella formazione della giunzione cellulare e la produzione di difese antimicrobiche e muco sono dipendenti da NF-κB (Figure6-8, Figura 7-figure supplement 1, Tsutsumi-Ishii e Nagaoka, 2002; Ahn et al., 2005). L’incapacità di montare un’efficace risposta di difesa innata in presenza dell’inibizione di NF-κB comporta il fallimento della barriera epiteliale HIO e la perdita di stabilità della co-cultura (Figura 8). Questo risultato sottolinea il ruolo critico della segnalazione di NF-κB nella formazione di un mutualismo ospite-microbo stabile all’interfaccia epiteliale immaturo.
La produzione disordinata di citochine pro-infiammatorie contribuisce alla perdita di integrità della barriera epiteliale in NEC(Tanner et al., 2015; Hackam et al., 2013; Neu e Walker, 2011; Nanthakumar et al., 2011; Halpern et al., 2003; Ford et al., 1997, 1996; Tan et al., 1993); questo è riassunto in HIO, in quanto l’esposizione a citochine pro-infiammatorie comporta la rapida perdita di integrità della barriera epiteliale e la dissoluzione delle giunzioni strette epiteliali(Figura 8). I probiotici possono promuovere l’integrità della barriera epiteliale in NEC(Robinson, 2014; Alfaleh et al., 2011; Underwood et al., 2014; Khailova et al., 2009) e gli IO colonizzati da E. coli mostrano una maggiore resilienza della barriera epiteliale(Figura 8). La maturazione funzionale derivante dalla colonizzazione dell’epitelio intestinale immaturo può quindi svolgere un ruolo essenziale nel promuovere la risoluzione dell’infiammazione fisiologica.
Mentre sono stati fatti grandi progressi nella caratterizzazione della composizione del microbiota intestinale in salute e malattia(Shreiner et al., 2015; Costello et al., 2012), questo approccio ha una limitata capacità di discernere i contributi dei singoli batteri alla creazione di simbiosi ospite-microbo. Il nostro lavoro stabilisce un approccio che ricapitola il mutualismo microbico ospite nell’intestino umano immaturo in un sistema di modelli in vitro sperimentalmente tracciabili. L’applicazione di questo approccio può facilitare lo sviluppo di modelli meccanicistici di interazioni microbiche ospite-microbiche nei tessuti umani in salute e nelle malattie. Per esempio, uno dei principali limiti nella nostra comprensione del NEC è stata la mancanza di un sistema modello appropriato per studiare la colonizzazione dell’intestino immaturo(Neu e Walker ,2011; Balimane e Chong, 2005; Tanner et al., 2015; Nguyen et al ., 2015). I nostri risultati suggeriscono che la colonizzazione dell’HIO con un batterio intestinale non patogeno porta alla maturazione funzionale della barriera epiteliale. I lavori futuri che esaminano gli effetti degli organismi associati all’intestino prematuro(Morrow et al., 2013; Greenwood et al., 2014; Ward et al., 2016) sulla maturazione molecolare, cellulare e funzionale dell’epitelio immaturo possono essere utili per chiarire i meccanismi di patogenesi della malattia associata a microbiota nell’intestino immaturo.
Materiali e metodi
Cultura HIO
Human ES cell line H9 (NIH registry #0062, RRID:CVCL_9773) è stato ottenuto dal WiCell Research Institute. Le cellule H9 sono state autenticate utilizzando Short Tandem Repeat (STR) DNA profiling(Matsuo et al., 1999) presso l’Università del Michigan DNA Sequencing Core ed esibivano un profilo STR identico alle caratteristiche STR pubblicate da(Josephson et al., 2006). La linea cellulare H9 era negativa per la contaminazione da micoplasma. Le cellule staminali sono state mantenute su Matrigel (BD Biosciences, San Jose, CA) in mezzo mTeSR1 (STEMCELL Technologies, Vancouver, Canada). Le cellule staminali HESC sono state passate e differenziate in tessuto organolettico intestinale umano come descritto in precedenza(Spence et al., 2011; McCracken et al., 2011). Gli HIO sono stati mantenuti in mezzi contenenti EGF, Noggin e R-spondin (mezzi ENR, vedi[McCracken et al., 2011]) in 50 μl Matrigel (8 mg/ml) senza antibiotici prima degli esperimenti di microiniezione. Per gli esperimenti di coltura ipossica, gli HIO sono stati trasferiti in una camera di incubazione modulare idratata e sigillata (MIC-101, Billups-Rothenburg, Inc. Del Mar CA) riempita con 1% di O2, 5% di CO2, e il bilancio N2 e mantenuta a 37 per 24 ore.
Trapianto HIO e coltura di enteroidi derivati da tessuti
Trapianti HIO: Questo studio è stato eseguito in stretta conformità con le raccomandazioni della Guida per la cura e l’uso degli animali da laboratorio degli Istituti Nazionali di Sanità. Tutti gli esperimenti sugli animali sono stati approvati dall’Università del Michigan Institutional Animal Care and Use Committee (IACUC; protocollo # PRO00006609). I trapianti HIO nella capsula renale sono stati eseguiti come descritto in precedenza(Finkbeiner et al., 2015; Dye et al., 2016) In breve, i topi sono stati anestetizzati con il 2% di isofluorano. Il fianco sinistro è stato sterilizzato con cloroesidina e alcool isopropilico, ed è stata praticata un’incisione per esporre il rene. HIOs sono stati inseriti manualmente in una tasca sottocapsulare del rene di maschio di 7-10 settimane NOD-SCID IL2Rgnull (NSG) topi usando il forcipe. Un lavaggio intraperitoneale di Zosyn (100 mg / kg; Pfizer Inc.) è stato somministrato prima della chiusura in due strati. I topi sono stati sacrificati e trapianto recuperato dopo 10 settimane. Tessuto umano: Normale, de-identificato tessuto intestinale fetale umano è stato ottenuto dal Laboratorio di Biologia dello Sviluppo dell’Università di Washington. Normale, de-identificato tessuto intestinale umano adulto intestinale è stato ottenuto da donatori di organi deceduti attraverso il Dono della Vita, Michigan. Tutti i tessuti umani utilizzati in questo lavoro sono stati ottenuti da donatori non viventi, sono stati de-identificati e sono stati condotti con l’approvazione dell’Università del Michigan IRB (protocollo # HUM00093465 e HUM00105750). L’isolamento e la coltura dell’epitelio HIO, l’epitelio HIO trapiantato, l’epitelio duodenale fetale e quello umano adulto sono stati effettuati come descritto in precedenza(Finkbeiner et al., 2015), e sono stati coltivati in una gocciolina di Matrigel utilizzando un mezzo condizionato L-WRN per stimolare la crescita epiteliale, come descritto in precedenza(Miyoshi et al., 2012; Miyoshi e Stappenbeck, 2013).
Coltura batterica
Escherichia coli ceppo ECOR2 (ATCC 35321) è stato coltivato in brodo di Luria (LB) media o piastre di agar LB 1,5% a 37 in condizioni di ossigeno atmosferico. Soluzioni a base di glicerolo sono disponibili su richiesta. Il genoma assemblato e annotato per l’isolato di Escherichia coli ceppo ECOR2 utilizzato in questi studi è disponibile suhttps://www.patricbrc.org/view/Genome/562.18521.E. coli ceppo K-12 MG1655 (CGSC #6300) è stato ottenuto dal Coli Genetic Stock Center presso l’Università di Yale(http://cgsc2.biology.yale.edu/) ed è stato utilizzato solo negli esperimenti di attività in vitro BD-2. L’intero sequenziamento del genoma del ceppo di E. coli ECOR2 è stato eseguito dal Laboratorio di Iniziativa Microbioma Host dell’Università del Michigan utilizzando la piattaforma Illumina MiSeq.
Microiniezione
Le microiniezioni sono state eseguite utilizzando un protocollo modificato da Leslie et al., 2015. In breve, gli HIO sono stati iniettati utilizzando capillari in vetro a parete sottile (TW100F-4, World Precision Instruments, Sarasota, FL) modellati con un estrattore per micropipette P-30 (Sutter Instruments, Novato, CA). I microcapillari tirati sono stati montati su un supporto per micropipette Xenoworks con tubo analogico (BR-MH2 e BR-AT, Sutter Instruments) collegato a una siringa di vetro da 10 ml riempita con olio minerale sterile (Fisher Scientific, Hampton, NH). Il controllo fine della micropippetta è stato ottenuto utilizzando un micromanipolatore (Narishge International Inc., East Meadow, NY) e la microiniezione è stata completata con un ingrandimento 1-2X su un cannocchiale di dissezione stereo SX61 (Olympus, Tokyo, Giappone). Gli HIO sospesi a Matrigel (Corning Inc., Corning, NY) sono stati iniettati con circa 1 μl di soluzione. Un protocollo di microiniezione HIO dettagliato e aggiornato è disponibile su Hill, 2017b; una copia è archiviata suhttps://github.com/elifesciences-publications/HIO_microinjection). Negli esperimenti di microiniezione batterica, i terreni di coltura HIO sono stati rimossi immediatamente dopo la microiniezione e le colture sono state risciacquate con PBS e trattate con terreni ENR contenenti penicillina e streptomicina per rimuovere i batteri introdotti nei terreni di coltura durante il processo di microiniezione. Dopo 1 ora e 37 minuti, gli HIO sono stati nuovamente lavati in PBS e i terreni di coltura sono stati sostituiti con nuovi mezzi ENR privi di antibiotici.
Misurazione dell’ossigeno luminale
Il contenuto di ossigeno luminoso è stato misurato in HIOs utilizzando un microsensore impiantabile rivestito otticamente con una punta conica a <50 μm (IMP-PSt1, PreSens Precision Sensing GmbH) collegato ad un micro ossimetro a fibre ottiche (Microx TX3, PreSens Precision Sensing GmbH, Regensburg, Germania). La sonda di ossigeno è stata calibrata secondo le istruzioni del produttore e le misurazioni dei supporti esterni e il contenuto di ossigeno HIO luminale sono stati raccolti montando il microsensore su un micromanipolatore (Narishge International Inc., East Meadow, NY) e guidando la punta del sensore in posizione usando l’ingrandimento 1-2X su un cannocchiale di dissezione stereo (Olympus, Tokyo, Giappone). Tutte le letture della concentrazione di ossigeno sono state analizzate usando il software PreSens Oxygen Calculator (TX3v531, PreSens Precision Sensing GmbH, Regensburg, Germania). Per la misurazione dell’ipossia citoplasmatica relativa, le colture HIO sono state trattate con 100 μM di pimonidazolo HCl (Hypoxyprobe, Inc., Burlington, MA) aggiunto ai terreni di coltura esterni e incubate al 37% e 5% di CO2 per 2 ore prima della fissazione in parafomaldeide al 4%. I coniugati di pimonidazolo sono stati colorati in sezioni di tessuto utilizzando l’anticorpo monoclonale IgG Ipoxyprobe-1 di topo (Hypoxyprobe, Inc., Burlington, MA, RRID:AB_2335667) con anticorpo secondario appropriato (vedi tabella delle diluizioni anticorpali).
Immunoistochimica
L’immunocolorazione è stata effettuata come descritto in precedenza(Finkbeiner et al., 2015). Informazioni sugli anticorpi e sulle diluizioni si trovano nel file supplementare 2. Tutte le immagini sono state scattate con un microscopio confocale Nikon A1 o con un microscopio epifluorescente Olympus IX71. 3. Il tampone di blocco CarboFree (SP-5040; Vector Laboratories, Inc. Burlingame, CA) è stato sostituito con il siero d’asino diluito in PBS nella colorazione per le mucine e i moieties dei carboidrati. Trattamento EdU e l’etichettatura fluorescente EdU utilizzando la chimica Click-iT è stato applicato secondo le istruzioni del produttore (# C10339 Thermo Fisher, Waltham, MA). Il file supplementare 2 contiene una tabella di tutti gli anticorpi primari e secondari, le condizioni di blocco e le informazioni per ordinare il prodotto.
Inibizione NF-κB
L’inibitore NF-κB SC-514 (Kishoreet al., 2003; Litvak et al., 2009) (Tocris Cookson, Bristol, UK) è stato risospeso in DMSO ad una concentrazione di 25 mM. Gli HIO sono stati trattati con SC-514 in sospensione in DMSO aggiunto ai mezzi di coltura esterni ENR ad una concentrazione finale di 1 μM. L’efficacia di SC-514 è stata verificata da Western blot di lisati da HIO iniettati con PBS o E. coli vivo o iniettati con E. coli vivo in presenza di 1 μM SC-514 aggiunto ai mezzi esterni. Gli IO sono stati raccolti dopo 24 ore in tampone di lisi composto da 300 mM NaCl, 50 mM Tris base, 1 mM EDTA, 10% glicerolo, 0,5% NP-40, e 1X Halt Phosphatase Inhibitor Cocktail (Pierce Biotechnology, Rockford, IL). I lisati sono stati separati su un gel di poliacrilamide Bis-Tris al 10% in condizioni riducenti (Invitrogen, Carlsbad, CA) e trasferiti al PVDF utilizzando un apparato di trasferimento a umido (Bio-Rad Laboratories, Hercules, CA) durante la notte alle 4. La membrana del PVDF è stata bloccata nel buffer di blocco Odyssey TBS (LI-COR Biosciences, Lincoln, NE). La membrana è stata immersa in tampone di blocco contenente anticorpi monoclonali primari di coniglio contro NF-κB p65 fosforilato (1:200, Cell Signaling Technology #3033S) o totale NF-κB p65 (1:400, Cell Signaling Technology #8242S) e incubata a temperatura ambiente per 2 ore. Tutti i lavaggi sono stati condotti in soluzione salina tamponata con Tris con 1% Tween-20 (TBST). La capra secondaria anti-coniglio IgG IRDye IgG 800CW è stato diluito 1:15.000 in TBST ed esposto alla membrana lavata per 1 ora a temperatura ambiente. Dopo ulteriori lavaggi, la membrana in PVDF è stata ripresa con un Odyssey Scanner (LI-COR Biosciences, Lincoln, NE).
Saggio di traslocazione batterica
L’incidenza della traslocazione batterica è stata determinata in HIO placcati singolarmente in singoli pozzetti di piastre a 24 pozzetti e microiniettati con E. coli. I terreni di coltura esterni sono stati raccolti e sostituiti quotidianamente. I terreni di coltura raccolti sono stati diluiti 1:10 in brodo LB in 96 piastre a 96 pozzetti e sono stati messi in coltura a 37 durante la notte. La densità ottica (600 nm) è stata misurata nelle colture di brodo LB a 96 pozzetti utilizzando un lettore di micropiastre VersaMax (Molecular Devices, LLC, Sunnyvale, CA). OD600> sterile brodo di brodo LB linea di base è stata considerata una coltura positiva.
Permeabilità FITC-dextran
Per i saggi di permeabilità epiteliale, gli HIO sono stati microiniettati con 4 kDa FITC-dextran in sospensione in PBS ad una concentrazione di 2 mg/ml come descritto in precedenza(Leslie et al., 2015) utilizzando il sistema di microiniezione descritto in precedenza. Le immagini sono state raccolte a intervalli di 10 minuti a 4X ingrandimento su un microscopio epifluorescente Olympus IX71 utilizzando un sistema di imaging a cellule vive Deltavision RT con il software di imaging Applied Precision softWoRx (GE Healthcare Bio-Sciences, Marlborough, MA). Le colture sono state mantenute al 37% e 5% diCO2 in tutto il corso del tempo di imaging. Per gli esperimenti che coinvolgono il trattamento delle citochine, ricombinante TNF-α (#210-TA-010, R e D Systems) e INF-γ (#AF-300-02, Peprotech) sono stati aggiunti ai mezzi di coltura esterna ad una concentrazione di 500 ng / ml all’inizio dell’esperimento. Un dettagliato e aggiornato protocollo di microiniezione HIO e di imaging dal vivo è disponibile su(Hill, 2017b).
Saggio di attività antimicrobica in vitro
BD-2 umano ricombinante (Abcam, Cambridge, MA) è stato ricostituito in brodo LB sterile e diluito a 0,1-1 μg/ml. Le colture di E. coli sono state diluite 1:1000 in LB sterile contenente 0-1 μg/ml di BD-2 e trasferite in una micropiastra a 96 pozzetti. Un lettore di micropiastre VersaMax (Molecular Devices, LLC, Sunnyvale, CA) è stato utilizzato per misurare OD600 a intervalli di 10 minuti in micropiastre mantenute a 37°C con scuotimento regolare su un arco di tempo di 18 ore. Per i saggi antimicrobici in fase stazionaria, colture notturne di E. coli str. ECOR2 sono state diluite in PBS contenente 1 μg / ml BD-2 o BD-2 inattivato a caldo (riscaldato a 120 per > 60 min) e posto in un incubatore batterico 37 per 5 ore. Le colture sono state poi diffuse su piastre di agar LB e coltivate durante la notte. Il numero di CFU è stato contato manualmente.
Saggi ELISA
Le concentrazioni di citochina secreta, peptide antimicrobico e fattore di crescita sono state determinate mediante ELISA (Duosets, R e D Systems, Minneapolis, MN) utilizzando le procedure raccomandate dal produttore presso il Centro di monitoraggio immunologico dell’Università del Michigan Cancer Center.
Sequenziamento e analisi dell’RNA
L’RNA è stato isolato utilizzando il kit di isolamento dell’RNA mirVana e seguendo il protocollo di isolamento ‘RNA totale’ (Thermo-Fisher Scientific, Waltham MA). RNA preparazione biblioteca RNA e RNA-sequenziamento (single-end, 50 bp lunghezza di lettura) sono state eseguite dalla University of Michigan DNA Sequenziamento Core utilizzando la Illumina Hi-Seq 2500 piattaforma. Tutte le sequenze sono state depositate nel database EMBL-EBI ArrayExpress (RRID:SCR_004473) utilizzando Annotare 2.0 e sono catalogate con il numero di adesione E-MTAB-5801. L’analisi quantitativa delle trascrizioni è stata condotta utilizzando la versione stabile a 64 bit di Debian Linux Linux 7.10 (‘Wheezy’). Lo pseudoallineamento dei dati RNA-seq è stato calcolato usando kallisto v0.43.0(Bray et al., 2016) e l’espressione differenziale delle sequenze pseudoallineate è stata calcolata usando il pacchetto R DEseq2(Love et al., 2014) (RRID:SCR_000154).
Analisi statistica
Se non diversamente indicato nelle leggende delle figure, le differenze tra i gruppi sperimentali o le condizioni sono state valutate utilizzando il t-test di uno studente non accoppiato. Un p-valore <0,05 è stato considerato rappresentare un risultato statisticamente significativo. Tutte le analisi statistiche sono state condotte utilizzando la versione R 3.4.1 (2017-06-30)(Core Team, 2017) e i grafici sono stati generati utilizzando il pacchetto R ggplot2(Wickham, 2009) con il pacchetto di espansione ggstance(Henry et al., 2016). Il test multiplo FDR regolato per il test è stato calcolato utilizzando l’implementazione DESeq2 del test di Wald(Love et al., 2014). I test di sovrarappresentazione del percorso genico e l’analisi dell’arricchimento del set di geni(Subramanian et al., 2005) sono stati implementati utilizzando i pacchetti R clusterProfiler(Yu et al., 2012) e ReactomePA(Yu and He, 2016). Le analisi condotte in R sono state raccolte utilizzando Emacs v25.2(Stallman, 1981) con Org-mode v8.3.5 e la carta è stata scritta in LaTeX utilizzando Emacs. Gli script di analisi completi sono disponibili nel repository Hill, 2017a GitHub (copia archiviata all’indirizzohttps://github.com/elifesciences-publications/Hill_HIO_Colonization_2017).
References
- Aagaard K, Ma J, Antony KM, Ganu R, Petrosino J, Versalovic J. The placenta harbors a unique microbiome. Science Translational Medicine. 2014; 6DOI | PubMed
- Abrahamsson TR, Jakobsson HE, Andersson AF, Björkstén B, Engstrand L, Jenmalm MC. Low gut microbiota diversity in early infancy precedes asthma at school age. Clinical & Experimental Allergy. 2014; 44:842-850. DOI | PubMed
- Abrams GD, Bauer H, Sprinz H. Influence of the normal flora on mucosal morphology and cellular renewal in the ileum. A comparison of germ-free and conventional mice. Laboratory Investigation; a Journal of Technical Methods and Pathology. 1963; 12:355-364. PubMed
- Afrazi A, Branca MF, Sodhi CP, Good M, Yamaguchi Y, Egan CE, Lu P, Jia H, Shaffiey S, Lin J, Ma C, Vincent G, Prindle T, Weyandt S, Neal MD, Ozolek JA, Wiersch J, Tschurtschenthaler M, Shiota C, Gittes GK, Billiar TR, Mollen K, Kaser A, Blumberg R, Hackam DJ. Toll-like receptor 4-mediated endoplasmic reticulum stress in intestinal crypts induces necrotizing enterocolitis. Journal of Biological Chemistry. 2014; 289:9584-9599. DOI | PubMed
- Ahmad R, Sorrell MF, Batra SK, Dhawan P, Singh AB. Gut permeability and mucosal inflammation: bad, good or context dependent. Mucosal Immunology. 2017; 10:307-317. DOI | PubMed
- Ahn DH, Crawley SC, Hokari R, Kato S, Yang SC, Li JD, Kim YS. TNF-alpha activates MUC2 transcription via NF-kappaB but inhibits via JNK activation. Cellular Physiology and Biochemistry. 2005; 15:029-040. DOI | PubMed
- Albenberg L, Esipova TV, Judge CP, Bittinger K, Chen J, Laughlin A, Grunberg S, Baldassano RN, Lewis JD, Li H, Thom SR, Bushman FD, Vinogradov SA, Wu GD. Correlation between intraluminal oxygen gradient and radial partitioning of intestinal microbiota. Gastroenterology. 2014; 147:1055-1063. DOI | PubMed
- Alfaleh K, Anabrees J, Bassler D, Al-Kharfi T. Probiotics for prevention of necrotizing enterocolitis in preterm infants. The Cochrane Database of Systematic Reviews. 2011; 3
- Arias-Loste MT, Fábrega E, López-Hoyos M, Crespo J. The Crosstalk between Hypoxia and Innate Immunity in the Development of Obesity-Related Nonalcoholic Fatty Liver Disease. BioMed Research International. 2015; 2015:1-8. DOI | PubMed
- Arrieta MC, Stiemsma LT, Dimitriu PA, Thorson L, Russell S, Yurist-Doutsch S, Kuzeljevic B, Gold MJ, Britton HM, Lefebvre DL, Subbarao P, Mandhane P, Becker A, McNagny KM, Sears MR, Kollmann T, Mohn WW, Turvey SE, Finlay BB, Investigators CS, Brett Finlay B, CHILD Study Investigators. Early infancy microbial and metabolic alterations affect risk of childhood asthma. Science Translational Medicine. 2015; 7DOI | PubMed
- Arteel GE, Thurman RG, Raleigh JA. Reductive metabolism of the hypoxia marker pimonidazole is regulated by oxygen tension independent of the pyridine nucleotide redox state. European Journal of Biochemistry. 1998; 253:743-750. DOI | PubMed
- Artis D. Epithelial-cell recognition of commensal bacteria and maintenance of immune homeostasis in the gut. Nature Reviews Immunology. 2008; 8:411-420. DOI | PubMed
- Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G. Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nature Genetics. 2000; 25:25-29. DOI | PubMed
- Aurora M, Spence JR. hPSC-derived lung and intestinal organoids as models of human fetal tissue. Developmental Biology. 2016; 420:230-238. DOI | PubMed
- Balimane PV, Chong S. Cell culture-based models for intestinal permeability: a critique. Drug Discovery Today. 2005; 10:335-343. DOI | PubMed
- Bastide P, Darido C, Pannequin J, Kist R, Robine S, Marty-Double C, Bibeau F, Scherer G, Joubert D, Hollande F, Blache P, Jay P. Sox9 regulates cell proliferation and is required for Paneth cell differentiation in the intestinal epithelium. The Journal of Cell Biology. 2007; 178:635-648. DOI | PubMed
- Bates JM, Mittge E, Kuhlman J, Baden KN, Cheesman SE, Guillemin K. Distinct signals from the microbiota promote different aspects of zebrafish gut differentiation. Developmental Biology. 2006; 297:374-386. DOI | PubMed
- Bergstrom KS, Xia L. Mucin-type O-glycans and their roles in intestinal homeostasis. Glycobiology. 2013; 23:1026-1037. DOI | PubMed
- Bevins CL, Salzman NH. Paneth cells, antimicrobial peptides and maintenance of intestinal homeostasis. Nature Reviews Microbiology. 2011; 9:356-368. DOI | PubMed
- Bischoff SC, Barbara G, Buurman W, Ockhuizen T, Schulzke JD, Serino M, Tilg H, Watson A, Wells JM. Intestinal permeability–a new target for disease prevention and therapy. BMC Gastroenterology. 2014; 14DOI | PubMed
- Borre YE, O’Keeffe GW, Clarke G, Stanton C, Dinan TG, Cryan JF. Microbiota and neurodevelopmental windows: implications for brain disorders. Trends in Molecular Medicine. 2014; 20:509-518. DOI | PubMed
- Bray NL, Pimentel H, Melsted P, Pachter L. Near-optimal probabilistic RNA-seq quantification. Nature Biotechnology. 2016; 34:525-527. DOI | PubMed
- Broderick NA, Buchon N, Lemaitre B. Microbiota-induced changes in drosophila melanogaster host gene expression and gut morphology. mBio. 2014; 5DOI | PubMed
- Brogden KA. Antimicrobial peptides: pore formers or metabolic inhibitors in bacteria?. Nature Reviews Microbiology. 2005; 3:238-250. DOI | PubMed
- Bry L, Falk PG, Midtvedt T, Gordon JI. A model of host-microbial interactions in an open mammalian ecosystem. Science. 1996; 273:1380-1383. DOI | PubMed
- Buffie CG, Pamer EG. Microbiota-mediated colonization resistance against intestinal pathogens. Nature Reviews Immunology. 2013; 13:790-801. DOI | PubMed
- Buisine MP, Devisme L, Savidge TC, Gespach C, Gosselin B, Porchet N, Aubert JP. Mucin gene expression in human embryonic and fetal intestine. Gut. 1998; 43:519-524. DOI | PubMed
- Buisine MP, Desreumaux P, Leteurtre E, Copin MC, Colombel JF, Porchet N, Aubert JP. Mucin gene expression in intestinal epithelial cells in Crohn’s disease. Gut. 2001; 49:544-551. DOI | PubMed
- Bäckhed F, Roswall J, Peng Y, Feng Q, Jia H, Kovatcheva-Datchary P, Li Y, Xia Y, Xie H, Zhong H, Khan MT, Zhang J, Li J, Xiao L, Al-Aama J, Zhang D, Lee YS, Kotowska D, Colding C, Tremaroli V, Yin Y, Bergman S, Xu X, Madsen L, Kristiansen K, Dahlgren J, Wang J. Dynamics and Stabilization of the Human Gut Microbiome during the First Year of Life. Cell Host & Microbe. 2015; 17DOI | PubMed
- Cash HL, Whitham CV, Behrendt CL, Hooper LV. Symbiotic bacteria direct expression of an intestinal bactericidal lectin. Science. 2006; 313:1126-1130. DOI | PubMed
- Cheesman SE, Neal JT, Mittge E, Seredick BM, Guillemin K. Epithelial cell proliferation in the developing zebrafish intestine is regulated by the Wnt pathway and microbial signaling via Myd88. PNAS. 2011; 108:4570-4577. DOI | PubMed
- Chin AM, Hill DR, Aurora M, Spence JR. Morphogenesis and maturation of the embryonic and postnatal intestine. Seminars in Cell & Developmental Biology. 2017; 66:81-93. DOI | PubMed
- Cho I, Yamanishi S, Cox L, Methé BA, Zavadil J, Li K, Gao Z, Mahana D, Raju K, Teitler I, Li H, Alekseyenko AV, Blaser MJ. Antibiotics in early life alter the murine colonic microbiome and adiposity. Nature. 2012; 488:621-626. DOI | PubMed
- Clarke G, O’Mahony SM, Dinan TG, Cryan JF. Priming for health: gut microbiota acquired in early life regulates physiology, brain and behaviour. Acta Paediatrica. 2014; 103:812-819. DOI | PubMed
- Colgan SP, Curtis VF, Campbell EL. The inflammatory tissue microenvironment in IBD. Inflammatory Bowel Diseases. 2013; 19:2238-2244. DOI | PubMed
- Core Team R. R Foundation for Statistical Computing: Vienna, Austria; 2017.
- Cornick S, Tawiah A, Chadee K. Roles and regulation of the mucus barrier in the gut. Tissue Barriers. 2015; 3DOI | PubMed
- Costello EK, Stagaman K, Dethlefsen L, Bohannan BJ, Relman DA. The application of ecological theory toward an understanding of the human microbiome. Science. 2012; 336:1255-1262. DOI | PubMed
- Croft D, Mundo AF, Haw R, Milacic M, Weiser J, Wu G, Caudy M, Garapati P, Gillespie M, Kamdar MR, Jassal B, Jupe S, Matthews L, May B, Palatnik S, Rothfels K, Shamovsky V, Song H, Williams M, Birney E, Hermjakob H, Stein L, D’Eustachio P. The Reactome pathway knowledgebase. Nucleic Acids Research. 2014; 42:D472-D477. DOI | PubMed
- Cullen TW, Schofield WB, Barry NA, Putnam EE, Rundell EA, Trent MS, Degnan PH, Booth CJ, Yu H, Goodman AL. Gut microbiota. antimicrobial peptide resistance mediates resilience of prominent gut commensals during inflammation. Science. 2015; 347:170-175. DOI | PubMed
- Dedhia PH, Bertaux-Skeirik N, Zavros Y, Spence JR. Organoid models of human gastrointestinal development and disease. Gastroenterology. 2016; 150:1098-1112. DOI | PubMed
- Desai MS, Seekatz AM, Koropatkin NM, Kamada N, Hickey CA, Wolter M, Pudlo NA, Kitamoto S, Terrapon N, Muller A, Young VB, Henrissat B, Wilmes P, Stappenbeck TS, Núñez G, Martens EC. A Dietary fiber-deprived gut microbiota degrades the colonic mucus barrier and enhances pathogen susceptibility. Cell. 2016; 167:1339-1353. DOI | PubMed
- Desbonnet L, Clarke G, Shanahan F, Dinan TG, Cryan JF. Microbiota is essential for social development in the mouse. Molecular Psychiatry. 2014; 19:146-148. DOI | PubMed
- Diaz Heijtz R, Wang S, Anuar F, Qian Y, Björkholm B, Samuelsson A, Hibberd ML, Forssberg H, Pettersson S. Normal gut microbiota modulates brain development and behavior. PNAS. 2011; 108:3047-3052. DOI | PubMed
- Donaldson GP, Lee SM, Mazmanian SK. Gut biogeography of the bacterial microbiota. Nature Reviews Microbiology. 2016; 14:20-32. DOI | PubMed
- Dupont A, Heinbockel L, Brandenburg K, Hornef MW. Antimicrobial peptides and the enteric mucus layer act in concert to protect the intestinal mucosa. Gut Microbes. 2014; 5:761-765. DOI | PubMed
- Dye BR, Dedhia PH, Miller AJ, Nagy MS, White ES, Shea LD, Spence JR. A bioengineered niche promotes in vivo engraftment and maturation of pluripotent stem cell derived human lung organoids. eLife. 2016; 5DOI | PubMed
- Emami CN, Mittal R, Wang L, Ford HR, Prasadarao NV. Role of neutrophils and macrophages in the pathogenesis of necrotizing enterocolitis caused by Cronobacter sakazakii. Journal of Surgical Research. 2012; 172:18-28. DOI | PubMed
- Engevik MA, Yacyshyn MB, Engevik KA, Wang J, Darien B, Hassett DJ, Yacyshyn BR, Worrell RT. Human Clostridium difficile infection: altered mucus production and composition. American Journal of Physiology – Gastrointestinal and Liver Physiology. 2015; 308:G510-G524. DOI | PubMed
- Erkosar B, Storelli G, Mitchell M, Bozonnet L, Bozonnet N, Leulier F. Pathogen virulence impedes mutualist-mediated enhancement of host juvenile growth via inhibition of protein digestion. Cell Host & Microbe. 2015; 18:445-455. DOI | PubMed
- Espey MG. Role of oxygen gradients in shaping redox relationships between the human intestine and its microbiota. Free Radical Biology and Medicine. 2013; 55:130-140. DOI | PubMed
- Fabregat A, Sidiropoulos K, Garapati P, Gillespie M, Hausmann K, Haw R, Jassal B, Jupe S, Korninger F, McKay S, Matthews L, May B, Milacic M, Rothfels K, Shamovsky V, Webber M, Weiser J, Williams M, Wu G, Stein L, Hermjakob H, D’Eustachio P. The Reactome pathway Knowledgebase. Nucleic Acids Research. 2016; 44:D481-D487. DOI | PubMed
- Fanaro S, Chierici R, Guerrini P, Vigi V. Intestinal microflora in early infancy: composition and development. Acta Paediatrica. 2003; 91:48-55. DOI | PubMed
- Favier CF, Vaughan EE, De Vos WM, Akkermans AD. Molecular monitoring of succession of bacterial communities in human neonates. Applied and Environmental Microbiology. 2002; 68:219-226. DOI | PubMed
- Finkbeiner SR, Zeng XL, Utama B, Atmar RL, Shroyer NF, Estes MK. Stem cell-derived human intestinal organoids as an infection model for rotaviruses. mBio. 2012; 3:e00159-12-12. DOI | PubMed
- Finkbeiner SR, Hill DR, Altheim CH, Dedhia PH, Taylor MJ, Tsai YH, Chin AM, Mahe MM, Watson CL, Freeman JJ, Nattiv R, Thomson M, Klein OD, Shroyer NF, Helmrath MA, Teitelbaum DH, Dempsey PJ, Spence JR. Transcriptome-wide Analysis Reveals Hallmarks of Human Intestine Development and Maturation In Vitro and In Vivo. Stem Cell Reports. 2015;1140-1155. DOI | PubMed
- Fisher EM, Khan M, Salisbury R, Kuppusamy P. Noninvasive monitoring of small intestinal oxygen in a rat model of chronic mesenteric ischemia. Cell Biochemistry and Biophysics. 2013; 67:451-459. DOI | PubMed
- Forbester JL, Goulding D, Vallier L, Hannan N, Hale C, Pickard D, Mukhopadhyay S, Dougan G. Interaction of salmonella enterica serovar typhimurium with intestinal organoids derived from human induced pluripotent stem cells. Infection and Immunity. 2015; 83:2926-2934. DOI | PubMed
- Ford HR, Sorrells DL, Knisely AS. Inflammatory cytokines, nitric oxide, and necrotizing enterocolitis. Seminars in pediatric surgery. 1996; 5:155-159. PubMed
- Ford H, Watkins S, Reblock K, Rowe M. The role of inflammatory cytokines and nitric oxide in the pathogenesis of necrotizing enterocolitis. Journal of Pediatric Surgery. 1997; 32:275-282. DOI | PubMed
- Forster R, Chiba K, Schaeffer L, Regalado SG, Lai CS, Gao Q, Kiani S, Farin HF, Clevers H, Cost GJ, Chan A, Rebar EJ, Urnov FD, Gregory PD, Pachter L, Jaenisch R, Hockemeyer D. Human intestinal tissue with adult stem cell properties derived from pluripotent stem cells. Stem Cell Reports. 2014; 2:838-852. DOI | PubMed
- Furuyama K, Kawaguchi Y, Akiyama H, Horiguchi M, Kodama S, Kuhara T, Hosokawa S, Elbahrawy A, Soeda T, Koizumi M, Masui T, Kawaguchi M, Takaori K, Doi R, Nishi E, Kakinoki R, Deng JM, Behringer RR, Nakamura T, Uemoto S. Continuous cell supply from a Sox9-expressing progenitor zone in adult liver, exocrine pancreas and intestine. Nature Genetics. 2011; 43:34-41. DOI | PubMed
- Fusunyan RD, Nanthakumar NN, Baldeon ME, Walker WA. Evidence for an innate immune response in the immature human intestine: toll-like receptors on fetal enterocytes. Pediatric Research. 2001; 49:589-593. DOI | PubMed
- Ganz T. Defensins: antimicrobial peptides of innate immunity. Nature Reviews Immunology. 2003; 3:710-720. DOI | PubMed
- García-Lafuente A, Antolín M, Guarner F, Crespo E, Malagelada JR. Modulation of colonic barrier function by the composition of the commensal flora in the rat. Gut. 2001; 48:503-507. DOI | PubMed
- Gene Ontology Consortium. Gene ontology consortium: going forward. Nucleic acids research. 2015; 43:D1049-1056. DOI | PubMed
- Gensollen T, Iyer SS, Kasper DL, Blumberg RS. How colonization by microbiota in early life shapes the immune system. Science. 2016; 352:539-544. DOI | PubMed
- Gerdes J, Lemke H, Baisch H, Wacker HH, Schwab U, Stein H. Cell cycle analysis of a cell proliferation-associated human nuclear antigen defined by the monoclonal antibody Ki-67. Journal of immunology. 1984; 133:1710-1715. PubMed
- Gilmore TD. Introduction to NF-kappaB: players, pathways, perspectives. Oncogene. 2006; 25:6680-6684. DOI | PubMed
- Glover LE, Lee JS, Colgan SP. Oxygen metabolism and barrier regulation in the intestinal mucosa. Journal of Clinical Investigation. 2016; 126:3680-3688. DOI | PubMed
- Gosalbes MJ, Llop S, Vallès Y, Moya A, Ballester F, Francino MP. Meconium microbiota types dominated by lactic acid or enteric bacteria are differentially associated with maternal eczema and respiratory problems in infants. Clinical & Experimental Allergy. 2013; 43:198-211. DOI | PubMed
- Goto Y, Obata T, Kunisawa J, Sato S, Ivanov II, Lamichhane A, Takeyama N, Kamioka M, Sakamoto M, Matsuki T, Setoyama H, Imaoka A, Uematsu S, Akira S, Domino SE, Kulig P, Becher B, Renauld JC, Sasakawa C, Umesaki Y, Benno Y, Kiyono H. Innate lymphoid cells regulate intestinal epithelial cell glycosylation. Science. 2014; 345DOI | PubMed
- Greenwood C, Morrow AL, Lagomarcino AJ, Altaye M, Taft DH, Yu Z, Newburg DS, Ward DV, Schibler KR. Early empiric antibiotic use in preterm infants is associated with lower bacterial diversity and higher relative abundance of Enterobacter. The Journal of Pediatrics. 2014; 165:23-29. DOI | PubMed
- Grenz A, Clambey E, Eltzschig HK. Hypoxia signaling during intestinal ischemia and inflammation. Current Opinion in Critical Care. 2012; 18:178-185. DOI | PubMed
- Gruette FK, Horn R, Haenel H. [Nutrition and biochemical micro-ecological processes in the rectum of infants]. Zeitschrift Fur Kinderheilkunde. 1965; 93:28-39. PubMed
- Hackam DJ, Good M, Sodhi CP. Mechanisms of gut barrier failure in the pathogenesis of necrotizing enterocolitis: Toll-like receptors throw the switch. Seminars in Pediatric Surgery. 2013; 22:76-82. DOI | PubMed
- Halpern MD, Holubec H, Dominguez JA, Meza YG, Williams CS, Ruth MC, McCuskey RS, Dvorak B. Hepatic inflammatory mediators contribute to intestinal damage in necrotizing enterocolitis. American Journal of Physiology – Gastrointestinal and Liver Physiology. 2003; 284:G695-G702. DOI | PubMed
- Hansson GC. Role of mucus layers in gut infection and inflammation. Current Opinion in Microbiology. 2012; 15:57-62. DOI | PubMed
- Harder J, Siebert R, Zhang Y, Matthiesen P, Christophers E, Schlegelberger B, Schröder JM. Mapping of the gene encoding human beta-defensin-2 (DEFB2) to chromosome region 8p22-p23.1. Genomics. 1997; 46:472-475. DOI | PubMed
- Henry L, Wickham H, Chang W. Ggstance: horizontal ’ggplot2’ components. Note: R package version 0.3. 2016.
- Hill DR, Spence JR. Gastrointestinal Organoids: Understanding the molecular basis of the host-microbe interface. Cellular and Molecular Gastroenterology and Hepatology. 2017; 3:138-149. DOI | PubMed
- Hill DR. Github. 2017a. Publisher Full Text
- Hill DR. Github. 2017b. Publisher Full Text
- Hooper LV, Xu J, Falk PG, Midtvedt T, Gordon JI. A molecular sensor that allows a gut commensal to control its nutrient foundation in a competitive ecosystem. PNAS. 1999; 96:9833-9838. DOI | PubMed
- Hviid A, Svanström H, Frisch M. Antibiotic use and inflammatory bowel diseases in childhood. Gut. 2011; 60:49-54. DOI | PubMed
- Ijssennagger N, Belzer C, Hooiveld GJ, Dekker J, van Mil SW, Müller M, Kleerebezem M, van der Meer R, Müller M. Gut microbiota facilitates dietary heme-induced epithelial hyperproliferation by opening the mucus barrier in colon. PNAS. 2015; 112:10038-10043. DOI | PubMed
- Johansson ME, Hansson GC. Immunological aspects of intestinal mucus and mucins. Nature Reviews Immunology. 2016; 16:639-649. DOI | PubMed
- Josephson R, Sykes G, Liu Y, Ording C, Xu W, Zeng X, Shin S, Loring J, Maitra A, Rao MS, Auerbach JM. A molecular scheme for improved characterization of human embryonic stem cell lines. BMC Biology. 2006; 4DOI | PubMed
- Kaiko GE, Ryu SH, Koues OI, Collins PL, Solnica-Krezel L, Pearce EJ, Pearce EL, Oltz EM, Stappenbeck TS. The colonic crypt protects stem cells from microbiota-derived metabolites. Cell. 2016; 165:1708-1720. DOI | PubMed
- Kanehisa M, Goto S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Research. 2000; 28:27-30. DOI | PubMed
- Kawai T, Akira S. Signaling to NF-kappaB by Toll-like receptors. Trends in Molecular Medicine. 2007; 13:460-469. DOI | PubMed
- Kelly CJ, Zheng L, Campbell EL, Saeedi B, Scholz CC, Bayless AJ, Wilson KE, Glover LE, Kominsky DJ, Magnuson A, Weir TL, Ehrentraut SF, Pickel C, Kuhn KA, Lanis JM, Nguyen V, Taylor CT, Colgan SP. Crosstalk between microbiota-derived short-chain fatty acids and intestinal epithelial hif augments tissue barrier function. Cell Host & Microbe. 2015; 17:662-671. DOI | PubMed
- Khailova L, Dvorak K, Arganbright KM, Halpern MD, Kinouchi T, Yajima M, Dvorak B. Bifidobacterium bifidum improves intestinal integrity in a rat model of necrotizing enterocolitis. AJP: Gastrointestinal and Liver Physiology. 2009; 297:G940-G949. DOI | PubMed
- Kim YS, Ho SB. Intestinal goblet cells and mucins in health and disease: recent insights and progress. Current Gastroenterology Reports. 2010; 12:319-330. DOI | PubMed
- Kim YG, Sakamoto K, Seo SU, Pickard JM, Gillilland MG, Pudlo NA, Hoostal M, Li X, Wang TD, Feehley T, Stefka AT, Schmidt TM, Martens EC, Fukuda S, Inohara N, Nagler CR, Núñez G, Nðñez G. Neonatal acquisition of Clostridia species protects against colonization by bacterial pathogens. Science. 2017; 356:315-319. DOI | PubMed
- Kishore N, Sommers C, Mathialagan S, Guzova J, Yao M, Hauser S, Huynh K, Bonar S, Mielke C, Albee L, Weier R, Graneto M, Hanau C, Perry T, Tripp CS. A selective IKK-2 inhibitor blocks NF-kappa B-dependent gene expression in interleukin-1 beta-stimulated synovial fibroblasts. Journal of Biological Chemistry. 2003; 278:32861-32871. DOI | PubMed
- Kisich KO, Heifets L, Higgins M, Diamond G. Antimycobacterial agent based on mRNA encoding human beta-defensin 2 enables primary macrophages to restrict growth of Mycobacterium tuberculosis. Infection and Immunity. 2001; 69:2692-2699. DOI | PubMed
- Koenig JE, Spor A, Scalfone N, Fricker AD, Stombaugh J, Knight R, Angenent LT, Ley RE. Succession of microbial consortia in the developing infant gut microbiome. PNAS. 2011; 108 Suppl 1:4578-4585. DOI | PubMed
- Koong AC, Chen EY, Giaccia AJ. Hypoxia causes the activation of nuclear factor kappa B through the phosphorylation of I kappa B alpha on tyrosine residues. Cancer Research. 1994; 54:1425-1430. PubMed
- Kremer N, Philipp EE, Carpentier MC, Brennan CA, Kraemer L, Altura MA, Augustin R, Häsler R, Heath-Heckman EA, Peyer SM, Schwartzman J, Rader BA, Ruby EG, Rosenstiel P, McFall-Ngai MJ. Initial symbiont contact orchestrates host-organ-wide transcriptional changes that prime tissue colonization. Cell Host & Microbe. 2013; 14:183-194. DOI | PubMed
- Lebenthal A, Lebenthal E. The ontogeny of the small intestinal epithelium. Journal of Parenteral and Enteral Nutrition. 1999; 23:S3-S6. DOI | PubMed
- Leslie JL, Huang S, Opp JS, Nagy MS, Kobayashi M, Young VB, Spence JR. Persistence and toxin production by Clostridium difficile within human intestinal organoids result in disruption of epithelial paracellular barrier function. Infection and Immunity. 2015; 83:138-145. DOI | PubMed
- Li H, Limenitakis JP, Fuhrer T, Geuking MB, Lawson MA, Wyss M, Brugiroux S, Keller I, Macpherson JA, Rupp S, Stolp B, Stein JV, Stecher B, Sauer U, McCoy KD, Macpherson AJ. The outer mucus layer hosts a distinct intestinal microbial niche. Nature Communications. 2015; 6DOI | PubMed
- Litvak V, Ramsey SA, Rust AG, Zak DE, Kennedy KA, Lampano AE, Nykter M, Shmulevich I, Aderem A. Function of C/EBPdelta in a regulatory circuit that discriminates between transient and persistent TLR4-induced signals. Nature Immunology. 2009; 10:437-443. DOI | PubMed
- Love MI, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 2014; 15DOI | PubMed
- Luo W, Brouwer C. Pathview: an R/Bioconductor package for pathway-based data integration and visualization. Bioinformatics. 2013; 29:1830-1831. DOI | PubMed
- Malago JJ. Contribution of microbiota to the intestinal physicochemical barrier. Beneficial Microbes. 2015; 6:295-311. DOI | PubMed
- Marcobal A, Southwick AM, Earle KA, Sonnenburg JL. A refined palate: bacterial consumption of host glycans in the gut. Glycobiology. 2013; 23:1038-1046. DOI | PubMed
- Matsuo Y, Nishizaki C, Drexler HG. Efficient DNA fingerprinting method for the identification of cross-culture contamination of cell lines. Human cell. 1999; 12:149-154. PubMed
- McCracken KW, Howell JC, Wells JM, Spence JR. Generating human intestinal tissue from pluripotent stem cells in vitro. Nature Protocols. 2011; 6:1920-1928. DOI | PubMed
- McCracken KW, Catá EM, Crawford CM, Sinagoga KL, Schumacher M, Rockich BE, Tsai YH, Mayhew CN, Spence JR, Zavros Y, Wells JM. Modelling human development and disease in pluripotent stem-cell-derived gastric organoids. Nature. 2014; 516:400-404. DOI | PubMed
- McLoughlin K, Schluter J, Rakoff-Nahoum S, Smith AL, Foster KR. Host selection of microbiota via differential adhesion. Cell Host & Microbe. 2016; 19:550-559. DOI | PubMed
- Michielan A, D’Incà R. Intestinal Permeability in Inflammatory bowel disease: pathogenesis, clinical evaluation, and therapy of leaky gut. Mediators of inflammation. 2015; 2015DOI | PubMed
- Miyoshi H, Ajima R, Luo CT, Yamaguchi TP, Stappenbeck TS. Wnt5a potentiates TGF-β signaling to promote colonic crypt regeneration after tissue injury. Science. 2012; 338:108-113. DOI | PubMed
- Miyoshi H, Stappenbeck TS. In vitro expansion and genetic modification of gastrointestinal stem cells in spheroid culture. Nature Protocols. 2013; 8:2471-2482. DOI | PubMed
- Mori-Akiyama Y, van den Born M, van Es JH, Hamilton SR, Adams HP, Zhang J, Clevers H, de Crombrugghe B. SOX9 is required for the differentiation of paneth cells in the intestinal epithelium. Gastroenterology. 2007; 133:539-546. DOI | PubMed
- Morrow AL, Lagomarcino AJ, Schibler KR, Taft DH, Yu Z, Wang B, Altaye M, Wagner M, Gevers D, Ward DV, Kennedy MA, Huttenhower C, Newburg DS. Early microbial and metabolomic signatures predict later onset of necrotizing enterocolitis in preterm infants. Microbiome. 2013; 1DOI | PubMed
- Muniz LR, Knosp C, Yeretssian G. Intestinal antimicrobial peptides during homeostasis, infection, and disease. Frontiers in Immunology. 2012; 3DOI | PubMed
- Ménard S, Förster V, Lotz M, Gütle D, Duerr CU, Gallo RL, Henriques-Normark B, Pütsep K, Andersson M, Glocker EO, Hornef MW, Ménard S, Förster V, Gütle D, Pütsep K. Developmental switch of intestinal antimicrobial peptide expression. The Journal of Experimental Medicine. 2008; 205:183-193. DOI | PubMed
- Nanthakumar N, Meng D, Goldstein AM, Zhu W, Lu L, Uauy R, Llanos A, Claud EC, Walker WA. The mechanism of excessive intestinal inflammation in necrotizing enterocolitis: an immature innate immune response. PLoS One. 2011; 6DOI | PubMed
- Neal JT, Peterson TS, Kent ML, Guillemin K. H. pylori virulence factor CagA increases intestinal cell proliferation by Wnt pathway activation in a transgenic zebrafish model. Disease Models & Mechanisms. 2013; 6:802-810. DOI | PubMed
- Neu J. Gastrointestinal maturation and implications for infant feeding. Early Human Development. 2007; 83:767-775. DOI | PubMed
- Neu J, Walker WA. Necrotizing enterocolitis. New England Journal of Medicine. 2011; 364:255-264. DOI | PubMed
- Nguyen TL, Vieira-Silva S, Liston A, Raes J. How informative is the mouse for human gut microbiota research?. Disease Models & Mechanisms. 2015; 8:1-16. DOI | PubMed
- O’Neil DA, O’Neil DA. Regulation of expression of beta-defensins: endogenous enteric peptide antibiotics. Molecular Immunology. 2003; 40:445-450. DOI | PubMed
- Ochman H, Selander RK. Standard reference strains of Escherichia coli from natural populations. Journal of Bacteriology. 1984; 157:690-693. PubMed
- Oliver KM, Taylor CT, Cummins EP. Hypoxia. regulation of nfkappab signalling during inflammation: the role of hydroxylases. Arthritis Research & Therapy. 2009; 11:215. DOI | PubMed
- Ostaff MJ, Stange EF, Wehkamp J. Antimicrobial peptides and gut microbiota in homeostasis and pathology. EMBO Molecular Medicine. 2013; 5:1465-1483. DOI | PubMed
- Palmer C, Bik EM, DiGiulio DB, Relman DA, Brown PO. Development of the human infant intestinal microbiota. PLoS Biology. 2007; 5DOI | PubMed
- Peterson LW, Artis D. Intestinal epithelial cells: regulators of barrier function and immune homeostasis. Nature Reviews Immunology. 2014; 14:141-153. DOI | PubMed
- Renz H, Brandtzaeg P, Hornef M. The impact of perinatal immune development on mucosal homeostasis and chronic inflammation. Nature Reviews Immunology. 2011; 12:9-23. DOI | PubMed
- Rhee SJ, Walker WA, Cherayil BJ. Developmentally regulated intestinal expression of IFN-gamma and its target genes and the age-specific response to enteric Salmonella infection. The Journal of Immunology. 2005; 175:1127-1136. DOI | PubMed
- Rius J, Guma M, Schachtrup C, Akassoglou K, Zinkernagel AS, Nizet V, Johnson RS, Haddad GG, Karin M. NF-kappaB links innate immunity to the hypoxic response through transcriptional regulation of HIF-1alpha. Nature. 2008; 453:807-811. DOI | PubMed
- Rivera-Chávez F, Zhang LF, Faber F, Lopez CA, Byndloss MX, Olsan EE, Xu G, Velazquez EM, Lebrilla CB, Winter SE, Bäumler AJ. Depletion of butyrate-producing clostridia from the gut microbiota drives an aerobic luminal expansion of salmonella. Cell Host & Microbe. 2016; 19:443-454. DOI | PubMed
- Robinson J. Cochrane in context: probiotics for prevention of necrotizing enterocolitis in preterm infants. Evidence-Based Child Health: A Cochrane Review Journal. 2014; 9:672-674. DOI | PubMed
- Rodríguez-Piñeiro AM, Bergström JH, Ermund A, Gustafsson JK, Schütte A, Johansson ME, Hansson GC. Studies of mucus in mouse stomach, small intestine, and colon. II. Gastrointestinal mucus proteome reveals Muc2 and Muc5ac accompanied by a set of core proteins. AJP: Gastrointestinal and Liver Physiology. 2013; 305:G348-G356. DOI | PubMed
- Round JL, Mazmanian SK. The gut microbiota shapes intestinal immune responses during health and disease. Nature Reviews Immunology. 2009; 9:313-323. DOI | PubMed
- Salzman NH, Ghosh D, Huttner KM, Paterson Y, Bevins CL. Protection against enteric salmonellosis in transgenic mice expressing a human intestinal defensin. Nature. 2003; 422:522-526. DOI | PubMed
- Salzman NH, Hung K, Haribhai D, Chu H, Karlsson-Sjöberg J, Amir E, Teggatz P, Barman M, Hayward M, Eastwood D, Stoel M, Zhou Y, Sodergren E, Weinstock GM, Bevins CL, Williams CB, Bos NA. Enteric defensins are essential regulators of intestinal microbial ecology. Nature Immunology. 2010; 11:76-82. DOI | PubMed
- Sander LE, Davis MJ, Boekschoten MV, Amsen D, Dascher CC, Ryffel B, Swanson JA, Müller M, Blander JM. Detection of prokaryotic mRNA signifies microbial viability and promotes immunity. Nature. 2011; 474:385-389. DOI | PubMed
- Sanderson IR, Walker WA. Development of the Gastrointestinal Tract. BC Decker: Hamilton, Canada; 2000.
- Schmidt TM, Kao JY. A little O2 may go a long way in structuring the GI microbiome. Gastroenterology. 2014; 147:956-959. DOI | PubMed
- Shaw SY, Blanchard JF, Bernstein CN. Association between the use of antibiotics in the first year of life and pediatric inflammatory bowel disease. The American Journal of Gastroenterology. 2010; 105:2687-2692. DOI | PubMed
- Shawki A, McCole DF. Mechanisms of intestinal epithelial barrier dysfunction by adherent-invasive escherichia coli. Cellular and Molecular Gastroenterology and Hepatology. 2017; 3:41-50. DOI | PubMed
- Shreiner AB, Kao JY, Young VB. The gut microbiome in health and in disease. Current Opinion in Gastroenterology. 2015; 31:69-75. DOI | PubMed
- Sommer F, Nookaew I, Sommer N, Fogelstrand P, Bäckhed F. Site-specific programming of the host epithelial transcriptome by the gut microbiota. Genome Biology. 2015; 16DOI | PubMed
- Spence JR, Mayhew CN, Rankin SA, Kuhar MF, Vallance JE, Tolle K, Hoskins EE, Kalinichenko VV, Wells SI, Zorn AM, Shroyer NF, Wells JM. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 2011; 470:105-109. DOI | PubMed
- Stallman RM. EMACS the extensible, customizable self-documenting display editor. ACM SIGOA Newsletter. 1981; 2:147-156. DOI
- Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA, Paulovich A, Pomeroy SL, Golub TR, Lander ES, Mesirov JP. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. PNAS. 2005; 102:15545-15550. DOI | PubMed
- Tan X, Hsueh W, Gonzalez-Crussi F. Cellular localization of tumor necrosis factor (TNF)-alpha transcripts in normal bowel and in necrotizing enterocolitis. TNF gene expression by Paneth cells, intestinal eosinophils, and macrophages. The American Journal of Pathology. 1993; 142:1858-1865. PubMed
- Tanner SM, Berryhill TF, Ellenburg JL, Jilling T, Cleveland DS, Lorenz RG, Martin CA. Pathogenesis of necrotizing enterocolitis: modeling the innate immune response. The American journal of pathology. 2015; 185:4-16. DOI | PubMed
- Tsai YH, Nattiv R, Dedhia PH, Nagy MS, Chin AM, Thomson M, Klein OD, Spence JR. In vitro patterning of pluripotent stem cell-derived intestine recapitulates in vivo human development. Development. 2017; 144:1045-1055. DOI | PubMed
- Tsutsumi-Ishii Y, Nagaoka I. NF-kappa B-mediated transcriptional regulation of human beta-defensin-2 gene following lipopolysaccharide stimulation. Journal of Leukocyte Biology. 2002; 71:154-162. PubMed
- Turner JR. Intestinal mucosal barrier function in health and disease. Nature Reviews Immunology. 2009; 9:799-809. DOI | PubMed
- Underwood MA, Arriola J, Gerber CW, Kaveti A, Kalanetra KM, Kananurak A, Bevins CL, Mills DA, Dvorak B. Bifidobacterium longum subsp. infantis in experimental necrotizing enterocolitis: alterations in inflammation, innate immune response, and the microbiota. Pediatric Research. 2014; 76:326-333. DOI | PubMed
- Upperman JS, Potoka D, Grishin A, Hackam D, Zamora R, Ford HR. Mechanisms of nitric oxide-mediated intestinal barrier failure in necrotizing enterocolitis. Seminars in Pediatric Surgery. 2005; 14:159-166. DOI | PubMed
- Vaishnava S, Behrendt CL, Ismail AS, Eckmann L, Hooper LV. Paneth cells directly sense gut commensals and maintain homeostasis at the intestinal host-microbial interface. PNAS. 2008; 105:20858-20863. DOI | PubMed
- Varki A. Essentials of Glycobiology. Cold Spring Harbor Laboratory Press: Cold Spring Harbor, New York; 2017.
- Veereman-Wauters G. Neonatal gut development and postnatal adaptation. European Journal of Pediatrics. 1996; 155:627-632. DOI | PubMed
- Vora P, Youdim A, Thomas LS, Fukata M, Tesfay SY, Lukasek K, Michelsen KS, Wada A, Hirayama T, Arditi M, Abreu MT. Beta-defensin-2 expression is regulated by TLR signaling in intestinal epithelial cells. The Journal of Immunology. 2004; 173:5398-5405. DOI | PubMed
- Wang F, Graham WV, Wang Y, Witkowski ED, Schwarz BT, Turner JR. Interferon-gamma and tumor necrosis factor-alpha synergize to induce intestinal epithelial barrier dysfunction by up-regulating myosin light chain kinase expression. The American Journal of Pathology. 2005; 166:409-419. DOI | PubMed
- Wang F, Schwarz BT, Graham WV, Wang Y, Su L, Clayburgh DR, Abraham C, Turner JR. IFN-gamma-induced TNFR2 expression is required for TNF-dependent intestinal epithelial barrier dysfunction. Gastroenterology. 2006; 131:1153-1163. DOI | PubMed
- Wang G, Li X, Wang Z. APD3: the antimicrobial peptide database as a tool for research and education. Nucleic Acids Research. 2016; 44:D1087-D1093. DOI | PubMed
- Ward DV, Scholz M, Zolfo M, Taft DH, Schibler KR, Tett A, Segata N, Morrow AL. Metagenomic sequencing with strain-level resolution implicates uropathogenic e. coli in necrotizing enterocolitis and mortality in preterm infants. Cell Reports. 2016; 14:2912-2924. DOI | PubMed
- Watson CL, Mahe MM, Múnera J, Howell JC, Sundaram N, Poling HM, Schweitzer JI, Vallance JE, Mayhew CN, Sun Y, Grabowski G, Finkbeiner SR, Spence JR, Shroyer NF, Wells JM, Helmrath MA. An in vivo model of human small intestine using pluripotent stem cells. Nature Medicine. 2014; 20:1310-1314. DOI | PubMed
- Wattam AR, Davis JJ, Assaf R, Boisvert S, Brettin T, Bun C, Conrad N, Dietrich EM, Disz T, Gabbard JL, Gerdes S, Henry CS, Kenyon RW, Machi D, Mao C, Nordberg EK, Olsen GJ, Murphy-Olson DE, Olson R, Overbeek R, Parrello B, Pusch GD, Shukla M, Vonstein V, Warren A, Xia F, Yoo H, Stevens RL. Improvements to patric, the all-bacterial bioinformatics database and analysis resource center. Nucleic Acids Research. 2017; 45:D535-D542. DOI | PubMed
- Wickham H. Ggplot2: elegant graphics for data analysis. Springer-Verlag New York. 2009.
- Wopereis H, Oozeer R, Knipping K, Belzer C, Knol J. The first thousand days – intestinal microbiology of early life: establishing a symbiosis. Pediatric Allergy and Immunology. 2014; 25:428-438. DOI | PubMed
- Wullaert A, Bonnet MC, Pasparakis M. NF-κB in the regulation of epithelial homeostasis and inflammation. Cell Research. 2011; 21:146-158. DOI | PubMed
- Xiao C, Ghosh S. NF-kappaB, an evolutionarily conserved mediator of immune and inflammatory responses. Advances in Experimental Medicine and Biology. 2005; 560:41-45. DOI | PubMed
- Xie L, Xue X, Taylor M, Ramakrishnan SK, Nagaoka K, Hao C, Gonzalez FJ, Shah YM. Hypoxia-inducible factor/MAZ-dependent induction of caveolin-1 regulates colon permeability through suppression of occludin, leading to hypoxia-induced inflammation. Molecular and Cellular Biology. 2014; 34:3013-3023. DOI | PubMed
- Yassour M, Vatanen T, Siljander H, Hämäläinen AM, Härkönen T, Ryhänen SJ, Franzosa EA, Vlamakis H, Huttenhower C, Gevers D, Lander ES, Knip M, Xavier RJ, DIABIMMUNE Study Group. Natural history of the infant gut microbiome and impact of antibiotic treatment on bacterial strain diversity and stability. Science Translational Medicine. 2016; 8DOI | PubMed
- Yu G, Wang LG, Han Y, He QY. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS: A Journal of Integrative Biology. 2012; 16:284-287. DOI | PubMed
- Yu G, He QY. ReactomePA: an R/Bioconductor package for reactome pathway analysis and visualization. Mol. BioSyst.. 2016; 12:477-479. DOI | PubMed
- Zeitouni NE, Chotikatum S, von Köckritz-Blickwede M, Naim HY. The impact of hypoxia on intestinal epithelial cell functions: consequences for invasion by bacterial pathogens. Molecular and Cellular Pediatrics. 2016; 3DOI | PubMed
- Zhang G, Ghosh S. Toll-like receptor-mediated NF-kappaB activation: a phylogenetically conserved paradigm in innate immunity. Journal of Clinical Investigation. 2001; 107:13-19. DOI | PubMed
- Zheng L, Kelly CJ, Colgan SP. Physiologic hypoxia and oxygen homeostasis in the healthy intestine. A Review in the Theme: Cellular Responses to Hypoxia. American Journal of Physiology – Cell Physiology. 2015; 309:C350-C360. DOI | PubMed
Fonte
Hill DR, Huang S, Nagy MS, Yadagiri VK, Fields C, et al. () Bacterial colonization stimulates a complex physiological response in the immature human intestinal epithelium. eLife 6e29132. https://doi.org/10.7554/eLife.29132





