Abstract
Introduzione
La famiglia dei recettori del fattore di crescita epidermico (EGFR) delle tirosina chinasi dei recettori svolge un ruolo importante nella segnalazione proliferativa di una varietà di tumori(Baselga e Swain, 2009; Yarden e Pines, 2012). Oltre all’EGFR (noto anche come ErbB1), la famiglia è composta dal recettore orfano HER2 (ErbB2), la pseudochinasi HER3 (ErbB3) e HER4 (ErbB4). La sovraespressione di HER2 è un driver oncogeno in circa il 20% di tutti i tumori al seno(Lovekin et al., 1991; Owens et al., 2004; Slamon et al., 1987). L’elevata rilevanza clinica di questi recettori li ha resi un bersaglio per la terapia diretta sia con anticorpi che con inibitori delle piccole molecole chinasi. Nel caso del tumore al seno HER2+, l’anticorpo monoclonale trastuzumab (Herceptin) e il suo derivato citotossico trastuzumab-emtansina (Kadcyla), l’anticorpo monoclonale che blocca la dimerizzazione pertuzumab HER2-HER3 (Perjeta) e l’inibitore della piccola molecola chinasi lapatinib (Tykerb/Tyverb) hanno avuto successo in clinica(Blackwell et al.,2010; Cameron et al., 2017; Diéras et al., 2017; Geyer et al., 2006; Krop et al., 2017; CLEOPATRA Study Group et al., 2015; EMILIA Study Group et al., 2012).
Mentre HER2 non ha un ligando noto, HER3 lega il fattore di crescita neuregulina (NRG, noto anche come eregulina o HRG) per indurre l’eterodimerizzazione e la segnalazione(Sliwkowski et al., 1994). HER3 è stato implicato nella resistenza terapeutica alla terapia mirata a HER2 attraverso una varietà di meccanismi, tra cui la refosforilazione dei recettori, la sovraespressione di HER3 e l’aumento della produzione di NRG (rivisto in[Claus et al., 2014]). In termini di segnalazione cellulare in risposta all’inibizione delle chinasi della famiglia HER, il buffering mediato da HER3 attraverso l’asse di segnalazione Akt/PKB ha dimostrato di essere un fattore importante nella resistenza terapeutica(Sergina et al., 2007).
La dimerizzazione dei membri della famiglia EGFR è un processo fluido mediato da dinamiche di interazione praticamente in ogni dominio del recettore. Per l’EGFR, il dimero attivo legato al ligando mostra un’interazione eretta, back-to-back extracellulare (ECD) in cui entrambi i recettori hanno legato il ligando, anche se possono verificarsi anche dimeri legati singolarmente(Garrett et al., 2002; Liu et al., 2012; Ogiso et al., 2002). Sebbene HER2 non abbia un ligando noto, adotta nativamente questa conformazione dell’ectodominio eretto, pronto per la dimerizzazione(Garrett et al., 2002). Sul lato intracellulare, la formazione del dimero del dominio chinasi attivo è criticamente influenzata dalla conformazione del dominio della juxtamembrana (JMD)(Jura et al., 2009a; Thiel e Carpenter, 2007). I domini chinasi si associano in un dimero asimmetrico, che assomiglia all’interfaccia del dimero asimmetrico CDK/ciclina(Jeffrey et al., 1995; Zhang et al., 2006). In questo dimero canonico, una chinasi (l'”attivatore”) permette al partner di dimerizzazione (il “ricevitore”) di adottare una conformazione attiva e diventare cataliticamente attivo. Queste varie conformazioni sono state osservate anche in recettori quasi completi utilizzando la microscopia elettronica a macchia negativa(Mi et al., 2011). Da notare in queste formazioni dimeriche dei recettori è stata la mancanza di interazioni attive e asimmetriche nel dominio delle chinasi quando il recettore era legato all’inibitore competitivo ATP lapatinib(Mi et al., 2011). Sebbene queste interazioni siano state descritte principalmente nel contesto dell’omogeneizzazione dell’EGFR, esse rimangono un modello per le interazioni del resto della famiglia EGFR. La conformazione dell’interazione nel dominio della chinasi attiva è stata convalidata per EGFR-HER3 e HER2-HER3(Jura et al., 2009b; Littlefield et al., 2014; van Lengerich et al., 2017).
Una moltitudine di studi, utilizzando una varietà di tecniche, hanno confermato che i recettori della famiglia EGFR possono formare oligomeri di ordine superiore e che l’esatta natura di questi oligomeri è modulata da una varietà di condizioni, tra cui la densità dei recettori, la presenza di ligandi, il tipo di ligandi e il comportamento della membrana dipendente dalla temperatura(Clayton et al.,2005; Clayton et al., 2007; Huang et al., 2016; Nagy et al., 2010; Needham et al., 2016; Saffarian et al., 2007; van Lengerich et al., 2017; Yang et al., 2007; Zhang et al., 2017).
Sullo sfondo di una tale moltitudine di modalità di associazione, è chiaro che le dinamiche conformazionali e i riarrangiamenti strutturali sono un regolatore integrale del comportamento proteico nella famiglia EGFR.
Abbiamo dimostrato in precedenza che all’interno di una chinasi, in questo caso PKCε, l’occupazione della tasca di legame nucleotidico con ATP (o un inibitore) è uno dei principali determinanti del comportamento delle proteine, conferendo la stabilità strutturale necessaria per le interazioni proteine-proteine e per i siti di priming per essere fosforilati in modo stabile (Cameron etal., 2009). Effetti simili sono stati osservati in diverse chinasi aggiuntive, tra cui PKB/Akt, IRE1, e AMPK(Okuzumi et al., 2009; Papa et al., 2003; Ross et al., 2017; Wang et al., 2012).
Un esempio degno di nota è stato descritto per la famiglia RAF, originariamente in cRAF, dove l’inibitore SB 203580 induceva paradossalmente un’attività indotta dall’inibitore SB 203580(Eyers et al., 1998). Più recentemente, un fenomeno simile è stato mostrato in BRAF, dove l’inibitore della piccola molecola chinasi vemurafenib blocca il mutante oncogeno V600E, ma stabilizza la proteina di tipo selvatico, promuovendo la segnalazione proliferativa a valle(Hatzivassiliou et al., 2010; McKay et al., 2011; Poulikakos et al., 2010; Thevakumaran et al., 2015). All’interno della famiglia EGFR, noi e altri abbiamo dimostrato in precedenza che gli inibitori della chinazolina possono causare la formazione di omodimeri dell’EGFR e l’eterodimerizzazione dell’EGFR-MET, stabilizzando particolari conformatori del dominio chinasi(Arteaga et al., 1997; Bublil et al., 2010; Lichtner et al., 2001; Ortiz-Zapater et al., 2017).
Il ruolo strutturale e conformazionale che l’occupazione delle tasche nucleotidiche può svolgere è particolarmente interessante nel contesto delle pseudochinasi, che hanno perso la loro attività catalitica. L’analisi delle sequenze mostra che molte pseudochinasi conservano molti dei residui conservati coinvolti nella legatura ATP(Boudeau et al., 2006; Claus et al., 2013). L’analisi in vitro dello pseudochinoma ha mostrato che molte pseudochinasi hanno capacità di legame nucleotidico(Murphy et al., 2014).
Nel caso di queste pseudochinasi leganti ATP, in cui il legame nucleotidico non provoca il fosfotrasferimento, la stabilità strutturale conferita dal legame ATP può essere parte integrante della funzione proteica. Questo è stato osservato per la pseudochinasi STRAD, che richiede il legame ATP per sostenere un complesso eterotrimerico con LKB e MO25(Zeqiraj et al., 2009a; Zeqiraj et al., 2009b). Allo stesso modo, nella pseudochinasi FAM20A ATP-binding, sebbene in un orientamento non canonico, è essenziale per stabilizzare il complesso FAM20A/FAM20C(Cui et al., 2015; Cui et al., 2017). Il legame ATP è un requisito strutturale per il mutante JAK2 JH2 V617F per promuovere la segnalazione patogena(Hammarén et al., 2015). Nella pseudochinasi MLKL, l’occupazione della tasca di legame ATP è essenziale per la traslocazione della membrana e il suo ruolo nella segnalazione necroptotica(Hildebrand et al., 2014; Murphy et al., 2013).
HER3 è in grado di legare l’ATP (cristallizzato come PDB ID 3KEX, 3LMG), così come l’inibitore Src/ABL Bosutinib (PDB ID 4OTW)(Levinson e Boxer, 2014; Davis et al., 2011; Jura et al., 2009b; Murphy et al., 2014; Shi et al., 2010). Considerando l’importanza di HER3 come partner conformazionale nell’eterodimero HER2-HER3, e l’importanza consolidata del legame ATP per la formazione complessa in altre pseudochinasi, il ruolo dell’occupazione delle tasche di legame nucleotidico nella funzione HER3 giustifica un’indagine.
Qui, abbiamo integrato lo studio degli effetti conformazionali chinasi-autonomi dell’occupazione della tasca di legame nucleotidico con quello delle modalità di eterointerazione HER2-HER3 e dei fenotipi proliferativi a valle in risposta al trattamento farmacologico. Mostriamo che l’occupazione delle tasche nucleotidiche sia in HER2 che nella pseudochinasi HER3 è di grande importanza conformazionale per l’eterodimerizzazione del dominio chinasi e la successiva segnalazione proliferativa. Nelle cellule tumorali della mammella HER2+ questo porta ad una sinergia inaspettata tra il ligando HER3 NRG e l’inibitore HER2 lapatinib, con il quale il loro concomitante legame promuove la proliferazione nei sistemi di coltura 2D e 3D. Lapatinib è in grado di promuovere l’eterodimerizzazione tra i domini chinasi di HER2 e HER3 in cellule. Tuttavia, questa interfaccia dimero è diversa dal dimero attivo canonico della famiglia EGFR, ed è necessaria per il fenotipo proliferativo combinatorio di lapatinib/NRG. Sia l’eterodimero indotto da lapatinib che gli effetti di proliferazione cooperativa dipendono fortemente dalla capacità della pseudochinasi HER3 di legare l’ATP. Coerentemente con il modello, occupare la pseudochinasi HER3 con l’inibitore Src/Abl bosutinib stabilizza il dominio della pseudochinasi nella misura in cui promuove effettivamente l’eterodimerizzazione e la proliferazione a valle di HER2-HER3.
Risultati
Il co-trattamento Lapatinib-NRG mostra un effetto sinergico sulla proliferazione, dipendente dal legame HER3 ATP
La sensibilità di una varietà di linee cellulari oncogenetiche agli inibitori delle piccole molecole chinasi può essere contrastata dall’aggiunta di fattori di crescita(Wilson et al., 2012). Questo include il caso delle linee cellulari di tumore al seno HER2+ trattate con lapatinib, dove l’NRG è visto come mediatore di un salvataggio della tossicità dei farmaci(Novotny et al., 2016; Wilson et al., 2012). Utilizzando diverse procedure sperimentali, abbiamo studiato ulteriormente questi effetti concorrenti di lapatinib e NRG sul comportamento proliferativo delle cellule tumorali della mammella HER2+.
Nelle cellule SKBR3, BT474, AU565, e HCC1419 trattate con una gamma di concentrazioni di lapatinib per 72 ore, l’aggiunta di 10 nM NRG salva la citotossicità indotta da farmaci, tranne che a concentrazioni di farmaci molto elevate(Figura 1a, Figura 1-figure supplement 1a-c).
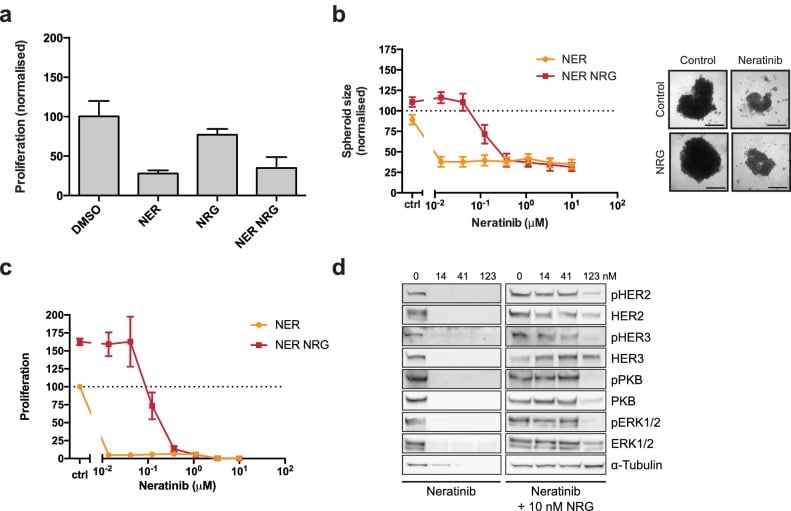
Figura 1-figure supplement 2-source data 1.Lapatinib e NRG hanno effetti sinergici sulla crescita SKBR3 nei sistemi di coltura 2D e 3D.dati numerici e statistiche relative alla Figura 1.Dati numerici e statistiche relative alla Figura 1.Effetti di lapatinib e NRG sulla proliferazione delle cellule tumorali della mammella.dati numerici e statistiche relative alla Figura 1-figure supplement 1.Dati numerici e statistiche relative alla Figura 1 . L’inibitore irreversibile neratinib non mostra una crescita sinergica in condizioni di co-trattamento del legante .(a) CellTiter-Glo proliferation test di cellule SKBR3 dopo il trattamento per 72 ore con una gamma di concentrazioni di lapatinib ± 10 nM NRG.b) Saggio di conteggio delle cellule SKBR3 trattate per 72 ore con DMSO o 250 nM lapatinib ±10 nM NRG, prima della quantificazione del numero di cellule su un contatore Vi-CELL.c) Quantificazione dell’area sferoidale SKBR3 3D dopo 8 giorni di trattamento con una gamma di concentrazioni di lapatinib ± 10 nM NRG, con micrografie rappresentative del campo luminoso. Barre di scala 0,5 mm. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti eseguiti ciascuno in quadruplicato. Dati corrispondenti e statistiche disponibili come Figura 1 – dati fonte 1.10.7554/eLife.32271.008Figure 1-source data 1.Numerical data e statistiche relative alla Figura 1.(a-f) BT474, AU565, HCC1419, ZR75, MCF7, e HCC1569 sono state saggiate per la proliferazione nella risposta alla lapatinib ±10 nM NRG come descritto nella Figura 1a. BT474 e AU565 rappresentano linee sensibili alla lapatinib. HCC1419 e ZR75 rappresentano linee parzialmente sensibili. MCF7 e HCC1569 rappresentano linee non sensibili alla lapatinib.(g) Analisi Western blot dei livelli di proteina della famiglia EGFR endogena in SKBR3, BT474, AU565, HCC1419, ZR75, MCF7 e HCC1569.(h-i) Le celluleSKBR3 e BT474 sono state trattate per 72 ore con una titolazione di lapatinib ±10 nM EGF, dopo di che la proliferazione è stata misurata utilizzando CellTiter-Glo.(j) CellTiter-Glo test di proliferazione CellTiter-Glo di cellule SKBR3 con siRNA transitorio abbattuto di HER3 utilizzando oligonucleotidi singoli. Western blot denota l’efficienza di abbattimento di HER3 si11 e HER3 si13 oligonucleotidi.k) CellTiter-Glo endpoint analisi della proliferazione di colture di sferoidi SKBR3 dopo 8 giorni di lapatinib ±NRG.(l) Analisi Western blot di colture di sferoidi SKBR3 in condizioni corrispondenti alla Figura 1c, Figura 1-figure supplement 1k. Viene mostrato un esempio rappresentativo di tre esperimenti indipendenti. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia, ad eccezione di (j), che rappresenta sei esperimenti indipendenti ciascuno eseguito in triplice copia. Dati corrispondenti e statistiche disponibili come figura 1-figure supplement 1-source data 1.10.7554/eLife.32271.005Figure 1-figure supplement 1-source data 1.Numerical data and statistics relating to Figure 1-figure supplement 1.a) Le cellule SKBR3 sono state trattate per 72 ore con DMSO o 250 nM neratinib ±10 nM NRG, prima della quantificazione del numero di cellule su un contatore Vi-CELL b) Quantificazione dell’area sferoidale dopo 8 giorni di trattamento con una titolazione di neratinib. Micrografie rappresentative del campo luminoso degli sferoidi 3D delle cellule SKBR3. Barre di scala 0,5 mm.c) Analisi CellTiter-Glo endpoint di colture di sferoidi da(b).(d) Analisi Western blot della segnalazione cellulare negli sferoidi SKBR3 dopo 8 giorni di trattamento. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia. Tutti i western blot mostrano un esempio rappresentativo di tre esperimenti indipendenti. I dati corrispondenti e le statistiche disponibili come Figura 1-figure supplemento 2-figure dati fonte 1.10.7554/eLife.32271.007Cifra 1-figure supplement 2 dati fonte 1.Dati numerici e statistiche relative alla Figura 1-figure supplement 1.
È interessante notare che, nel caso delle linee cellulari SKBR3, BT474 e AU565, basse concentrazioni di lapatinib (~ 40-400 nM) sono in grado di aumentare la proliferazione in combinazione con 10 nM NRG del 25-30% rispetto al solo fattore di crescita (Figura 1a,Figura 1-figure supplement 1a-b). Una risposta parziale di questo fenotipo cooperativo è osservata nelle cellule ZR75 e HCC1419(Figura 1-figure supplement 1c-d). Questo fenotipo nelle cellule SKBR3, mentre osservato in precedenza, è passato inosservato(Novotny et al., 2016; Wilson et al., 2012). Abbiamo corroborato i nostri risultati con un test di conteggio delle cellule, in cui le cellule SKBR3 sono stati trattati per 72 ore con 250 nM lapatinib o veicolo ± 10 nM NRG (Figura 1b). L’effetto emergente di lapatinib più NRG dipende dalla sensibilità di lapatinib. Due linee cellulari per il cancro al seno con bassa sensibilità alla lapatinib, MCF7 e HCC1569, mostrano una bassa cooperazione del fattore di crescita dell’inibitore(Figura 1-figure supplement 1e-f). Il fenotipo di crescita in ZR75 può essere parzialmente spiegato dalla sua espressione HER4, considerando che NRG è anche un ligando per HER4(Figura 1-figure supplement 1g).
Anche se HER3 ha dimostrato di legare lapatinib in vitro con affinità molto bassa (Kd = 5,5 μM) (Davis et al., 2011), il comportamentosinergico tra lapatinib e NRG si verifica in cellule ad una dose di ~ 50 x inferiore rispetto al Kd in vitro, indicando che qualsiasi legame di lapatinib a HER3 sarebbe probabilmente minore in queste condizioni. Utilizzando un test di spostamento termico (TSA), che misura uno spostamento nella stabilità termica di una proteina dopo il legame ligando / inibitore in vitro, si mostra anche che lapatinib non si lega fortemente HER3 rispetto a ATP e un pannello di altri inibitori(Figura 2a, vedi più avanti).
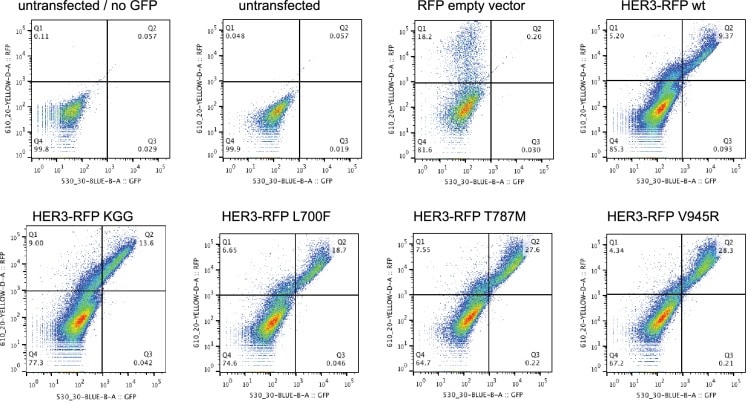
Figura 2-figure supplement 2.I dati numerici e le statistiche relative alla Figura 2.I dati numerici e le statistiche relative alla Figura 2.I dati numerici e le statistiche relative alla Figura 2.Gli effetti dell’occupazione della tasca legata all’ATP di HER3 ATP sulla proliferazione cellulare indotta dalla droga.I dati numerici e le statistiche relative alla Figura 2-figure supplement 1.I dati numerici e le statistiche relative alla Figura 2-figure supplement 1.Cell surface expression of HER3 mutants.a) Saggio di legame TSA in vitro di HER3 con inibitori della chinasi selezionati.(b) Saggio in vitro di legame TSA di HER2 con lapatinib e bosutinib.c) Quantificazione della dimensione degli sferoidi dopo otto giorni di trattamento con una titolazione di bosutinib con micrografie in campo luminoso rappresentative degli sferoidi cellulari SKBR3 dopo otto giorni di trattamento con bosutinib. Le barre di scala indicano 0,5 mm.d) Coinfezione transitoria di cellule MCF7 con HER2wt-GFP e HER3wt-RFP o HER3KGGG-RFP. Le cellule sono state affamate di siero per un’ora, seguite da 10 nM NRG o veicolo per dieci minuti. La fosforilazione di HER3 su Y1289 è stata misurata con Western blot e analizzata con densitometria rispetto al totale di HER3.e) Le cellule SKBR3 sono state trasfettate con vettore vuoto RFP, HER3wt-RFP, HER3T787M-RFP o HER3KGG-RFP. 72 ore di trattamento con bosutinib sono state avviate 24 ore dopo la trasfezione. La proliferazione è stata misurata utilizzando CellTiter-Glo. I dati TSA rappresentati come media ±SEM di (a) due esperimenti indipendenti ciascuno eseguito quadruplicato, o (b) tre esperimenti indipendenti ciascuno eseguito in almeno quadruplicato. Dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito almeno in triplice copia. Dati Western blot rappresentati come media ±SD di tre esperimenti indipendenti. Le quantificazioni del Western blot sono state analizzate da ANOVA a senso unico. ****p≤0.0001 Dati corrispondenti e statistiche disponibili come Figura 2 dati fonte 1.10.7554/eLife.32271.013Cifra dati a 2 fonti 1.Dati numerici e statistiche relative alla Figura 2.a) TSA del dominio chinasi HER3 e una titolazione di bosutinib mostra un ΔTm50di 4,15 ± 1,94 nM.(b) Le cellule SKBR3 sono state trattate con una gamma di concentrazioni di bosutinib per 72 ore e la proliferazione è stata misurata utilizzando CellTiter-Glo(c) CellTiter-Glo quantificazione del punto finale delle colture di sferoidi dalla Figura 2b.(d) Western blot analisi delle colture di sferoidi trattati come in(c).e) Proliferazione 2D di cellule SKBR3 utilizzando una titolazione di bosutinib ±lapatinib (50 nM o 1 μM) per 72 ore.f) le cellule SKBR3 sono state transitoriamente trasfettate con vettore-RFP, HER3wt-RFP o HER3KGG-RFP e trattate con lapatinib ±10 nM NRG per 72 ore.g) analisi CETSA del legame del bosutinib con HER3wt, HER3KGG o HER3T787M. I lisati delle cellule COS7 che esprimono ectopicamente HER3-RFP sono stati trattati con DMSO o 50 nM bosutinib, dopo di che i campioni sono stati divisi e i campioni corrispondenti incubati a 42°C o 50°C. L’analisi Western blot mostra il recupero di HER3 a 50°C rispetto a 42°C. I dati in(a-f) presentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia. I dati Western blot in(d) mostrano un esempio rappresentativo di tre esperimenti indipendenti. I dati in(g) presentati come media ±SD di quattro esperimenti indipendenti. Dati corrispondenti e statistiche disponibili come Figura 2-figure supplement 1-source data 1.10.7554/eLife.32271.011Figure 2-figure supplement 1-source data 1.Numerical data and statistics relating to Figure 2-figure supplement 1.Analisi citometrica a flusso di localizzazione della membrana di tutti i costrutti HER3-RFP utilizzati in questo studio. Le cellule SKBR3 vive sono state colorate con GFP-coniugato anti-HER3 per mostrare la combinazione di HER3 trasfettato ed endogeno sulla membrana. Tutti i costrutti HER3-RFP HER3 mostrano la localizzazione della membrana, come rappresentato dai quadranti in alto a destra. Rappresentativo diagrammi di citometria a flusso da uno dei due esperimenti indipendenti.
Mentre il trattamento EGF salvato cellule SKBR3 dagli effetti del trattamento a bassa concentrazione lapatinib, effetti sinergici di crescita come quelli osservati con lapatinib-NRG co-trattamento non sono stati osservati per lapatinib-EGF co-trattamento SKBR3 o BT474 cellule(Figura 1-figure supplemento 1h-i). Anche se NRG è anche un fattore di crescita ligando per HER4, i livelli proteici di HER4 nelle cellule SKBR3 sono molto bassi(Figura 1-figure supplement 1g). Inoltre, lapatinib è un forte inibitore sia di EGFR che di HER4(Davis et al., 2011). Presi insieme, questi dati sembrano escludere un ruolo significativo per EGFR e HER4 nella crescita sinergica osservata per il co-trattamento di lapatinib-NRG. Inoltre, l’abbattimento transitorio di HER3 con due diversi oligonucleotidi siRNA mostra una modesta, ma coerente riduzione dell’effetto proliferativo del co-trattamento di ligandi inibitori, implicando HER3 come il recettore rilevante del fattore di crescita vincolante per questa risposta NRG(Figura 1-figure supplement 1j).
Gli effetti proliferativi di lapatinib e NRG sulle cellule SKBR3 sono stati osservati anche in colture di sferoidi 3D. Come si è visto nei sistemi di coltura 2D, in coltura sferoide 3D l’aggiunta di NRG alle cellule trattate con lapatinib salva le cellule SKBR3 dalla citotossicità indotta da lapatinib/citostasi(Figura 1c, Figura 1-figure supplement 1k-l). Lapatinib e NRG hanno in comune un effetto cooperativo sull’induzione della proliferazione nelle colture di sferoidi 3D, dove la dimensione degli sferoidi è maggiore per le condizioni di co-trattamento inibitore-ligando che per quelle trattate con il solo fattore di crescita.
La neratinib, inibitore irreversibile, lega la stessa conformazione inattiva della lapatinib e con analoga affinità di legame(Davis et al., 2011). Tuttavia, la neratinib è un inibitore irreversibile e forma un legame covalente con HER2C805, un residuo conservato in EGFR e HER4 ma non HER3. Neratinib-NRG co-trattamento non ha mostrato il fenotipo proliferativo sinergico osservato con lapatinib-NRG, sia in un test di conteggio delle cellule, o nella formazione di sferoidi 3D(Figura 1-figura supplemento 2a-d). Allo stesso modo, l’induzione della fosforilazione HER2 e HER3 osservata nell’analisi western blot degli sferoidi 3D co-trattati con lapatinib-NRG era assente nel co-trattamento di neratinib-NRG(Figura 1-figure supplement 1l, Figura 1-figure supplement 2d). Ciò indica che il fenotipo proliferativo osservato per lapatinib necessita probabilmente di un legame dinamico e reversibile con un inibitore.
Collettivamente, i dati provenienti da entrambe le culture 2D e 3D mostrano che c’è una sinergia controintuitiva tra l’inibitore HER2 lapatinib e il ligando HER3 NRG nel guidare la proliferazione delle cellule SKBR3. Questo ci ha spinto ad esaminare il potenziale per una nuova regolazione allosterica delle interazioni eterotipiche HER2-HER3 da parte di entrambi i ligandi e inibitori.

Figura 1-figure supplemento 2-figure dati fonte 1.I dati numerici e le statistiche relative alla Figura 1. Lapatinib e NRG hanno effetti sinergici sulla crescita di SKBR3 nei sistemi di coltura 2D e 3D.dati numerici e statistiche relative alla Figura 1.Dati numerici e statistiche relative alla Figura 1.Effetti di lapatinib e NRG sulla proliferazione delle cellule tumorali della mammella.dati numerici e statistiche relative alla Figura 1-figure supplement 1.Dati numerici e statistiche relative alla Figura 1 . L’inibitore irreversibile neratinib non mostra una crescita sinergica in condizioni di co-trattamento del legante .(a) CellTiter-Glo proliferation test di cellule SKBR3 dopo il trattamento per 72 ore con una gamma di concentrazioni di lapatinib ± 10 nM NRG.b) Saggio di conteggio delle cellule SKBR3 trattate per 72 ore con DMSO o 250 nM lapatinib ±10 nM NRG, prima della quantificazione del numero di cellule su un contatore Vi-CELL.c) Quantificazione dell’area sferoidale SKBR3 3D dopo 8 giorni di trattamento con una gamma di concentrazioni di lapatinib ± 10 nM NRG, con micrografie rappresentative del campo luminoso. Barre di scala 0,5 mm. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti eseguiti ciascuno in quadruplicato. Dati corrispondenti e statistiche disponibili come Figura 1 – dati fonte 1.10.7554/eLife.32271.008Figure 1-source data 1.Numerical data e statistiche relative alla Figura 1.(a-f) BT474, AU565, HCC1419, ZR75, MCF7, e HCC1569 sono state saggiate per la proliferazione nella risposta alla lapatinib ±10 nM NRG come descritto nella Figura 1a. BT474 e AU565 rappresentano linee sensibili alla lapatinib. HCC1419 e ZR75 rappresentano linee parzialmente sensibili. MCF7 e HCC1569 rappresentano linee non sensibili alla lapatinib.(g) Analisi Western blot dei livelli di proteina della famiglia EGFR endogena in SKBR3, BT474, AU565, HCC1419, ZR75, MCF7 e HCC1569.(h-i) Le celluleSKBR3 e BT474 sono state trattate per 72 ore con una titolazione di lapatinib ±10 nM EGF, dopo di che la proliferazione è stata misurata utilizzando CellTiter-Glo.(j) CellTiter-Glo test di proliferazione CellTiter-Glo di cellule SKBR3 con siRNA transitorio abbattuto di HER3 utilizzando oligonucleotidi singoli. Western blot denota l’efficienza di abbattimento di HER3 si11 e HER3 si13 oligonucleotidi.k) CellTiter-Glo endpoint analisi della proliferazione di colture di sferoidi SKBR3 dopo 8 giorni di lapatinib ±NRG.(l) Analisi Western blot di colture di sferoidi SKBR3 in condizioni corrispondenti alla Figura 1c, Figura 1-figure supplement 1k. Viene mostrato un esempio rappresentativo di tre esperimenti indipendenti. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia, ad eccezione di (j), che rappresenta sei esperimenti indipendenti ciascuno eseguito in triplice copia. Dati corrispondenti e statistiche disponibili come figura 1-figure supplement 1-source data 1.10.7554/eLife.32271.005Figure 1-figure supplement 1-source data 1.Numerical data and statistics relating to Figure 1-figure supplement 1.a) Le cellule SKBR3 sono state trattate per 72 ore con DMSO o 250 nM neratinib ±10 nM NRG, prima della quantificazione del numero di cellule su un contatore Vi-CELL b) Quantificazione dell’area sferoidale dopo 8 giorni di trattamento con una titolazione di neratinib. Micrografie rappresentative del campo luminoso degli sferoidi 3D delle cellule SKBR3. Barre di scala 0,5 mm.c) Analisi CellTiter-Glo endpoint di colture di sferoidi da(b).(d) Analisi Western blot della segnalazione cellulare negli sferoidi SKBR3 dopo 8 giorni di trattamento. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia. Tutti i western blot mostrano un esempio rappresentativo di tre esperimenti indipendenti. I dati corrispondenti e le statistiche disponibili come Figura 1-figure supplemento 2-figure dati fonte 1.10.7554/eLife.32271.007Cifra 1-figure supplement 2 dati fonte 1.Dati numerici e statistiche relative alla Figura 1-figure supplement 1.
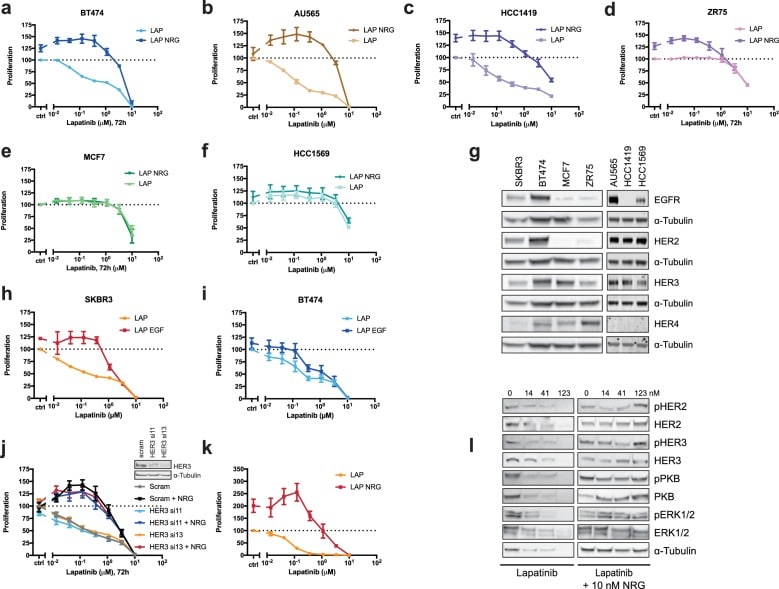
Figura 1-figure supplement 1-source data 1.2. Effetti di lapatinib e NRG sulla proliferazione delle cellule del cancro al seno;dati numerici e statistiche relative alla figura 1-figure supplement 1.Dati numerici e statistiche relative alla figura 1-figure supplement 1.(a-f) Le linee cellulari del tumore al senoBT474, AU565, HCC1419, ZR75, MCF7 e HCC1569 sono state analizzate per la proliferazione nella risposta alla lapatinib ±10 nM NRG come descritto nella Figura 1a. BT474 e AU565 rappresentano linee sensibili alla lapatinib. HCC1419 e ZR75 rappresentano linee parzialmente sensibili. MCF7 e HCC1569 rappresentano linee non sensibili alla lapatinib.(g) Analisi Western blot dei livelli di proteina della famiglia EGFR endogena in SKBR3, BT474, AU565, HCC1419, ZR75, MCF7 e HCC1569.(h-i) Le celluleSKBR3 e BT474 sono state trattate per 72 ore con una titolazione di lapatinib ±10 nM EGF, dopo di che la proliferazione è stata misurata utilizzando CellTiter-Glo.(j) CellTiter-Glo test di proliferazione CellTiter-Glo di cellule SKBR3 con siRNA transitorio abbattuto di HER3 utilizzando oligonucleotidi singoli. Western blot denota l’efficienza di abbattimento di HER3 si11 e HER3 si13 oligonucleotidi.k) CellTiter-Glo endpoint analisi della proliferazione di colture di sferoidi SKBR3 dopo 8 giorni di lapatinib ±NRG.(l) Analisi Western blot di colture di sferoidi SKBR3 in condizioni corrispondenti alla Figura 1c, Figura 1-figure supplement 1k. Viene mostrato un esempio rappresentativo di tre esperimenti indipendenti. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia, ad eccezione di (j), che rappresenta sei esperimenti indipendenti ciascuno eseguito in triplice copia. Dati corrispondenti e statistiche disponibili come figura 1-figure supplement 1-source data 1.10.7554/eLife.32271.005Figure 1-figure supplement 1-source data 1.Numerical data and statistics relating to Figure 1-figure supplement 1.

Figura 1-figure supplement 2 dati fonte 1.I dati numerici e le statistiche relative alla figura 1-figure supplement 1.Numerical data and statistics relating to Figure 1-figure supplement 1.Numerical data and statistics relating to Figure 1-figure supplement 1.(a) Le cellule SKBR3 sono state trattate per 72 ore con DMSO o 250 nM neratinib ±10 nM NRG, prima della quantificazione del numero di cellule su un contatore Vi-CELL (b) Quantificazione dell’area sferoidale dopo 8 giorni di trattamento con una titolazione di neratinib. Micrografie rappresentative del campo luminoso degli sferoidi 3D delle cellule SKBR3. Barre di scala 0,5 mm.c) Analisi CellTiter-Glo endpoint di colture di sferoidi da(b).(d) Analisi Western blot della segnalazione cellulare negli sferoidi SKBR3 dopo 8 giorni di trattamento. Tutti i dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia. Tutti i western blot mostrano un esempio rappresentativo di tre esperimenti indipendenti. I dati corrispondenti e le statistiche disponibili come Figura 1-figure supplemento 2-figure dati fonte 1.10.7554/eLife.32271.007Cifra 1-figure supplement 2 dati fonte 1.Dati numerici e statistiche relative alla Figura 1-figure supplement 1.

Figura 2-figure supplement 2.I dati numerici e le statistiche relative alla Figura 2.I dati numerici e le statistiche relative alla Figura 2.I dati numerici e le statistiche relative alla Figura 2.Gli effetti dell’occupazione della tasca HER3 ATP-binding ATP-binding pocket sulla proliferazione cellulare indotta dalla droga.I dati numerici e le statistiche relative alla Figura 2-figure supplement 1.I dati numerici e le statistiche relative alla Figura 2-figure supplement 1.Cell surface expression of HER3 mutants.a) Saggio di legame TSA in vitro di HER3 con inibitori della chinasi selezionati.(b) Saggio in vitro di legame TSA di HER2 con lapatinib e bosutinib.c) Quantificazione della dimensione degli sferoidi dopo otto giorni di trattamento con una titolazione di bosutinib con micrografie in campo luminoso rappresentative degli sferoidi cellulari SKBR3 dopo otto giorni di trattamento con bosutinib. Le barre di scala indicano 0,5 mm.d) Coinfezione transitoria di cellule MCF7 con HER2wt-GFP e HER3wt-RFP o HER3KGGG-RFP. Le cellule sono state affamate di siero per un’ora, seguite da 10 nM NRG o veicolo per dieci minuti. La fosforilazione di HER3 su Y1289 è stata misurata con Western blot e analizzata con densitometria rispetto al totale di HER3.e) Le cellule SKBR3 sono state trasfettate con vettore vuoto RFP, HER3wt-RFP, HER3T787M-RFP o HER3KGG-RFP. 72 ore di trattamento con bosutinib sono state avviate 24 ore dopo la trasfezione. La proliferazione è stata misurata utilizzando CellTiter-Glo. I dati TSA rappresentati come media ±SEM di (a) due esperimenti indipendenti ciascuno eseguito quadruplicato, o (b) tre esperimenti indipendenti ciascuno eseguito in almeno quadruplicato. Dati di proliferazione rappresentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito almeno in triplice copia. Dati Western blot rappresentati come media ±SD di tre esperimenti indipendenti. Le quantificazioni del Western blot sono state analizzate da ANOVA a senso unico. ****p≤0.0001 Dati corrispondenti e statistiche disponibili come Figura 2 dati fonte 1.10.7554/eLife.32271.013Cifra dati a 2 fonti 1.Dati numerici e statistiche relative alla Figura 2.a) TSA del dominio chinasi HER3 e una titolazione di bosutinib mostra un ΔTm50di 4,15 ± 1,94 nM.(b) Le cellule SKBR3 sono state trattate con una gamma di concentrazioni di bosutinib per 72 ore e la proliferazione è stata misurata utilizzando CellTiter-Glo(c) CellTiter-Glo quantificazione del punto finale delle colture di sferoidi dalla Figura 2b.(d) Western blot analisi delle colture di sferoidi trattati come in(c).e) Proliferazione 2D di cellule SKBR3 utilizzando una titolazione di bosutinib ±lapatinib (50 nM o 1 μM) per 72 ore.f) le cellule SKBR3 sono state transitoriamente trasfettate con vettore-RFP, HER3wt-RFP o HER3KGG-RFP e trattate con lapatinib ±10 nM NRG per 72 ore.g) analisi CETSA del legame del bosutinib con HER3wt, HER3KGG o HER3T787M. I lisati delle cellule COS7 che esprimono ectopicamente HER3-RFP sono stati trattati con DMSO o 50 nM bosutinib, dopo di che i campioni sono stati divisi e i campioni corrispondenti incubati a 42°C o 50°C. L’analisi Western blot mostra il recupero di HER3 a 50°C rispetto a 42°C. I dati in(a-f) presentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia. I dati Western blot in(d) mostrano un esempio rappresentativo di tre esperimenti indipendenti. I dati in(g) presentati come media ±SD di quattro esperimenti indipendenti. Dati corrispondenti e statistiche disponibili come Figura 2-figure supplement 1-source data 1.10.7554/eLife.32271.011Figure 2-figure supplement 1-source data 1.Numerical data and statistics relating to Figure 2-figure supplement 1.Analisi citometrica a flusso di localizzazione della membrana di tutti i costrutti HER3-RFP utilizzati in questo studio. Le cellule SKBR3 vive sono state colorate con GFP-coniugato anti-HER3 per mostrare la combinazione di HER3 trasfettato ed endogeno sulla membrana. Tutti i costrutti HER3-RFP HER3 mostrano la localizzazione della membrana, come rappresentato dai quadranti in alto a destra. Rappresentativo diagrammi di citometria a flusso da uno dei due esperimenti indipendenti.
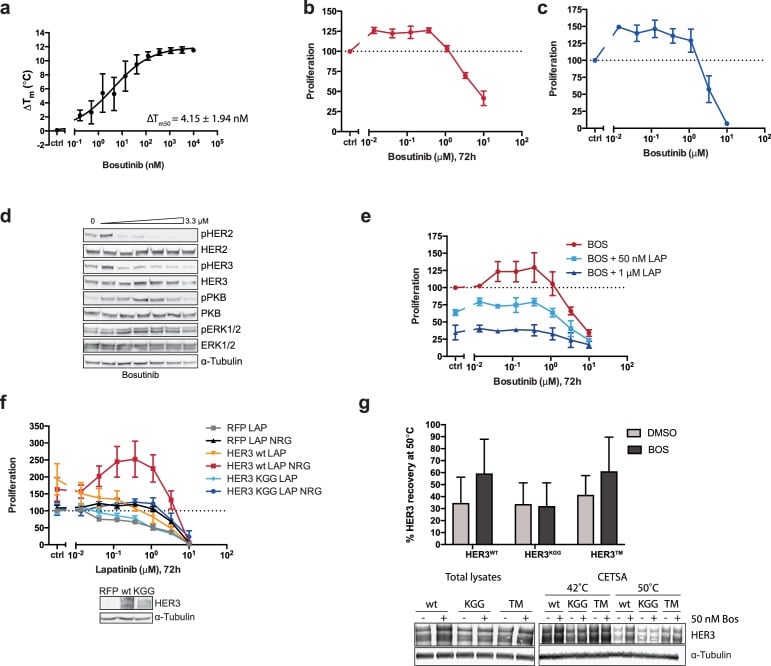
Figura 2-figure supplemento 1-figure dati fonte 1.Gli effetti di HER3 ATP-binding occupazione tasca ATP-binding su droga-proliferazione cellulare indotta da droga.dati numerici e le statistiche relative alla figura 2-figure supplemento 1.Numerical dati e statistiche relative alla figura 2-figure supplemento 1.(a) Il TSA del dominio chinasi HER3 e una titolazione di bosutinib mostra un ΔTm50di 4,15 ± 1,94 nM.(b) Le cellule SKBR3 sono state trattate con una gamma di concentrazioni di bosutinib per 72 ore e la proliferazione è stata misurata utilizzando CellTiter-Glo(c) CellTiter-Glo quantificazione del punto finale delle colture di sferoidi dalla Figura 2b.(d) Western blot analisi delle colture di sferoidi trattati come in(c).e) Proliferazione 2D di cellule SKBR3 utilizzando una titolazione di bosutinib ±lapatinib (50 nM o 1 μM) per 72 ore.f) le cellule SKBR3 sono state transitoriamente trasfettate con vettore-RFP, HER3wt-RFP o HER3KGG-RFP e trattate con lapatinib ±10 nM NRG per 72 ore.g) analisi CETSA del legame del bosutinib con HER3wt, HER3KGG o HER3T787M. I lisati delle cellule COS7 che esprimono ectopicamente HER3-RFP sono stati trattati con DMSO o 50 nM bosutinib, dopo di che i campioni sono stati divisi e i campioni corrispondenti incubati a 42°C o 50°C. L’analisi Western blot mostra il recupero di HER3 a 50°C rispetto a 42°C. I dati in(a-f) presentati come media ±SEM di tre esperimenti indipendenti ciascuno eseguito in triplice copia. I dati Western blot in(d) mostrano un esempio rappresentativo di tre esperimenti indipendenti. I dati in(g) presentati come media ±SD di quattro esperimenti indipendenti. Dati corrispondenti e statistiche disponibili come Figura 2-figure supplement 1-source data 1.10.7554/eLife.32271.011Figure 2-figure supplement 1-source data 1.Numerical data and statistics relating to Figure 2-figure supplement 1.

Figura 2-figure supplement 2.Espressione della superficie cellulare dei mutanti HER3.3. Analisi citometrica a flusso di localizzazione della membrana di tutti i costrutti HER3-RFP utilizzati in questo studio. Live SKBR3 cellule SKBR3 sono state colorate con GFP-coniugato anti-HER3 per mostrare la combinazione di HER3 trasfettato ed endogeno sulla membrana. Tutti i costrutti HER3-RFP HER3 mostrano la localizzazione della membrana, come rappresentato dai quadranti in alto a destra. Rappresentativo diagrammi di citometria a flusso da uno dei due esperimenti indipendenti.
HER3 occupazione tasca nucleotide HER3 è di importanza strutturale
Per studiare gli effetti del legame ATP sulla funzione HER3, abbiamo cercato di stabilizzare e destabilizzare la pseudochinasi nucleotide-binding pocket. Questo ci permetterebbe di indagare l’importanza del ruolo strutturale che l’occupazione della tasca di legame nucleotidico ha dimostrato di svolgere in diverse (pseudo)chinasi.
Per separare il ruolo strutturale e tracciare i ruoli catalitici che l’ATP-binding potrebbe svolgere in HER3, abbiamo usato il bosutinib, inibitore Src/Abl-competitivo dell’ATP, che ha dimostrato di legarsi fortemente a HER3 ma non ad altri membri della famiglia EGFR(Levinson e Boxer, 2014; Davis et al., 2011). Abbiamo confrontato il bosutinib con un piccolo pannello di inibitori della famiglia EGFR e con un ulteriore inibitore Src, il dasatinib, e misurato la stabilità termica di HER3 da parte del TSA(Figura 2a, Figura 2-figure supplement 1a). In linea con le osservazioni precedenti, abbiamo confermato che HER3 lega fortemente il bosutinib. Significativamente, lapatinib non è stato in grado di fornire un notevole spostamento termico, che corrisponde ai risultati pubblicati in precedenza che indicano che HER3 non lega lapatinib con alta affinità(Davis et al., 2011). Mentre lapatinib è stato in grado di conferire una maggiore stabilità termica a HER2, bosutinib non lo è stato(Figura 2b). Questo è in linea con i dati pubblicati in precedenza che indicano che HER2 non è un forte legante di bosutinib(Davis et al., 2011).
Abbiamo ipotizzato che bosutinib potrebbe essere in grado di aiutare la proliferazione in un contesto cellulare stabilizzando la tasca di legame nucleotidico di HER3 e aiutando a sostenere la formazione di dimeri, analogamente al comportamento legato al vemurafenib di BRAF. In un test di proliferazione 2D, le cellule SKBR3 trattate con bosutinib oltre 72 ore mostrano un’induzione dose dipendente della proliferazione senza stimolazione aggiuntiva NRG(Figura 2-figure supplement 1b). Questo effetto proliferativo è sostenuto in trattamenti di otto giorni in colture sferoidali 3D(Figura 2c, Figura 2-figure supplement 1d,e). La capacità del bosutinib di indurre la proliferazione delle cellule SKBR3 sembra essere un evento mediato dalla famiglia EGFR, in quanto il trattamento con lapatinib può ridurre i suoi effetti in modo dose-dipendente(Figura 2-figure supplement 1e).
Al fine di destabilizzare la tasca di legame del nucleotide HER3 abbiamo realizzato il triplo mutante HER3KGG. HER3K742 è stato mutato in metionina per ostacolare il coordinamento dell’ATP α-fosfato, che da solo ha dimostrato di ridurre l’affinità del legame mant-ATP di HER3 (Shiet al., 2010). Per ostacolare ulteriormente il legame con l’ATP, sono stati introdotti dei doppi aspartato nell’anello ricco di glicina (HER3G716D/G718D) per imitare il residuo di aspartato pseudochinase-specifico osservato nell’anello ricco di glicina del VRK3(Scheeff et al., 2009), aggiungendo una carica negativa nell’area in cui i fosfati dell’ATP si troverebbero normalmente. L’introduzione di questo ATP-binding HER3KGG mutante carente HER3KGG in cellule MCF7 mostra l’abrogazione della trans-fosforilazione indotta da ligandi di HER3 da HER2(Figura 2d). SKBR3 cellule SKBR3 ectopicamente esprimendo HER3wt o HER3KGG mostrano un comportamento differenziale proliferativo su lapatinib ± NRG trattamento. Questo indica un ruolo critico per HER3 ATP vincolante al fine di sostenere inibitore di crescita fattore di proliferazione cooperativa(Figura 2-figure supplemento 1f).
Il legame bosutinib di HER3wt, HER3KGG, e il farmaco proposto de-sensibilizzato HER3T787M(Levinson e Boxer, 2014; Dong et al., 2017), è stato studiato utilizzando un test di spostamento termico in cella (CETSA) (Jafari etal., 2014; Reinhard etal., 2015). Dove il tipo selvatico HER3 ha mostrato una maggiore stabilità termica nelle cellule in presenza di 50 nM bosutinib, HER3KGG non lo ha fatto(Figura 2-figure supplement 1g). L’espressione ectopica del tipo selvatico HER3, ma non HER3KGGG o HER3T787M, migliora la proliferazione mediata dal bosutinib, indicando che questo comportamento è guidato dal legame del bosutinib direttamente a HER3(Figura 2e). Sia HER3KGGG che HER3T787M hanno mostrato una localizzazione normale alla membrana plasmatica, come misurato dalla citometria a flusso, indicando che queste mutazioni non hanno compromesso il recettore e il suo traffico verso la membrana plasmatica(Figura 2-figure supplement 2).
I risultati di HER3KGGG e bosutinib indicano che l’occupazione delle tasche nucleotidiche in HER3 è essenziale per la sua capacità di sostenere un percorso di segnalazione proliferativa in circostanze distinte: nella risposta acuta al fattore di crescita, nella promozione della proliferazione cooperativa di ligandi inibitori e anche dopo il trattamento con un inibitore legante HER3. Ciò indica un ruolo strutturale critico per l’occupazione tascabile di HER3 ATP-binding ATP nella sua capacità di sostenere le eterointerazioni e la proliferazione. Considerando gli effetti proliferativi osservati con l’inibitore HER3-binding bosutinib, i nostri risultati suggeriscono anche che qualsiasi attività residua di transferasi che HER3 mantiene non sembra essere importante in queste risposte in vivo, a meno che non invochiamo un meccanismo di azione hit-and-run per il bosutinib su HER3 che sembrerebbe improbabile.
Il legame di lapatinib induce l’eterodimerizzazione di HER2-HER3
La stabilità conferita ad una proteina chinasi da un legame inibitore di piccole molecole ha dimostrato di svolgere un ruolo importante nella promozione delle interazioni proteina-proteina. Abbiamo studiato il ruolo potenziale di lapatinib per promuovere in modo simile l’eterodimerizzazione di HER2-HER3 stabilizzando particolari conformazioni proteiche in HER2 con un approccio FRET-FLIM. Abbiamo misurato l’eterodimerizzazione indotta da farmaci di HER2 e HER3, come abbiamo fatto in precedenza nel caso della dimerizzazione indotta da farmaci del recettore EGF(Bublil et al., 2010; Coban et al., 2015).
A livelli di proteine endogene nelle cellule SKBR3, osserviamo l’eterodimerizzazione HER2-HER3 guidata da lapatinib a livelli simili a quelli osservati con NRG(Figura 3a). È interessante notare che la dimerizzazione indotta da lapatinib si verifica in assenza di NRG aggiunto esogeneamente, indicando un dimero HER2-HER3 dimero che è guidato principalmente attraverso interazioni intracellulari dominio. Le cellule MCF7, che esprimono bassi livelli di HER2 e HER3 endogeno rispetto a SKBR3, mostrano anche l’eterodimerizzazione indotta da lapatinib di GFP-HER2wt e HA-HER3wt ectopicamente espressi(Figura 3b).
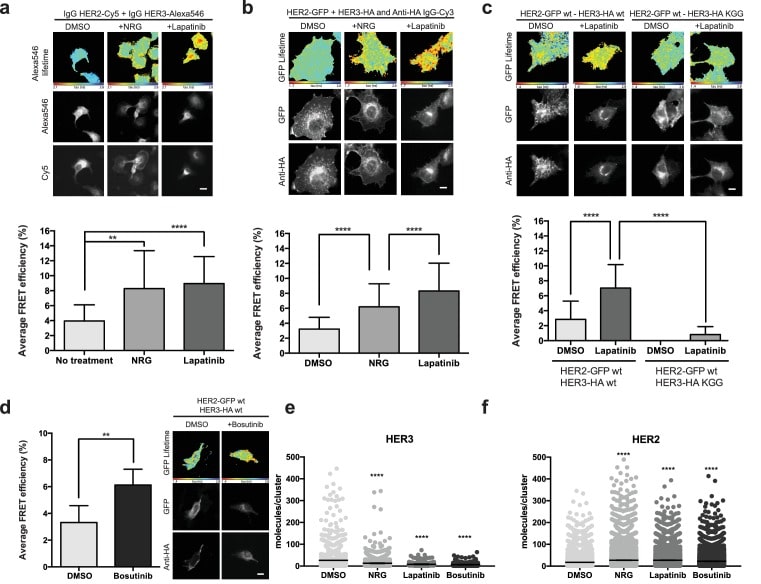
Figura 3 – Dati sorgente 1.Figura 3—dati fonte 1. Interazioni eterotipiche indotte da inibitore HER2-HER3.Dati numerici e statistiche relative alla Figura 3.Dati numerici e statistiche relative alla Figura 3.a) FRET-FLIM analisi di associazione endogena HER2-HER3 in cellule SKBR3, siero affamato per 1 ora, e stimolato con 6,7 nM NRG per 15 min, o inibito con lapatinib (10 µM) per 1 ora, prima della fissazione e la colorazione con IgG anti-HER2-Cy5 e IgG anti-HER3-Alexa546 durante la notte, a 4 ° C.b) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP e HER3wt-HA. Le cellule sono state incubate come in(a) e colorate con anticorpo anti-HA coniugato con Alexa-546 (controlli trattati con veicolo).c) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP e HER3wt-HA o HER3KGGG-HA. Celle trattate con lapatinib (10 µM) per 1 ora, prima della fissazione e della colorazione con anticorpo anti-HA coniugato con Alexa-546.d) Le cellule SKBR3 sono state trattate con bosutinib (50 nM, 1 ora) e colorate come in(b).(e, f) Misurazioni di molecole / cluster da dati STORM presi da dati STORM di cellule SKBR3 etichettate con HER2Affibody-Alexa488 e HER3Affibody-Alexa647 o NRG-Alexa647 ±14 nM lapatinib o 41 nM bosutinib. Gli istogrammi cumulativi FRET-FLIM mostrano l’efficienza media FRET di tre esperimenti indipendenti. **p≤0,01; ****p≤0,0001 Barre di scala 5 μm. I dati di clustering rappresentano la combinazione media di due esperimenti indipendenti con ciascuno di essi che misura >1000 cluster. I dati di clustering sono presentati come media con il 95% di CI. Dati corrispondenti e statistiche disponibili come Figura 3 – dati sorgente 1.10.7554/eLife.32271.015Figure 3-source data 1.Numerical data and statistics relating to Figure 3 .
Come discusso in precedenza, l’occupazione della tasca di legame del nucleotide in HER3 è importante per la sua capacità di sostenere la proliferazione. Ciò si riflette anche nel caso della formazione di eterodimeri indotta da lapatinib, dove l’introduzione della tasca nucleotidica ha compromesso il mutante HER3KGG di HER3 perturba fortemente l’eterodimerizzazione promossa dagli inibitori(Figura 3c). In linea con gli effetti proliferativi sopra descritti, bosutinib è stato anche in grado di promuovere direttamente l’eterodimerizzazione tra HER2 e HER3(Figura 3d).
Utilizzando la microscopia di ricostruzione ottica stocastica (STORM), abbiamo analizzato il clustering dei recettori nelle cellule SKBR3. Il trattamento con NRG, lapatinib, o bosutinib ha mostrato uno spostamento nella dimensione della popolazione di cluster rispetto al controllo, che implica la formazione di oligomeri di ordine superiore, piuttosto che dimeri(Figura 3e,f). L’esatta stechiometria HER2-HER3 in questi oligomeri trattati con farmaci rimane elusiva, perché queste condizioni sperimentali ci hanno permesso di contare solo la dimensione dei cluster per HER2 o HER3, non entrambi allo stesso tempo. Pertanto, ci si aspetta che i cluster HER3 osservati contengano anche recettori HER2 non conteggiati, e viceversa, come risulta evidente dai dati FRET-FLIM.

Figura 3 – Dati sorgente 1.Figura 3—dati fonte 1. Interazioni eterotipiche HER2-HER3 indotte da inibitori.dati numerici e statistiche relative alla Figura 3.Dati numerici e statistiche relative alla Figura 3.a) FRET-FLIM analisi di associazione endogena HER2-HER3 in cellule SKBR3, siero affamato per 1 ora, e stimolato con 6,7 nM NRG per 15 min, o inibito con lapatinib (10 µM) per 1 ora, prima della fissazione e la colorazione con IgG anti-HER2-Cy5 e IgG anti-HER3-Alexa546 durante la notte, a 4 ° C.b) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP e HER3wt-HA. Le cellule sono state incubate come in(a) e colorate con anticorpo anti-HA coniugato con Alexa-546 (controlli trattati con veicolo).c) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP e HER3wt-HA o HER3KGGG-HA. Celle trattate con lapatinib (10 µM) per 1 ora, prima della fissazione e della colorazione con anticorpo anti-HA coniugato con Alexa-546.d) Le cellule SKBR3 sono state trattate con bosutinib (50 nM, 1 ora) e colorate come in(b).(e, f) Misurazioni di molecole / cluster da dati STORM presi da dati STORM di cellule SKBR3 etichettate con HER2Affibody-Alexa488 e HER3Affibody-Alexa647 o NRG-Alexa647 ±14 nM lapatinib o 41 nM bosutinib. Gli istogrammi cumulativi FRET-FLIM mostrano l’efficienza media FRET di tre esperimenti indipendenti. **p≤0,01; ****p≤0,0001 Barre di scala 5 μm. I dati di clustering rappresentano la combinazione media di due esperimenti indipendenti con ciascuno di essi che misura >1000 cluster. I dati di clustering sono presentati come media con il 95% di CI. Dati corrispondenti e statistiche disponibili come Figura 3 – dati sorgente 1.10.7554/eLife.32271.015Figure 3-source data 1.Numerical data and statistics relating to Figure 3 .
Interruzione dell’interfaccia attiva HER2-HER3
Il dimero di segnalazione attiva nella famiglia EGFR adotta un orientamento asimmetrico, in cui c’è una distinta divisione del lavoro nell’accoppiamento attivatore-ricevitore. Una chinasi (la chinasi dell’attivatore) non è un substrato fosforilato, ma si lega in modo da aiutare il suo partner di eterodimerizzazione (la chinasi del ricevitore) ad adottare una conformazione attiva. La chinasi ricevente è quindi in grado di fosforilare il substrato. Originariamente descritta per l’omogeneizzazione EGFR, e simile alla modalità di legame ciclina/CDK(Jeffrey et al., 1995; Zhang et al., 2006), questa interfaccia di dimerizzazione attiva canonica è stata riportata in tutta la famiglia EGFR, compresa l’eterodimerizzazione di HER3, che può svolgere solo il ruolo di attivatore(Jura et al., 2009b; Littlefield et al., 2014; van Lengerich et al., 2017). Le mutazioni che interrompono questa interfaccia attiva sia nell’attivatore che nelle chinasi del partner ricevente sono ben documentate e sono schematicamente evidenziate(Figura 4a, Figura 4-video 1).
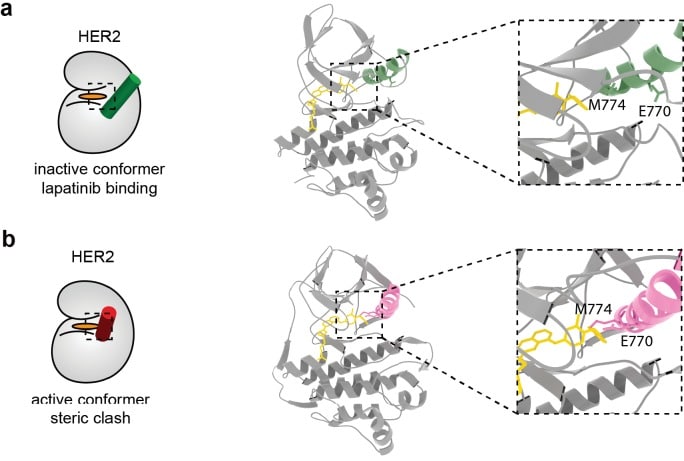
Figura 4-video 1.I dati numerici e le statistiche relative al file della struttura di Figura 4.PDB del modello di interazione molecolare in Figura 4a.I dati numerici e le statistiche relative al file della struttura di Figura 4.PDB del modello di interazione molecolare in Figura 4a.Il modello del legame di lapatinib in HER2-HER3 in conformazioni attive e inattive mostra un potenziale scontro sterico.File di struttura PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.Interface view of the molecular model of an active HER2-HER3 heterodimer, con HER2I714 e HER3V945 evidenziati. a) Rappresentazione schematica e modello molecolare del modello molecolare di HER2-HER3 attivo, orientamento del dimero a dominio asimmetrico della chinasi. Inset denota l’interfaccia di interazione.(b) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP o HER2I714Q-GFP e HER3wt-HA. Le cellule sono state trattate come descritto in Figura 3 e HER2-HER3 associazione è stata misurata da FRET-FLIM.c) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2-GFP e HER3wt-RFP o HER3V945R-RFP. Le cellule sono state incubate come descritto sopra, e trattati con DMSO, lapatinib o NRG prima della fissazione. I dati rappresentano la media ±SEM. *p≤0,05; **p≤0,01, ***p≤0,001; ****p≤0,0001 da ANOVA a senso unico. Le barre di scala rappresentano 5 μm. I dati corrispondenti e le statistiche disponibili come Figura 4 dati fonte 1. Modello molecolare per l’interazione in (a) disponibile come Figura 4—dati sorgente 2.10.7554/eLife.32271.020Cifra dati a 4 fonti 1.Dati numerici e statistiche relative alla Figura 4.10.7554/eLife.32271.021Figure 4 dati fonte 2.PDB file di struttura del modello di interazione molecolare in Figura 4a.a) Lapatinib docking in HER2, con il sito attivo HER2 visualizzato nel riquadro. Lapatinib lega la conformazione inattiva di HER2 dove l’elica α-C è in posizione ‘fuori’. E770 e M774 sull’elica di HER2 α-C evidenziata.b) Lapatinib agganciato alla conformazione attiva di HER2. L’elica α-C è in posizione “in”, causando uno scontro sterico tra E770/M774 e lapatinib. Modelli molecolari per l’aggancio dell’inibitore in (a) e (b) disponibili come Figura 4-figure supplement 1-source datas 1 e 2. 10.7554/eLife.32271.018Figure 4-figure supplement 1-figure source data 1-source file di struttura 1.PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.10.7554/eLife.32271.019Figure 4-figure supplement 1-source data 1-figure file di struttura 2.PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.
Nel caso dell’interfaccia attiva, attivatore/ricevitore, HER3 rinforza l’orientamento verso l’interno dell’elica HER2 α-C, non lasciando spazio all’elica HER2 α-C per adottare la caratteristica di orientamento ‘fuori’ della conformazione inattiva. Abbiamo modellato i potenziali effetti del posizionamento dell’elica HER2 α-C sul legame di lapatinib per verificare se l’orientamento canonico dell’attivatore/ricevitore (in cui l’elica HER2 α-C è spinta verso l’interno) darebbe spazio sufficiente per accogliere ancora lapatinib. La nostra modellazione ha mostrato che, per un’elica HER2 α-C in posizione attiva, ‘in’, il legame di lapatinib risulta in un potenziale scontro sterico con HER2E770/M774 (Figura 4-figuresupplement 1a,b). Una diminuzione generale del volume della tasca di legame nucleotide da 756 Å 3 a232 Å 3 (calcolato con SURFNET v1.5 (Laskowski, 1995)) supporta queste previsioni.
Per verificare ulteriormente se l’HER2-HER3 indotto da lapatinib sta adottando l’orientamento canonico dell’attivatore/ricevitore, abbiamo usato FRET-FLIM per studiare la formazione di dimeri indotta da lapatinib. La mutazione I714Q in HER2, che rende il ricevitore ricevitore-imparo del recettore, ha interrotto l’associazione HER2-HER3 guidata da lapatinib, indicando che è mantenuta nell’interfaccia del dimero indotto da lapatinib(Figura 4b). Tuttavia, la mutazione reciproca attivatore-malato in HER3 (HER3V945R) non ha perturbato l’eterodimerizzazione mediata da lapatinib, anche se ha efficacemente soppresso il dimero attivo canonico dopo l’eterodimerizzazione indotta da ligandi(Figura 4c).
Si suppone che il legame dell’inibitore sia in grado di indurre in modo robusto un eterodimero tra HER2 e HER3, che è distinto dall’eterodimero attivo canonico indotto dopo la stimolazione del fattore di crescita. L’orientamento di questo eterodimero non canonico guidato da lapatinib mantiene HER2I714 nell’interfaccia del dimero, dandoci un punto di partenza per la modellazione molecolare in silico per indagare le potenziali conformazioni del dimero distinte dal dimero attivo ben descritto.

Figura 4-video 1.I dati numerici e le statistiche relative al file della struttura di Figura 4.PDB del modello di interazione molecolare in Figura 4a.I dati numerici e le statistiche relative al file della struttura di Figura 4.PDB del modello di interazione molecolare in Figura 4a.Il modello del legame di lapatinib nelle conformazioni attive e inattive di HER2-HER3 mostra un potenziale scontro sterico.File di struttura PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.Interface view of the molecular model of an active HER2-HER3 heterodimer, con HER2I714 e HER3V945 evidenziati. a) Rappresentazione schematica e modello molecolare del modello molecolare di HER2-HER3 attivo, orientamento del dimero a dominio asimmetrico della chinasi. Inset denota l’interfaccia di interazione.(b) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP o HER2I714Q-GFP e HER3wt-HA. Le cellule sono state trattate come descritto in Figura 3 e HER2-HER3 associazione è stata misurata da FRET-FLIM.c) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2-GFP e HER3wt-RFP o HER3V945R-RFP. Le cellule sono state incubate come descritto sopra, e trattati con DMSO, lapatinib o NRG prima della fissazione. I dati rappresentano la media ±SEM. *p≤0,05; **p≤0,01, ***p≤0,001; ****p≤0,0001 da ANOVA a senso unico. Le barre di scala rappresentano 5 μm. I dati corrispondenti e le statistiche disponibili come Figura 4 dati fonte 1. Modello molecolare per l’interazione in (a) disponibile come Figura 4—dati sorgente 2.10.7554/eLife.32271.020Cifra dati a 4 fonti 1.Dati numerici e statistiche relative alla Figura 4.10.7554/eLife.32271.021Figure 4 dati fonte 2.PDB file di struttura del modello di interazione molecolare in Figura 4a.a) Lapatinib docking in HER2, con il sito attivo HER2 visualizzato nel riquadro. Lapatinib lega la conformazione inattiva di HER2 dove l’elica α-C è in posizione ‘fuori’. E770 e M774 sull’elica di HER2 α-C evidenziata.b) Lapatinib agganciato alla conformazione attiva di HER2. L’elica α-C è in posizione “in”, causando uno scontro sterico tra E770/M774 e lapatinib. Modelli molecolari per l’aggancio dell’inibitore in (a) e (b) disponibili come Figura 4-figure supplement 1-source datas 1 e 2. 10.7554/eLife.32271.018Figure 4-figure supplement 1-figure source data 1-source file di struttura 1.PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.10.7554/eLife.32271.019Figure 4-figure supplement 1-source data 1-figure file di struttura 2.PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.

Figura 4-figure supplement 1-source data 2.Il modello di legame di lapatinib in HER2 inattivo e attivo mostra un potenziale file di struttura sterica clash.PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.PDB file di struttura del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.a) Lapatinib docking in HER2, con il sito attivo HER2 visualizzato nel riquadro. Lapatinib lega la conformazione inattiva di HER2 dove l’elica α-C è in posizione ‘fuori’. E770 e M774 sull’elica di HER2 α-C evidenziata.b) Lapatinib agganciato alla conformazione attiva di HER2. L’elica α-C è in posizione “in”, causando uno scontro sterico tra E770/M774 e lapatinib. Modelli molecolari per l’aggancio dell’inibitore in (a) e (b) disponibili come Figura 4-figure supplement 1-source datas 1 e 2. 10.7554/eLife.32271.018Figure 4-figure supplement 1-figure source data 1-source file di struttura 1.PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1a.10.7554/eLife.32271.019Figure 4-figure supplement 1-source data 1-figure file di struttura 2.PDB del modello di aggancio dell’inibitore in Figura 4-figure supplement 1b.

Figura 4-video 1.2. Vista dell’interfaccia del modello molecolare di un eterodimero attivo HER2-HER3, con HER2I714 e HER3V945 evidenziati.
Lapatinib guida una nuova interfaccia di eterodimerizzazione HER2-HER3
Nel caso degli inibitori della chinasi di tipo II come la lapatinib, l’inibitore stabilizza una conformazione inattiva del dominio chinasi, dove l’elica α-C è inclinata verso l’esterno. Poiché HER3 manca del residuo di glutammato conservato nell’elica α-C, HER3K742 nonè in grado di formare il ponte di sale normalmente osservato nelle strutture del dominio chinasi attivo (Husee Kuriyan, 2002). La conformazione HER3 legata all’ATP non mostra quindi una conformazione attiva classica con l’elica α-C inclinata verso l’interno (Juraet al., 2009b; Shi et al., 2010), ma assomiglia invece alla conformazione inattiva osservata nelle chinasi legate ad inibitori di tipo II come lapatinib. Poiché l’HER2 legato alla lapatinib e l’HER3 legato all’ATP adottano conformazioni simili, c’è la possibilità che il dimero inattivo indotto dalla lapatinib sia orientato simmetricamente.
Nei reticoli cristallini dei domini chinasi EGFR e HER3 sono state osservate due diverse interfacce di interazione simmetrica(Jura et al., 2009a; Jura et al., 2009b). Abbiamo usato la modellazione molecolare per studiare il potenziale per HER3 e lapatinib-bound HER2 per adottare uno di questi conformatori(Figura 5a-b, Figura 5-figure supplement 1a-b). HER2I714 è presente nell’interfaccia di interazione sia dell’orientamento sfalsato, simile all’EGFR, sia dell’orientamento testa a testa, simile all’HER3. Questo è in linea con i dati FRET-FLIM della Figura 4 che suggerisce la presenza del residuo di HER2I714 nell’interfaccia del dimero indotto da lapatinib.
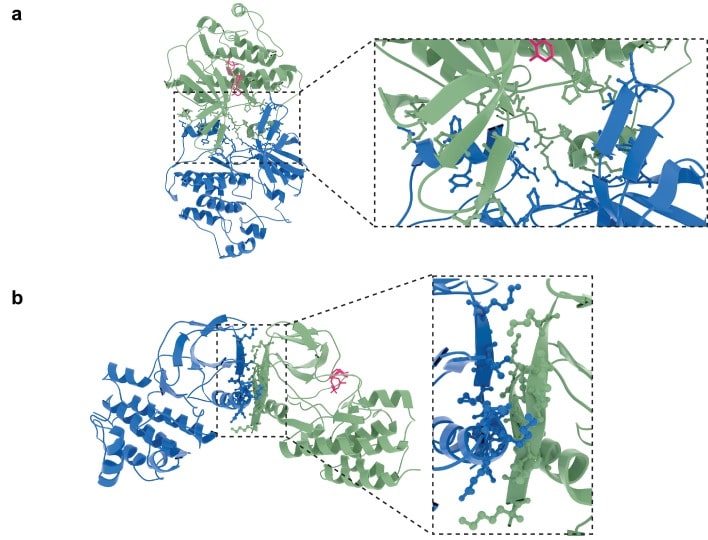
Figura 5-video 2.I dati numerici e le statistiche relative al file della struttura di Figura 5.PDB del modello di interazione molecolare in Figura 5a.PDB del modello di interazione molecolare in Figura 5b.Tabella con i residui di interfaccia modellati, inclusa l’area superficiale accessibile al solvente per residuo in Å 2.I dati numerici e le statistiche relative al file della struttura di Figura 5.PDB del modello di interazione molecolare in Figura 5a. PDB del modello di interazione molecolare in Figura 5b.Tabella con i residui di interfaccia modellati, compresa l’area superficiale accessibile al solvente per residuo in Å 2.Modelli molecolari di potenziali orientamenti del dimero HER2-HER3 indotto da lapatinib.vista di interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione simile all’EGFR, con HER2I714e HER3V945 evidenziati, così come i residui di interfaccia specifici del modello HER2I748/V750.Evidenziazione dell’interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione simile a HER3, con HER2I714 e HER3V945 evidenziati, così come residui di interfaccia specifici del modello HER2N764/K765.a) Lapatinib-bound HER2 (blu, lapatinib in giallo) e ATP-bound analogico HER3 (verde, AMP-PNP in rosa) sono stati modellati in un orientamento simmetrico del dimero simile a EGFR(Jura et al., 2009a). Inset evidenzia l’interfaccia di interazione. La rappresentazione schematica mostra i residui attivi dell’interfaccia del dimero HER2I714 e HER3V945, così come i due residui in HER2 unici per questa interfaccia per un’ulteriore analisi mutazionale.b) HER2 legato con Lapatinib nell’orientamento simmetrico testa a testa del dimero simile a HER3(Jura et al., 2009b). I residui specifici del dimero sono evidenziati nello schema.(c) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP, HER2N764R/K765F-GFP o HER2I748R/V750R-GFP e HER3wt-HA. Le cellule sono state incubate per 24 ore e inibite con 10 µM lapatinib per 1 ora, prima della fissazione e della colorazione con anticorpo anti-HA coniugato con Alexa-546. I dati rappresentati come media ±SEM. ****p≤0,0001, come analizzato da ANOVA unidirezionale. Barre di scala 5 μm dati corrispondenti e statistiche disponibili come Figura 5 – dati fonte 1. Modello molecolare per le interazioni in(a) e(b) disponibile come Figura 5 – dati sorgente 2 e 3. 2. I residui che segnano l’interfaccia del dimero dell’eterodimero HER2-HER3 indotto da lapatinib, nelle conformazioni modellate come EGFR o come HER3, compresa l’area superficiale accessibile al solvente per residuo (in Å 2),sono disponibili come dati fonte della Figura 5 4.10.7554/eLife.32271.025Figure 5-source data 1.Numerical data and statistics relating to Figure 5.10.7554/eLife.32271.026Figure 5-figure 5-source data file di struttura 2.PDB del modello di interazione molecolare in Figura 5a.10.7554/eLife.32271.027Figure 5-figure 5-source data 3.PDB structure file del modello di interazione molecolare in Figura 5b.10.7554/eLife.32271.028Figure 5-source data 4.Table con i residui di interfaccia modellati, compreso il per-residue solvent-area superficiale accessibile in Å 2.2. Modello molecolare del dimero HER2-HER3 indotto da lapatinib nel(a) EGFR-like e(b) orientamento HER3-like con residui di interfaccia mostrati come bastoncini.
Sulla base di questi modelli, abbiamo progettato coppie di mutazioni in HER2 che perturberebbero esclusivamente uno dei potenziali orientamenti dell’eterodimero(Figura 5-figure supplement 1, Figura 5-video 1 e 2). Per il dimero sfalsato di tipo EGFR abbiamo sostituito due residui idrofobici su HER2 con due residui con carica positiva, HER2I748R/V750R, che dovrebbero portare alla repulsione dei residui con carica positiva, K998 e K999, che si trovano sul lato HER3 dell’interfaccia.
Allo stesso modo, per il dimeratore simile a HER3, testa a testa, abbiamo previsto che il mutante HER2N764R/K765F avrebbe interrotto l’interfaccia di dimerizzazione. La sostituzione di un residuo di asparagina (HER2N764) con un’arginina caricata positivamente dovrebbe portare alla repulsione di un residuo di HER3 caricato positivamente (HER3R702), che si trova entro un raggio di 4 Å e di fronte a HER2N764, causando quindi una grave perturbazione dell’interfaccia del dimero simile a HER3. Inoltre, la sostituzione di un residuo di lisina (HER2K765) con un residuo ingombrante e idrofobico come la fenilalanina dovrebbe generare scontri su questa interfaccia HER2-HER3.
Questi mutanti di interfaccia dimero sono stati introdotti nel nostro saggio FRET-FLIM per l’indagine del conformer di eterodimerizzazione indotto da lapatinib(Figura 5c). Il mutante HER2N764R/K765F ha interrotto l’eterodimerizzazione perturbata al legame della lapatinib, mentre HER2I748R/V750R non ha mostrato alcuna differenza nella formazione dell’eterodimero. Questi dati mutazionali FRET/FLIM sono coerenti con il nostro modello secondo cui l’eterodimero HER2-HER3 indotto dalla lapatinib adotta un orientamento simmetrico, testa a testa, simile a quello osservato nel reticolo cristallino del dominio chinasi HER3 (Juraet al., 2009b) (Figura 5b).

Figura 5-video 2.I dati numerici e le statistiche relative al file della struttura di Figura 5.PDB del modello di interazione molecolare in Figura 5a.PDB del modello di interazione molecolare in Figura 5b.Tabella con i residui di interfaccia modellati, compreso l’area superficiale accessibile al solvente per residuo in Å 2.I dati numerici e le statistiche relative al file della struttura di Figura 5.PDB del modello di interazione molecolare in Figura 5a. PDB del modello di interazione molecolare in Figura 5b .Tabella con i residui di interfaccia modellati, compresa l’area superficiale accessibile al solvente per residuo in Å 2.Modelli molecolari di potenziali orientamenti del dimero HER2-HER3 indotto da lapatinib.vista di interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione simile all’EGFR, con HER2I714e HER3V945 evidenziati, così come i residui di interfaccia specifici del modello HER2I748/V750.Evidenziazione dell’interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione simile a HER3, con HER2I714 e HER3V945 evidenziati, così come residui di interfaccia specifici del modello HER2N764/K765.a) Lapatinib-bound HER2 (blu, lapatinib in giallo) e ATP-bound analogico HER3 (verde, AMP-PNP in rosa) sono stati modellati in un orientamento simmetrico del dimero simile a EGFR(Jura et al., 2009a). Inset evidenzia l’interfaccia di interazione. La rappresentazione schematica mostra i residui attivi dell’interfaccia del dimero HER2I714 e HER3V945, così come i due residui in HER2 unici per questa interfaccia per un’ulteriore analisi mutazionale.b) HER2 legato con Lapatinib nell’orientamento simmetrico testa a testa del dimero simile a HER3(Jura et al., 2009b). I residui specifici del dimero sono evidenziati nello schema.(c) Le cellule MCF7 sono state trasfettate con la codifica vettoriale HER2wt-GFP, HER2N764R/K765F-GFP o HER2I748R/V750R-GFP e HER3wt-HA. Le cellule sono state incubate per 24 ore e inibite con 10 µM lapatinib per 1 ora, prima della fissazione e della colorazione con anticorpo anti-HA coniugato con Alexa-546. I dati rappresentati come media ±SEM. ****p≤0,0001, come analizzato da ANOVA unidirezionale. Barre di scala 5 μm dati corrispondenti e statistiche disponibili come Figura 5 – dati fonte 1. Modello molecolare per le interazioni in(a) e(b) disponibile come Figura 5 – dati sorgente 2 e 3. 2. I residui che segnano l’interfaccia del dimero dell’eterodimero HER2-HER3 indotto da lapatinib, nelle conformazioni modellate come EGFR o come HER3, compresa l’area superficiale accessibile al solvente per residuo (in Å 2),sono disponibili come dati fonte della Figura 5 4.10.7554/eLife.32271.025Figure 5-source data 1.Numerical data and statistics relating to Figure 5.10.7554/eLife.32271.026Figure 5-figure 5-source data file di struttura 2.PDB del modello di interazione molecolare in Figura 5a.10.7554/eLife.32271.027Figure 5-figure 5-source data 3.PDB structure file del modello di interazione molecolare in Figura 5b.10.7554/eLife.32271.028Figure 5-source data 4.Table con i residui di interfaccia modellati, compreso il per-residue solvent-area superficiale accessibile in Å 2.2. Modello molecolare del dimero HER2-HER3 indotto da lapatinib nel(a) EGFR-like e(b) orientamento HER3-like con residui di interfaccia mostrati come bastoncini.

Figura 5-figure supplement 1.Modelli molecolari di potenziali orientamenti del dimero HER2-HER3 indotto da lapatinib.2. Modello molecolare del dimero HER2-HER3 indotto da lapatinib nel(a) EGFR-like e(b) orientamento HER3-like con residui di interfaccia mostrati come bastoncini.

Figura 5-video 1.Figura 5—video 1. Vista dell’interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione EGFR-like, con HER2I714 e HER3V945 evidenziati, così come i residui di interfaccia specifici del modello HER2I748/V750.

Figura 5-video 2.Figura 5—video 2. Evidenziazione dell’interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione simile a HER3, con HER2I714 e HER3V945 evidenziati, così come i residui di interfaccia specifici del modello HER2N764/K765.
La dimerizzazione testa a testa HER2-HER3 è necessaria per la proliferazione indotta da inibitore
Dopo aver presentato la modellazione e i dati FRET/FLIM coerenti con un orientamento del dimero HER2-HER3 indotto da lapatinib, essendo distinto dall’interfaccia attiva del dimero attivatore/ricevitore, abbiamo cercato di identificare quale tipo di interazione HER2-HER3 ha causato la crescita co-stimolante NRG-lapatinib osservata nei saggi di proliferazione 2D.
In questi saggi, non abbiamo ectopicamente introdurre il mutante HER2N764R/K765F ectopicamente, perché, in primo luogo, potrebbe anche interrompere l’attivo, asimmetrico HER2-HER3 interfaccia eterodimero HER2-HER3 e in secondo luogo, le cellule SKBR3 hanno un gran numero di recettori endogeni HER2 che ostacolerebbe l’analisi del comportamento di HER2N764R/K765F ectopicamente espresso. Invece abbiamo identificato HER3L700F come il mutante reciproco di HER2N764R/K765F(Figura 6a, Figura 6-video 1). Abbiamo introdotto HER3L700F in cellule SKBR3 per indagare il ruolo del testa a testa, l’interfaccia dimero simmetrica nella proliferazione sinergica lapatinib-NRG descritto sopra. Mentre il mutante HER3V945R dimero attivo HER3V945R non ha interrotto la proliferazione cooperativa fattore di crescita della droga, il mutante HER3L700F ha fatto(Figura 6d-e). Sia HER3L700F e HER3V945R sono stati espressi sulla superficie cellulare, come misurato dalla citometria a flusso(Figura 2-figure supplemento 2). Combinato, questo indica che l’eterodimero inibitore-indotto di HER2 e HER3 è coerente con un testa a testa, la conformazione simmetrica, e svolge un ruolo importante negli effetti sinergici proliferativi di lapatinib e NRG. Anche se questa conformazione è stata descritta dal reticolo cristallino del dominio chinasi HER3(Jura et al., 2009b), a nostra conoscenza è la prima volta che un ruolo funzionale è stato attribuito agli eterodimeri coerente con questa interfaccia nelle cellule.
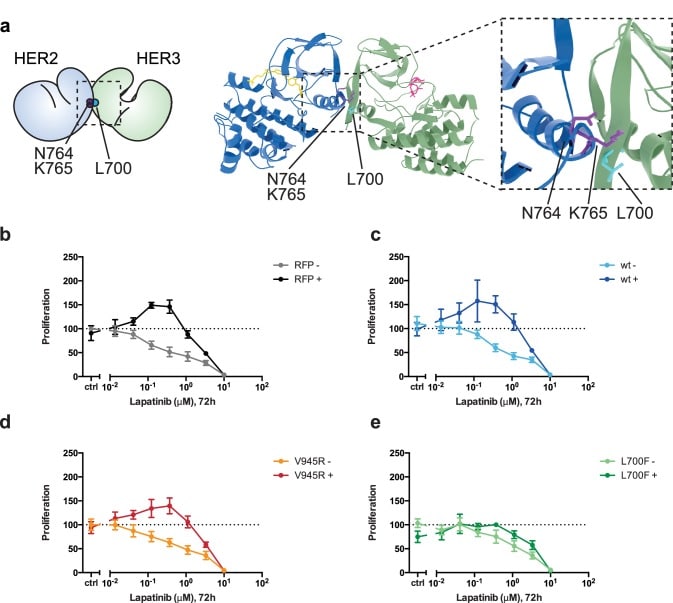
Figura 6-video 1.Figura 6—video 1. Perturbazione del dimero indotto da lapatinib inibisce la crescita sinergica lapatinib-NRG.dati numerici e statistiche relative alla Figura 6.Dati numerici e statistiche relative alla Figura 6.Interface evidenziano il modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione HER3-like, con HER2I714 e HER3V945 evidenziato, così come i residui di interfaccia modello-specifici HER2N764/K765 e HER3L700.a) Modello molecolare del dimero HER2-HER3 indotto da lapatinib con i residui di interfaccia lapatinib-dimero HER2N764 e HER2K765 evidenziati (viola) e un potenziale residuo reciproco HER3L700F (ciano).b-e) saggi di proliferazione2D di cellule SKBR3 trasfettate con b) vettore vuoto RFP, c) HER3wt,d) HER3V945R,o e) HER3L700Fe trattate con lapatinib ±10 nM NRG come prima. I dati rappresentano la media ±SEM per sei esperimenti indipendenti, ciascuno eseguito in triplice copia. Dati corrispondenti e statistiche disponibili come Figura 6 – dati fonte 1.10.7554/eLife.32271.033Cifra dati a 6 fonti 1.Dati numerici e statistiche relative alla Figura 6.

Figura 6-video 1.Dati numerici e statistiche relative alla Figura 6. Dati numerici e statistiche relative alla Figura 6. Evidenziazione dell’interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione simile a HER3, con HER2I714 e HER3V945 evidenziati, così come i residui di interfaccia specifici del modello HER2N764/K765 e HER3L700.a) Modello molecolare del dimero HER2-HER3 indotto da lapatinib con i residui di interfaccia lapatinib-dimero HER2N764 e HER2K765 evidenziati (viola) e un potenziale residuo reciproco HER3L700F (ciano).b-e) saggi di proliferazione2D di cellule SKBR3 trasfettate con b) vettore vuoto RFP, c) HER3wt,d) HER3V945R,o e) HER3L700Fe trattate con lapatinib ±10 nM NRG come prima. I dati rappresentano la media ±SEM per sei esperimenti indipendenti, ciascuno eseguito in triplice copia. Dati corrispondenti e statistiche disponibili come Figura 6 – dati fonte 1.10.7554/eLife.32271.033Cifra dati a 6 fonti 1.Dati numerici e statistiche relative alla Figura 6.

Figura 6-video 1.2. Evidenziazione dell’interfaccia del modello molecolare di un eterodimero HER2-HER3 indotto da lapatinib nella conformazione simile a HER3, con HER2I714 e HER3V945 evidenziati, così come i residui di interfaccia specifici del modello HER2N764/K765 e HER3L700.
Discussione
Le dinamiche conformazionali dell’eterodimerizzazione HER2-HER3 sono una considerazione importante per valutare le strategie di intervento terapeutico mirate esistenti e future contro il cancro al seno HER2+ e altri tumori della famiglia HER. Qui dimostriamo che l’inibitore HER2 lapatinib è paradossalmente in grado di promuovere un comportamento proliferativo nelle cellule tumorali della mammella HER2+ se somministrato in presenza del ligando HER3 NRG. La sinergia tra fattore di crescita e inibitore richiede una cascata intricata e multifase di eventi conformazionali.
Lapatinib è in grado di promuovere l’eterodimerizzazione tra i domini chinasi di HER2 e HER3, stabilizzando un orientamento coerente con un eterodimero simmetrico, testa a testa del dominio chinasi che è distinto dall’orientamento canonico, asimmetrico, testa a coda del dominio chinasi attivo che si verifica in tutta la famiglia EGFR. Un’interfaccia analoga è stata osservata in precedenza nel reticolo cristallino del dominio chinasi HER3(Jura et al., 2009b); in questo caso, abbiamo fornito prove modellistiche e cellulari di un eterodimero con un’interfaccia coerente con quella osservata nel reticolo cristallino del dominio chinasi HER3. Sequestrare HER2 e HER3 in questi eterodimeri inattivi, legati a lapatinib, è stato di beneficio per la segnalazione proliferativa mediata da NRG. I nostri risultati, in cui il legame inibitore guida la formazione di dimeri che aumenta la segnalazione e la proliferazione, mostra alcuni paralleli con i fenotipi di segnalazione indotti dall’inibitore nella famiglia RAF(Eyers et al., 1998; Hatzivassiliou et al., 2010; McKay et al., 2011; Poulikakos et al., 2010; Thevakumaran et al., 2015).
Mentre l’analisi FRET-FLIM della dimerizzazione indotta da lapatinib non è stata in grado di distinguere tra eterodimeri o oligomeri di ordine superiore, i nostri dati di clustering mostrano che lapatinib è in grado di indurre oligomeri di ordine superiore. A causa della natura simmetrica modellata di questi dimeri indotti da lapatinib, in cui sia HER2 che HER3 legati a lapatinib sarebbero conformativamente disponibili come recettori “attivatori” per ulteriori partner di oligomerizzazione, non è inconcepibile che essi possano agire come punti di nucleazione per piattaforme di segnalazione oligomeriche più grandi. Tali array di segnalazione, in cui la cooperatività reciproca aumenta l’output di segnalazione, sono stati proposti per gli oligomeri EGFR(Huang et al., 2016).
L’aggiunta del ligando causa potenzialmente riarrangiamenti all’interno di queste piattaforme attraverso il balletto conformazionale indotto dal ligando di interazioni multilivello tra i vari domini extracellulari e intracellulari dei recettori della famiglia EGFR (rivisto in(Lemmon et al., 2014)). La formazione di piattaforme oligomeriche indotte da lapatinib può facilitare la transizione in eterodimeri di segnalazione attiva su legante ligando, grazie alla disponibilità di partner di dimerizzazione nelle immediate vicinanze all’interno di queste piattaforme oligomeriche indotte da farmaci.
Sia l’eterodimerizzazione HER2-HER3 indotta da lapatinib che gli effetti sinergici a valle di lapatinib-NRG sulla proliferazione dipendono dalla capacità di HER3 di legare l’ATP. Anche se di solito classificato come pseudochinasi, HER3 ha dimostrato di mantenere una misura dell’attività di autofosforilazione (non di transfosforilazione) in circostanze specifiche(Shi et al., 2010). Mostriamo che l’eterodimerizzazione di HER2-HER3 e gli effetti proliferativi a valle possono essere generati dall’aggiunta dell’inibitore HER3-binding bosutinib, indicando che l’occupazione delle tasche di legame nucleotidico svolge un ruolo strutturale critico per la funzione di HER3, e apparentemente indipendente da qualsiasi attività catalitica mantenuta. L’osservazione di un aumento delle eterointerazioni e della proliferazione cellulare dovuto all’inibizione di una chinasi carente di attività è una forte indicazione dell’importanza del legame ATP in alcune pseudochinasi, e la necessità di conformatori strutturali occupati dalla tasca nel sostenere le interazioni proteina-proteina e la successiva uscita di segnalazione a valle.
A causa dell’importanza di HER3 nella resistenza alla terapia mirata HER2, il suo legame ATP conservato ha aumentato la possibilità di mirare HER3 con gli inibitori delle chinasi ATP-competitive. I nostri dati mostrano, tuttavia, che la stabilizzazione del dominio chinasi HER3 con un inibitore della chinasi competitivo ATP può avere un effetto stimolante sulla proliferazione delle cellule tumorali della mammella HER2+. Ciò indica che lo sviluppo di una terapia mirata a piccole molecole contro l’HER3 per l’impiego nel carcinoma mammario HER2+ deve essere indirizzato lontano dalla stabilizzazione del conformer occupato dalla tasca di legame dell’ATP HER3 e piuttosto verso la stabilizzazione dell’apo, conformer inattivo. Un’eccezione a questo potrebbe essere lo sviluppo di inibitori irreversibili e adamantanici dell’HER3, legati in modo irreversibile, che colpiscono il recettore per la degradazione proteosomica(Xie et al., 2014).
L’effetto sostanziale che dosi più basse di lapatinib hanno sulla proliferazione in presenza di fattore di crescita può avere un impatto sulla costituzione di lapatinib-resistenza in vivo. Questo è in accordo con l’osservazione dei modelli di xenotrapianto che la resistenza si verifica molto più facilmente se lapatinib viene somministrato in modo continuo a basse dosi che se viene utilizzato in modo intermittente ad alte dosi (Aminet al., 2010). L’aumento della produzione di fattori di crescita (compreso l’NRG) è un meccanismo di resistenza ben descritto contro la terapia mirata HER2 (rivisto in [Clauset al., 2014]). La produzione di NRG da parte del microambiente ha anche dimostrato di avere un ruolo nella diffusione metastatica delle cellule tumorali ovariche che esprimono alti livelli di HER3(Pradeep et al., 2014). Gli elevati livelli di espressione di NRG nelle pazienti con tumore al seno HER2+ hanno mostrato una forte correlazione con la recidiva della malattia(Xia et al., 2013). Diverse mutazioni somatiche in HER3 osservate nel cancro rientrano nel dominio extracellulare e hanno un potenziale effetto sull’affinità legante(Jaiswal et al., 2013). Queste mutazioni possono esacerbare il comportamento sinergico del fattore di crescita inibitore-fattore di crescita qui riportato.
I nostri risultati forniscono un potenziale meccanismo molecolare per i risultati deludenti osservati in un recente studio di Fase III di lapatinib utilizzato in un ambiente adiuvante (studio ALTTO)(Piccart-Gebhart et al., 2016). Il braccio di lapatinib di questo studio è stato terminato prematuramente, e gli effetti osservati nel setting adiuvante sia per il co-trattamento lapatinib-trastuzumab che per il trattamento trastuzumab seguito da lapatinib non sono stati significativi. Questi risultati clinici indicano che ci sono fattori complicanti nell’ostacolare l’efficacia di lapatinib nei pazienti, che possono coinvolgere i livelli di espressione della stimolazione HER3 e NRG da un microambiente tumorale complesso. Le complesse relazioni tra le distinte dinamiche di conformazione delle proteine, la formazione di assemblaggi oligomerici, la disponibilità di ligandi e i vari effetti sulla segnalazione a valle devono essere considerati quando si applica una terapia mirata per evitare la proliferazione delle cellule tumorali potenzialmente inaspettata dopo il trattamento con inibitori.
Materiali e metodi
Reagenti e anticorpi
NRG1 è stato acquistato da PeproTech. Lapatinib è stato un gentile regalo del professor György Kéri (Vichem Chemie Research Ltd Ungheria). Bosutinib è stato acquistato da LC Labs. Gli anticorpi totali HER2, HER3, PKB, HER2 pY877, HER3 pY1289, PKB pS473 e ERK1/2 pT202/pY204 sono stati acquistati da Cell Signaling Technology, gli anticorpi anti-α-tubulina di Sigma, ERK1/2 totale di Merck e Alexa Fluor-488 coniugati con anticorpo anti-HER3 dei sistemi di ricerca e sviluppo.
Coltura cellulare e trasfezione di plasmidi
Le cellule MCF7 e ZR75 sono state coltivate in DMEM integrato con il 10% di FCS, le cellule SKBR3 sono state coltivate in McCoys medio integrato con il 10% di FCS. BT474, AU565, HCC1419, e HCC1569 cellule sono state coltivate in RPMI con il 10% FCS. Per le cellule BT474 10 μg / ml di insulina bovina è stato incluso nel terreno di coltura.
Le cellule sono state trasfettate con DNA plasmidico utilizzando FuGENE 6, FuGENE HD (Roche), o Lipofectamina LTX (Thermo Fisher Scientific) secondo il protocollo del produttore.
Tutte le linee cellulari sono state ottenute dalla struttura Cell Services del Francis Crick Institute, dove sono state testate negative per il micoplasma e autenticate tramite profilazione STR.
Saggi di proliferazione
Per i saggi di proliferazione 2D, le cellule sono state placcate a 1 × 104 cellule/pozzetto in una piastra a 96 pozzetti. Il giorno successivo sono state sottoposte a trattamento per 72 ore, seguite dall’aggiunta del reagente CellTiter-Glo (Promega) e misurate su un lettore di piastre EnVision (Perkin Elmer). I dati CellTiter-Glo sono stati normalizzati al fattore di crescita-nullo/inibitore-nullo controllo non trattato. Ciò ha causato alcuni appezzamenti trattati con il fattore di crescita a partire da livelli superiori alla linea di base, il che è un’indicazione dell’effetto proliferativo che il trattamento con il fattore di crescita ha avuto in queste cellule.
Per i saggi sferoidi 3D, 3 × 103 cellule sono state placcate in una piastra di attacco a 96 pozzetti, a fondo tondo, ultra-bassa (Corning) in presenza dell’1% di Matrigel (Corning). Dopo tre giorni di crescita, un volume uguale di 2x media contenenti condizioni di trattamento è stato aggiunto e rinfrescato ogni tre giorni per un totale di otto giorni di trattamento. Le immagini a contrasto di fase sono state scattate con un microscopio Zeiss Axiovert 40 CFL con obiettivo Zeiss 5x A-plan e analizzate con ImageJ.
Citometria a flusso
Le cellule SKBR3 sono state trasfettate con mutanti RFP-HER3 per 48 ore. Le cellule sono state pretrattate con 0,5 mM EDTA per facilitare la rimozione dal substrato e colorate per l’espressione extracellulare HER3 utilizzando l’anticorpo anti-HER3 coniugato con Alexa Fluor-488 (sistemi R&D, clone 66223) secondo le istruzioni del produttore. In breve, le cellule sono state bloccate utilizzando il mouse IgG (Santa Cruz Anticorpi) per 15 minuti a temperatura ambiente, seguita da incubazione con anticorpo coniugato per 30 minuti a temperatura ambiente al buio. Le cellule sono state lavate in PBS, 0,5% BSA, 0,1% sodio azide tre volte prima dell’analisi citometrica a flusso utilizzando uno strumento BD Fortessa (BD). I risultati sono stati analizzati utilizzando il software Flo-Jo.
Determinazione FRET mediante misurazioni FLIM
Il trasferimento di energia di risonanza a fluorescenza (FRET) è utilizzato per quantificare le interazioni dirette proteina-proteina e le modifiche post-traslazionali. L’elaborazione delle cellule per la determinazione del FRET da parte di FLIM è stata precedentemente descritta(Barber et al., 2009; Parsons e Ng, 2002). FLIM è stata eseguita utilizzando il conteggio a singolo fotone correlato al tempo (TCSPC) con un sistema di microscopio multifotone come descritto in precedenza(Peter et al., 2005). Per gli esperimenti di misurazione delle proteine endogene, le coppie FRET erano Cy5-coniugate anti-HER2 IgG, e Alexa546-coniugate anti-HER3 IgG. Per le misurazioni delle proteine esogene, le coppie FRET erano HER2-GFP e HER3-HA con un anti-HA IgG, etichettate con un fluoroforo Cy3. L’efficienza FRET tra le proteine legate al donatore e all’accettore è stata calcolata con la seguente equazione in ogni pixel e la media per cella: FRET eff = 1-tau(DA)/tau(controllo) dove tau(DA) è la vita visualizzata dalle cellule che co-esprimono il donatore e l’accettore, mentre tau (controllo) è la vita media del donatore (GFP), misurata in assenza dell’accettore.
Modellazione dei dimeri HER2-HER3
Abbiamo modellato il dimero HER2-HER3 mediante la modellazione comparativa dell’omologia utilizzando un approccio a modelli multipli. Il dimero attivo e asimmetrico HER2-HER3 è stato modellato utilizzandocome modelli la struttura cristallina del dominio attivo della chinasi EGFR (PDB ID 2GS2) (Zhanget al., 2006) e una catena della struttura cristallina dell’omodimero HER3 (PDB ID 3KEX) (Jura et al., 2009b). Per costruire il dimero simmetrico EGFR, inattivo, abbiamo utilizzato la struttura cristallina dell’omodimero EGFR (PDB ID 3GT8) (Juraet al., 2009a), la struttura cristallina dell’EGFR complessata con lapatinib (PDB ID 1XKK) (Wood et al., 2004) e una sola catena della struttura cristallina dell’omodimero HER3 (PDB ID 3KEX) (Jura et al.,2009b). Per costruire il dimero simile a HER3, abbiamo utilizzato la struttura dell’omodimero HER3 (PDB ID 3KEX) (Juraet al., 2009b), la struttura cristallina del lapatinib legato con EGFR (PDB ID 1XKK) (Wood et al., 2004) e la struttura cristallina del legato inattivo EGFR AMP-PNP (PDB ID 2GS7) (Zhang et al.,2006). L’allineamento sequenziale utilizzato per costruire il modello è stato creato utilizzando PRALINE con l’omologa strategia di allineamento esteso(Simossis et al., 2005). Abbiamo generato 200 modelli tridimensionali utilizzando il pacchetto MODELLER (Salie Blundell, 1993). I modelli selezionati sono stati scelti sulla base del punteggio DOPE della funzione obiettivo di MODELLER.
Il volume della tasca di legame HER2 ATP è stato calcolato con il pacchetto SURFNET 1.5 (Laskowski, 1995), dove le regioni delle cavità di una proteina sono costruite inserendo una sfera di sonda di 1.4 Å 3 negli spazi tra gli atomi.
L’allineamento strutturale è stato eseguito utilizzando lo strumento multi-seq del pacchetto VMD 1.9.1 (Humphreyet al., 1996), e la misurazione dei residui interrati di superficie di interazione è stata effettuata utilizzando POPScomp (Kleinjunge Fraternali, 2005).
Saggi di clustering del recettore
L’imaging STORM e le analisi dei cluster sono descritte più in dettaglio al Bioprotocollo(Roberts et al., 2018). Le cellule SKBR3 sono state trattate con 14 nM Lapatinib o 41 nM Bosutinib. HER2 e HER3 Affibody ligandi sono stati utilizzati per etichettare gli stati non attivati dei recettori (HER2 da Affibody Inc. e la codifica plasmide HER3 affibody è stato un dono di John Löfblom, proteina fatta in casa e ha dimostrato di legarsi specificamente ai recettori HER3) e NRG-β1 (Peprotech) è stato utilizzato per stimolare le cellule. La coniugazione di coloranti (Invitrogen) a HER2 e HER3 ligandi è stato fatto in casa e il rapporto di colorante: ligando è stato confermato essere ~ 1:1. Il coniugato NRG-dye coniugato ha dimostrato di essere attivo come la proteina non etichettata. Abbiamo incubato le cellule in 100 nM HER2Affibody-Alexa488 + 50 nM HER3Affibody-Alexa647 o 100 nM HER2Affibody-Alexa488 + 10 nM NRG-Alexa647 ± droga per 1 ora. Le cellule sono state fissate chimicamente utilizzando il 4% di paraformaldeide (soluzioni EMS) +0,5% di glutaraldeide (Sigma-Aldrich) diluito in PBS ghiacciato.
I campioni sono stati ripresi utilizzando un microscopio a super-risoluzione Zeiss Elyra per eccitare stocasticamente i fluorofori Alexa488 e Alexa647 legati ai recettori nelle cellule e alle singole molecole di immagine. L’imaging è stato fatto in modalità TIRF utilizzando un obiettivo ad immersione ad olio 100x. Abbiamo utilizzato una linea laser a 405 nm per aiutare il lampeggio del fluoroforo e linee laser a 488 nm o 640 nm per eccitare i fluorofori, alternando i laser per l’immagine dei due recettori in modo indipendente ogni 300 fotogrammi, su un totale di ~10.000 fotogrammi. Il tempo di esposizione è stato di 20 ms. Un minimo di due repliche di ogni campione sono stati ripresi generando almeno 12 regioni (25,6 μm x 25,6 μm) che coprono almeno una cella per regione. Il software Zen ha localizzato i singoli punti molecolari nelle cellule, è stata impostata una soglia per scartare i punti di sfondo e le coordinate delle localizzazioni positive (tipicamente 30.000 + per HER2 e 5.000 + per HER3 per regione) sono state passate nell’algoritmo di identificazione dei cluster bayesiani (Rubin-Delanchy et al., 2015).
L’algoritmo di clustering prevede che lo sfondo e i cluster siano distribuiti uniformemente su un ROI rettangolare(Rubin-Delanchy et al., 2015). Le immagini analizzate hanno mostrato principalmente singole celle. Di interesse sono i recettori HER2 e HER3 nella membrana cellulare, che erano visibili come forma circolare. Al fine di conformarsi ai prerequisiti degli algoritmi di clustering, sono state selezionate manualmente regioni rettangolari che coprono strettamente la membrana cellulare utilizzando gli angoli più adatti (valutati con l’ispezione visiva). L’intera membrana cellulare è stata coperta in questo modo. I dati selezionati da queste regioni sono stati ruotati in modo che i lati dei rettangoli diventassero paralleli all’asse delle coordinate. Il risultato è stato utilizzato come input per l’algoritmo di clustering e l’algoritmo è stato applicato come descritto dal protocollo. Per la presentazione sono state utilizzate le liste complete delle molecole per cluster prodotte dall’algoritmo.
Purificazione HER3 KD ricombinante
La costruzione del dominio baculovirale HER3 kinase è stata gentilmente fornita dal Prof. Mark Lemmon, University of Pennsylvania. Sf21 cellule a 1 × 106 cellule/ml sono state infettate dal virus P3 (7 × 107 pfu/ml) con un MOI di 1,0 e hanno potuto crescere per tre giorni. Le cellule sono state lisate in tampone di lisi contenente inibitori della proteasi, 1 mM DTT e 2 mM BME. Il lisato è stato chiarito mediante centrifugazione e incubato con resina NiNTA (Qiagen) per 30 minuti a 4°C, dopo di che la resina è stata lavata ampiamente con tampone contenente 50 mM Hepes (pH 7,6), 300 mM NaCl, 2 mM BME, 5% glicerolo, 10 mM imidazolo. HER3 è stato eluito nello stesso tampone con 200 mM imidazolo aggiunto.
Ogni eluizione è stata centrifugata a 10.000 rpm per rimuovere qualsiasi precipitato o resina e applicata ad una colonna di filtrazione in gel S200 in 50 mM HEPES (pH 7,6), 300 mM NaCl, 2 mM BME, 2 mM BME, 2,5% di glicerolo.
Saggio di spostamento termico (TSA)
I saggi di spostamento termico sono stati effettuati come descritto in(Niesen et al., 2007). In breve, in una piastra RT-PCR a 96 pozzetti (Life Technologies) 1 μg di dominio chinasi HER3 è stato incubato con 1 μM inibitore o 200 μM ATP/10 mM MgCl2 (come indicato) per 30 minuti a 4°C in presenza del colorante Sypro Orange (Sigma). HER2 esperimenti TSA sono stati eseguiti in una piastra RT-PCR a 384 pozzetti (Thermo Fisher Scientific). 0,5 μg di HER2 dominio chinasi HER2 / pozzetto è stato incubato con 1 μM lapatinib, 1 μM bosutinib, o 200 μM ATP/10 mM MgCl2 per 20 minuti a 4 ° C. Le misurazioni HER3 sono state effettuate su una macchina PCR Applied Biosystems 7500 Fast Real-Time PCR, e le misurazioni HER2 su una macchina PCR Applied Biosystems Quant Studio 7. I dati sono stati tagliati e una curva sigmoidale di Boltzmann montata in GraphPad Prism 6. Il punto di inflessione del sigmoidal di Boltzmann è stato preso come Tm. I valori di spostamento termico ΔTm sono stati ottenuti sottraendo il valore Tm del solo controllo del dominio chinasi.
Analisi del Western blot
Le celle sono state placcate a 0,5 × 105 celle/pozzetto in piastre da 24 pozzetti. Le cellule sono state lisate in 1x tampone campione (contenente 1 mM DTT), sonicate e centrifugate. Dopo la centrifugazione, i lisati sono stati sottoposti a SDS-PAGE e analizzati da western blotting.
Saggio di spostamento termico cellulare (CETSA)
Il CETSA è stato eseguito con cellule COS7 trasfettate con HER3wt-RFP, HER3T787M-RFP o plasmidi HER3KGG-RFP come descritto in(Jafari et al., 2014; Reinhard et al., 2015). In breve, i COS7 sono stati trattati con DMSO o 50 nM bosutinib per 1 ora a 37°C. Le cellule sono state lavate con PBS, staccate e lavate di nuovo due volte con PBS fredda. Pellet cellulari sono stati risospesi in PBS a freddo con inibitori della proteasi (Roche) e 100 microlitri di ogni sospensione cellulare è stato trasferito in provette PCR 0,2 ml. Le provette PCR sono state riscaldate per 3 minuti a 42°C o 50°C in un termociclatore (DNA Engine DYAD, MJ research, Peltier thermal cycler) e incubate a temperatura ambiente per 3 minuti. Le provette sono state poi immediatamente trasferite su ghiaccio, 35 μl di PBS 1,4% NP-40 freddo con inibitori della proteasi sono stati aggiunti e le provette sono state congelate a scatto. I campioni sono stati poi sottoposti a due cicli di gelo-disgelo (a 25 ° C) e lisati cellulari sono stati centrifugati a 20.000 g per 1 ora a 4 ° C. I surnatanti sono stati accuratamente rimossi e analizzati da western blot.
References
- Amin DN, Sergina N, Ahuja D, McMahon M, Blair JA, Wang D, Hann B, Koch KM, Shokat KM, Moasser MM. Resiliency and vulnerability in the HER2-HER3 tumorigenic driver. Science Translational Medicine. 2010; 2DOI | PubMed
- Arteaga CL, Ramsey TT, Shawver LK, Guyer CA. Unliganded epidermal growth factor receptor dimerization induced by direct interaction of quinazolines with the ATP binding site. Journal of Biological Chemistry. 1997; 272:23247-23254. DOI | PubMed
- Barber PR, Ameer-Beg SM, Gilbey J, Carlin LM, Keppler M, Ng TC, Vojnovic B. Multiphoton time-domain fluorescence lifetime imaging microscopy: practical application to protein-protein interactions using global analysis. Journal of the Royal Society Interface. 2009; 6:S93-S105. DOI
- Baselga J, Swain SM. Novel anticancer targets: revisiting ERBB2 and discovering ERBB3. Nature Reviews Cancer. 2009; 9:463-475. DOI | PubMed
- Blackwell KL, Burstein HJ, Storniolo AM, Rugo H, Sledge G, Koehler M, Ellis C, Casey M, Vukelja S, Bischoff J, Baselga J, O’Shaughnessy J. Randomized Study of Lapatinib Alone or in Combination With Trastuzumab in Women With ErbB2-Positive, Trastuzumab-Refractory Metastatic Breast Cancer. Journal of Clinical Oncology. 2010; 28:1124-1130. DOI | PubMed
- Boudeau J, Miranda-Saavedra D, Barton GJ, Alessi DR. Emerging roles of pseudokinases. Trends in Cell Biology. 2006; 16:443-452. DOI | PubMed
- Bublil EM, Pines G, Patel G, Fruhwirth G, Ng T, Yarden Y. Kinase-mediated quasi-dimers of EGFR. The FASEB Journal. 2010; 24:4744-4755. DOI | PubMed
- Cameron AJM, Escribano C, Saurin AT, Kostelecky B, Parker PJ. PKC maturation is promoted by nucleotide pocket occupation independently of intrinsic kinase activity. Nature Structural & Molecular Biology. 2009; 16:624-630. DOI
- Cameron D, Piccart-Gebhart MJ, Gelber RD, Procter M, Goldhirsch A, de Azambuja E, Castro G, Untch M, Smith I, Gianni L, Baselga J, Al-Sakaff N, Lauer S, McFadden E, Leyland-Jones B, Bell R, Dowsett M, Jackisch C. 11 years’ follow-up of trastuzumab after adjuvant chemotherapy in HER2-positive early breast cancer: final analysis of the HERceptin Adjuvant (HERA) trial. The Lancet. 2017; 389:1195-1205. DOI
- Claus J, Cameron AJM, Parker PJ. Pseudokinase drug intervention: a potentially poisoned chalice. Biochemical Society Transactions. 2013; 41:1083-1088. DOI | PubMed
- Claus J, Patel G, Ng T, Parker PJ. A role for the pseudokinase HER3 in the acquired resistance against EGFR- and HER2-directed targeted therapy. Biochemical Society Transactions. 2014; 42:831-836. DOI | PubMed
- Clayton AHA, Walker F, Orchard SG, Henderson C, Fuchs D, Rothacker J, Nice EC, Burgess AW. Ligand-induced dimer-tetramer transition during the activation of the cell surface epidermal growth factor receptor-A multidimensional microscopy analysis. Journal of Biological Chemistry. 2005; 280:30392-30399. DOI | PubMed
- Clayton AHA, Tavarnesi ML, Johns TG. Unligated epidermal growth factor receptor forms higher order oligomers within microclusters on A431 cells that are sensitive to tyrosine kinase inhibitor binding. Biochemistry. 2007; 46:4589-4597. DOI | PubMed
- CLEOPATRA Study Group, Swain SM, Baselga J, Kim SB, Ro J, Semiglazov V, Campone M, Ciruelos E, Ferrero JM, Schneeweiss A, Heeson S, Clark E, Ross G, Benyunes MC, Cortés J. Pertuzumab, trastuzumab, and docetaxel in HER2-positive metastatic breast cancer. New England Journal of Medicine. 2015; 372:724-734. DOI | PubMed
- Coban O, Zanetti-Dominguez LC, Matthews DR, Rolfe DJ, Weitsman G, Barber PR, Barbeau J, Devauges V, Kampmeier F, Winn M, Vojnovic B, Parker PJ, Lidke KA, Lidke DS, Ameer-Beg SM, Martin-Fernandez ML, Ng T. Effect of phosphorylation on EGFR dimer stability probed by single-molecule dynamics and FRET/FLIM. Biophysical Journal. 2015; 108:1013-1026. DOI | PubMed
- Cui J, Xiao J, Tagliabracci VS, Wen J, Rahdar M, Dixon JE. A secretory kinase complex regulates extracellular protein phosphorylation. eLife. 2015; 4DOI | PubMed
- Cui J, Zhu Q, Zhang H, Cianfrocco MA, Leschziner AE, Dixon JE, Xiao J. Structure of Fam20A reveals a pseudokinase featuring a unique disulfide pattern and inverted ATP-binding. eLife. 2017; 6DOI | PubMed
- Davis MI, Hunt JP, Herrgard S, Ciceri P, Wodicka LM, Pallares G, Hocker M, Treiber DK, Zarrinkar PP. Comprehensive analysis of kinase inhibitor selectivity. Nature Biotechnology. 2011; 29:1046-1051. DOI
- Diéras V, Miles D, Verma S, Pegram M, Welslau M, Baselga J, Krop IE, Blackwell K, Hoersch S, Xu J, Green M, Gianni L. Trastuzumab emtansine versus capecitabine plus lapatinib in patients with previously treated HER2-positive advanced breast cancer (EMILIA): a descriptive analysis of final overall survival results from a randomised, open-label, phase 3 trial. The Lancet Oncology. 2017; 18:732-742. DOI | PubMed
- Dong C-L, Guo F-C, Xue J. Computational insights into HER3 gatekeeper T768I resistance mutation to bosutinib in HER3-related breast cancer. Medicinal Chemistry Research. 2017; 26:1926-1934. DOI
- EMILIA Study Group, Verma S, Miles D, Gianni L, Krop IE, Welslau M, Baselga J, Pegram M, Oh DY, Diéras V, Guardino E, Fang L, Lu MW, Olsen S, Blackwell K. Trastuzumab emtansine for HER2-positive advanced breast cancer. New England Journal of Medicine. 2012; 367:1783-1791. DOI | PubMed
- Eyers PA, Craxton M, Morricel N, Cohen P, Goedert M. Conversion of SB 203580-insensitive MAP kinase family members to drug-sensitive forms by a single amino-acid substitution. Chemistry & Biology. 1998; 5:321-328. DOI | PubMed
- Garrett TPJ, McKern NM, Lou M, Elleman TC, Adams TE, Lovrecz GO, Zhu H-J, Walker F, Frenkel MJ, Hoyne PA, Jorissen RN, Nice EC, Burgess AW, Ward CW. Crystal structure of a truncated epidermal growth factor receptor extracellular domain bound to transforming growth factor α. Cell. 2002; 110:763-773. DOI | PubMed
- Geyer CE, Forster J, Lindquist D, Chan S, Romieu CG, Pienkowski T, Jagiello-Gruszfeld A, Crown J, Chan A, Kaufman B, Skarlos D, Campone M, Davidson N, Berger M, Oliva C, Rubin SD, Stein S, Cameron D. Lapatinib plus capecitabine for HER2-positive advanced breast cancer. New England Journal of Medicine. 2006; 355:2733-2743. DOI | PubMed
- Hammarén HM, Ungureanu D, Grisouard J, Skoda RC, Hubbard SR, Silvennoinen O. ATP binding to the pseudokinase domain of JAK2 is critical for pathogenic activation. PNAS. 2015; 112:4642-4647. DOI | PubMed
- Hatzivassiliou G, Song K, Yen I, Brandhuber BJ, Anderson DJ, Alvarado R, Ludlam MJC, Stokoe D, Gloor SL, Vigers G, Morales T, Aliagas I, Liu B, Sideris S, Hoeflich KP, Jaiswal BS, Seshagiri S, Koeppen H, Belvin M, Friedman LS, Malek S. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth. Nature. 2010; 464:431-435. DOI | PubMed
- Hildebrand JM, Tanzer MC, Lucet IS, Young SN, Spall SK, Sharma P, Pierotti C, Garnier J-M, Dobson RCJ, Webb AI, Tripaydonis A, Babon JJ, Mulcair MD, Scanlon MJ, Alexander WS, Wilks AF, Czabotar PE, Lessene G, Murphy JM, Silke J. Activation of the pseudokinase MLKL unleashes the four-helix bundle domain to induce membrane localization and necroptotic cell death. PNAS. 2014; 111:15072-15077. DOI | PubMed
- Huang Y, Bharill S, Karandur D, Peterson SM, Marita M, Shi X, Kaliszewski MJ, Smith AW, Isacoff EY, Kuriyan J. Molecular basis for multimerization in the activation of the epidermal growth factor receptor. eLife. 2016; 5DOI | PubMed
- Humphrey W, Dalke A, Schulten K. VMD: visual molecular dynamics. Journal of Molecular Graphics. 1996; 14:33-38. DOI | PubMed
- Huse M, Kuriyan J. The conformational plasticity of protein kinases. Cell. 2002; 109:275-282. DOI | PubMed
- Jafari R, Almqvist H, Axelsson H, Ignatushchenko M, Lundbäck T, Nordlund P, Martinez Molina D. The cellular thermal shift assay for evaluating drug target interactions in cells. Nature Protocols. 2014; 9:2100-2122. DOI | PubMed
- Jaiswal BS, Kljavin NM, Stawiski EW, Chan E, Parikh C, Durinck S, Chaudhuri S, Pujara K, Guillory J, Edgar KA, Janakiraman V, Scholz RP, Bowman KK, Lorenzo M, Li H, Wu J, Yuan W, Peters BA, Kan Z, Stinson J, Mak M, Modrusan Z, Eigenbrot C, Firestein R, Stern HM, Rajalingam K, Schaefer G, Merchant MA, Sliwkowski MX, de Sauvage FJ, Seshagiri S. Oncogenic ERBB3 mutations in human cancers. Cancer Cell. 2013; 23:603-617. DOI | PubMed
- Jeffrey PD, Russo AA, Polyak K, Gibbs E, Hurwitz J, Massagué J, Pavletich NP. Mechanism of CDK activation revealed by the structure of a cyclinA-CDK2 complex. Nature. 1995; 376:313-320. DOI | PubMed
- Jura N, Endres NF, Engel K, Deindl S, Das R, Lamers MH, Wemmer DE, Zhang X, Kuriyan J. Mechanism for activation of the EGF receptor catalytic domain by the juxtamembrane segment. Cell. 2009a; 137:1293-1307. DOI | PubMed
- Jura N, Shan Y, Cao X, Shaw DE, Kuriyan J. Structural analysis of the catalytically inactive kinase domain of the human EGF receptor 3. PNAS. 2009b; 106:21608-21613. DOI | PubMed
- Kleinjung J, Fraternali F. POPSCOMP: an automated interaction analysis of biomolecular complexes. Nucleic Acids Research. 2005; 33:W342-W346. DOI | PubMed
- Krop IE, Kim S-B, Martin AG, LoRusso PM, Ferrero J-M, Badovinac-Crnjevic T, Hoersch S, Smitt M, Wildiers H. Trastuzumab emtansine versus treatment of physician’s choice in patients with previously treated HER2-positive metastatic breast cancer (TH3RESA): final overall survival results from a randomised open-label phase 3 trial. The Lancet Oncology. 2017; 18:743-754. DOI | PubMed
- Laskowski RA. SURFNET: A program for visualizing molecular surfaces, cavities, and intermolecular interactions. Journal of Molecular Graphics. 1995; 13:323-330. DOI | PubMed
- Lemmon MA, Schlessinger J, Ferguson KM. The EGFR family: not so prototypical receptor tyrosine kinases. Cold Spring Harbor Perspectives in Biology. 2014; 6DOI | PubMed
- Levinson NM, Boxer SG. a conserved water-mediated hydrogen bond network defines bosutinib’s kinase selectivity. Nature Chemical Biology. 2014; 10:127-132. DOI | PubMed
- Lichtner RB, Menrad A, Sommer A, Klar U, Schneider MR. Signaling-inactive epidermal growth factor receptor/ligand complexes in intact carcinoma cells by quinazoline tyrosine kinase inhibitors. Cancer research. 2001; 61:5790-5795. PubMed
- Littlefield P, Liu L, Mysore V, Shan Y, Shaw DE, Jura N. Structural analysis of the EGFR/HER3 heterodimer reveals the molecular basis for activating HER3 mutations. Science Signaling. 2014; 7DOI | PubMed
- Liu P, Cleveland TE, Bouyain S, Byrne PO, Longo PA, Leahy DJ. A single ligand is sufficient to activate EGFR dimers. PNAS. 2012; 109:10861-10866. DOI | PubMed
- Lovekin C, Ellis IO, Locker A, Robertson JF, Bell J, Nicholson R, Gullick WJ, Elston CW, Blamey RW. c-erbB-2 oncoprotein expression in primary and advanced breast cancer. British Journal of Cancer. 1991; 63:439-443. DOI | PubMed
- McKay MM, Ritt DA, Morrison DK. RAF Inhibitor-Induced KSR1/B-RAF Binding and Its Effects on ERK Cascade Signaling. Current Biology. 2011; 21:563-568. DOI | PubMed
- Mi L-Z, Lu C, Li Z, Nishida N, Walz T, Springer TA. Simultaneous visualization of the extracellular and cytoplasmic domains of the epidermal growth factor receptor. Nature Structural & Molecular Biology. 2011; 18:984-989. DOI
- Murphy JM, Czabotar PE, Hildebrand JM, Lucet IS, Zhang J-G, Alvarez-Diaz S, Lewis R, Lalaoui N, Metcalf D, Webb AI, Young SN, Varghese LN, Tannahill GM, Hatchell EC, Majewski IJ, Okamoto T, Dobson RCJ, Hilton DJ, Babon JJ, Nicola NA, Strasser A, Silke J, Alexander WS. The Pseudokinase MLKL Mediates Necroptosis via a Molecular Switch Mechanism. Immunity. 2013; 39:443-453. DOI | PubMed
- Murphy JM, Zhang Q, Young SN, Reese ML, Bailey FP, Eyers PA, Ungureanu D, Hammaren H, Silvennoinen O, Varghese LN, Chen K, Tripaydonis A, Jura N, Fukuda K, Qin J, Nimchuk Z, Mudgett MB, Elowe S, Gee CL, Liu L, Daly RJ, Manning G, Babon JJ, Lucet IS. A robust methodology to subclassify pseudokinases based on their nucleotide-binding properties. Biochemical Journal. 2014; 457:323-334. DOI | PubMed
- Nagy P, Claus J, Jovin TM, Arndt-Jovin DJ. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. PNAS. 2010; 107:16524-16529. DOI | PubMed
- Needham SR, Roberts SK, Arkhipov A, Mysore VP, Tynan CJ, Zanetti-Domingues LC, Kim ET, Losasso V, Korovesis D, Hirsch M, Rolfe DJ, Clarke DT, Winn MD, Lajevardipour A, Clayton AHA, Pike LJ, Perani M, Parker PJ, Shan Y, Shaw DE, Martin-Fernandez ML. EGFR oligomerization organizes kinase-active dimers into competent signalling platforms. Nature Communications. 2016; 7DOI
- Niesen FH, Berglund H, Vedadi M. The use of differential scanning fluorimetry to detect ligand interactions that promote protein stability. Nature Protocols. 2007; 2:2212-2221. DOI | PubMed
- Novotny CJ, Pollari S, Park JH, Lemmon MA, Shen W, Shokat KM. Overcoming resistance to HER2 inhibitors through state-specific kinase binding. Nature Chemical Biology. 2016; 12:923-930. DOI | PubMed
- Ogiso H, Ishitani R, Nureki O, Fukai S, Yamanaka M, Kim J-H, Saito K, Sakamoto A, Inoue M, Shirouzu M, Yokoyama S. Crystal Structure of the Complex of Human Epidermal Growth Factor and Receptor Extracellular Domains. Cell. 2002; 110:775-787. DOI | PubMed
- Okuzumi T, Fiedler D, Zhang C, Gray DC, Aizenstein B, Hoffman R, Shokat KM. Inhibitor hijacking of Akt activation. Nature Chemical Biology. 2009; 5:484-493. DOI | PubMed
- Ortiz-Zapater E, Lee RW, Owen W, Weitsman G, Fruhwirth G, Dunn RG, Neat MJ, McCaughan F, Parker P, Ng T, Santis G. MET-EGFR dimerization in lung adenocarcinoma is dependent on EGFR mtations and altered by MET kinase inhibition. PLoS One. 2017; 12DOI | PubMed
- Owens MA, Horten BC, Da Silva MM. HER2 Amplification Ratios by Fluorescence In Situ Hybridization and Correlation with Immunohistochemistry in a Cohort of 6556 Breast Cancer Tissues. Clinical Breast Cancer. 2004; 5:63-69. DOI | PubMed
- Papa FR, Zhang C, Shokat KM, Walter P. Bypassing a Kinase Activity with an ATP-Competitive Drug. Science. 2003; 302:1533-1537. DOI | PubMed
- Parsons M, Ng T. Intracellular coupling of adhesion receptors: molecular proximity measurements. Methods in Cell Biology. 2002; 69:261-278. DOI | PubMed
- Peter M, Ameer-Beg SM, Hughes MKY, Keppler MD, Prag S, Marsh M, Vojnovic B, Ng T. Multiphoton-FLIM Quantification of the EGFP-mRFP1 FRET Pair for Localization of Membrane Receptor-Kinase Interactions. Biophysical Journal. 2005; 88:1224-1237. DOI | PubMed
- Piccart-Gebhart M, Holmes E, Baselga J, de Azambuja E, Dueck AC, Viale G, Zujewski JA, Goldhirsch A, Armour A, Pritchard KI, McCullough AE, Dolci S, McFadden E, Holmes AP, Tonghua L, Eidtmann H, Dinh P, Di Cosimo S, Harbeck N, Tjulandin S, Im Y-H, Huang C-S, Diéras V, Hillman DW, Wolff AC, Jackisch C, Lang I, Untch M, Smith I, Boyle F, Xu B, Gomez H, Suter T, Gelber RD, Perez EA. Adjuvant Lapatinib and Trastuzumab for Early Human Epidermal Growth Factor Receptor 2–Positive Breast Cancer: Results From the Randomized Phase III Adjuvant Lapatinib and/or Trastuzumab Treatment Optimization Trial. Journal of Clinical Oncology. 2016; 34:1034-1042. DOI | PubMed
- Poulikakos PI, Zhang C, Bollag G, Shokat KM, Rosen N. RAF inhibitors transactivate RAF dimers and ERK signalling in cells with wild-type BRAF. Nature. 2010; 464:427-430. DOI | PubMed
- Pradeep S, Kim SW, Wu SY, Nishimura M, Chaluvally-Raghavan P, Miyake T, Pecot CV, Kim S-J, Choi HJ, Bischoff FZ, Mayer JA, Huang L, Nick AM, Hall CS, Rodriguez-Aguayo C, Zand B, Dalton HJ, Arumugam T, Lee HJ, Han HD, Cho MS, Rupaimoole R, Mangala LS, Sehgal V, Oh SC, Liu J, Lee J-S, Coleman RL, Ram P, Lopez-Berestein G, Fidler IJ, Sood AK. Hematogenous Metastasis of Ovarian Cancer: Rethinking Mode of Spread. Cancer Cell. 2014; 26:77-91. DOI | PubMed
- Reinhard FBM, Eberhard D, Werner T, Franken H, Childs D, Doce C, Savitski MF, Huber W, Bantscheff M, Savitski MM, Drewes G. Thermal proteome profiling monitors ligand interactions with cellular membrane proteins. Nature Methods. 2015; 12:1129-1131. DOI | PubMed
- Roberts S, Hirsch M, McStea A, Zanetti-Domingues L, Clarke D, Claus J, Parker P, Wang L, Martin-Fernandez M. Cluster analysis of endogenous HER2 and HER3 receptors in SKBR3 cells. Bio-Protocol. 2018; 8DOI
- Ross FA, Hawley SA, Auciello FR, Gowans GJ, Atrih A, Lamont DJ, Hardie DG. Mechanisms of Paradoxical Activation of AMPK by the Kinase Inhibitors SU6656 and Sorafenib. Cell Chemical Biology. 2017; 24:813-824. DOI | PubMed
- Rubin-Delanchy P, Burn GL, Griffié J, Williamson DJ, Heard NA, Cope AP, Owen DM. Bayesian cluster identification in single-molecule localization microscopy data. Nature Methods. 2015; 12:1072-1076. DOI | PubMed
- Saffarian S, Li Y, Elson EL, Pike LJ. Oligomerization of the EGF receptor investigated by live cell fluorescence intensity distribution analysis. Biophysical Journal. 2007; 93:1021-1031. DOI | PubMed
- Sali A, Blundell TL. Comparative protein modelling by satisfaction of spatial restraints. Journal of Molecular Biology. 1993; 234:779-815. DOI | PubMed
- Scheeff ED, Eswaran J, Bunkoczi G, Knapp S, Manning G. Structure of the pseudokinase VRK3 reveals a degraded catalytic site, a highly conserved kinase fold, and a putative regulatory binding site. Structure. 2009; 17:128-138. DOI | PubMed
- Sergina NV, Rausch M, Wang D, Blair J, Hann B, Shokat KM, Moasser MM. Escape from HER-family tyrosine kinase inhibitor therapy by the kinase-inactive HER3. Nature. 2007; 445:437-441. DOI | PubMed
- Shi F, Telesco SE, Liu Y, Radhakrishnan R, Lemmon MA. ErbB3/HER3 intracellular domain is competent to bind ATP and catalyze autophosphorylation. PNAS. 2010; 107:7692-7697. DOI | PubMed
- Simossis VA, Kleinjung J, Heringa J. Homology-extended sequence alignment. Nucleic Acids Research. 2005; 33:816-824. DOI | PubMed
- Slamon DJ, Clark GM, Wong SG, Levin WJ, Ullrich A, McGuire WL. Human breast cancer: correlation of relapse and survival with amplification of the HER-2/neu oncogene. Science. 1987; 235:177-182. DOI | PubMed
- Sliwkowski MX, Schaefer G, Akita RW, Lofgren JA, Fitzpatrick VD, Nuijens A, Fendly BM, Cerione RA, Vandlen RL, Carraway KL. Coexpression of erbB2 and erbB3 proteins reconstitutes a high affinity receptor for heregulin. The Journal of Biological Chemistry. 1994; 269:14661-14665. PubMed
- Thevakumaran N, Lavoie H, Critton DA, Tebben A, Marinier A, Sicheri F, Therrien M. Crystal structure of a BRAF kinase domain monomer explains basis for allosteric regulation. Nature Structural & Molecular Biology. 2015; 22:37-43. DOI | PubMed
- Thiel KW, Carpenter G. Epidermal growth factor receptor juxtamembrane region regulates allosteric tyrosine kinase activation. PNAS. 2007; 104:19238-19243. DOI | PubMed
- van Lengerich B, Agnew C, Puchner EM, Huang B, Jura N. EGF and NRG induce phosphorylation of HER3/ERBB3 by EGFR using distinct oligomeric mechanisms. PNAS. 2017; 114:E2836-E2845. DOI | PubMed
- Wang L, Perera BG, Hari SB, Bhhatarai B, Backes BJ, Seeliger MA, Schürer SC, Oakes SA, Papa FR, Maly DJ. Divergent allosteric control of the IRE1α endoribonuclease using kinase inhibitors. Nature Chemical Biology. 2012; 8:982-989. DOI | PubMed
- Wilson TR, Fridlyand J, Yan Y, Penuel E, Burton L, Chan E, Peng J, Lin E, Wang Y, Sosman J, Ribas A, Li J, Moffat J, Sutherlin DP, Koeppen H, Merchant M, Neve R, Settleman J. Widespread potential for growth-factor-driven resistance to anticancer kinase inhibitors. Nature. 2012; 487:505-509. DOI | PubMed
- Wood ER, Truesdale AT, McDonald OB, Yuan D, Hassell A, Dickerson SH, Ellis B, Pennisi C, Horne E, Lackey K, Alligood KJ, Rusnak DW, Gilmer TM, Shewchuk L. A unique structure for epidermal growth factor receptor bound to GW572016 (Lapatinib): relationships among protein conformation, inhibitor off-rate, and receptor activity in tumor cells. Cancer Research. 2004; 64:6652-6659. DOI | PubMed
- Xia W, Petricoin EF, Zhao S, Liu L, Osada T, Cheng Q, Wulfkuhle JD, Gwin WR, Yang X, Gallagher RI, Bacus S, Lyerly HK, Spector NL. An heregulin-EGFR-HER3 autocrine signaling axis can mediate acquired lapatinib resistance in HER2+ breast cancer models. Breast Cancer Research. 2013; 15DOI | PubMed
- Xie T, Lim SM, Westover KD, Dodge ME, Ercan D, Ficarro SB, Udayakumar D, Gurbani D, Tae HS, Riddle SM, Sim T, Marto JA, Jänne PA, Crews CM, Gray NS. Pharmacological targeting of the pseudokinase Her3. Nature Chemical Biology. 2014; 10:1006-1012. DOI | PubMed
- Yang S, Raymond-Stintz MA, Ying W, Zhang J, Lidke DS, Steinberg SL, Williams L, Oliver JM, Wilson BS. Mapping ErbB receptors on breast cancer cell membranes during signal transduction. Journal of Cell Science. 2007; 120:2763-2773. DOI | PubMed
- Yarden Y, Pines G. The ERBB network: at last, cancer therapy meets systems biology. Nature Reviews Cancer. 2012; 12:553-563. DOI | PubMed
- Zeqiraj E, Filippi BM, Deak M, Alessi DR, van Aalten DM. Structure of the LKB1-STRAD-MO25 complex reveals an allosteric mechanism of kinase activation. Science. 2009a; 326:1707-1711. DOI | PubMed
- Zeqiraj E, Filippi BM, Goldie S, Navratilova I, Boudeau J, Deak M, Alessi DR, van Aalten DM. ATP and MO25alpha regulate the conformational state of the STRADalpha pseudokinase and activation of the LKB1 tumour suppressor. PLoS Biology. 2009b; 7DOI | PubMed
- Zhang X, Gureasko J, Shen K, Cole PA, Kuriyan J. An allosteric mechanism for activation of the kinase domain of epidermal growth factor receptor. Cell. 2006; 125:1137-1149. DOI | PubMed
- Zhang R, Fruhwirth GO, Coban O, Barrett JE, Burgoyne T, Lee SH, Simonson PD, Baday M, Kholodenko BN, Futter CE, Ng T, Selvin PR. Probing the heterogeneity of protein kinase activation in cells by super-resolution microscopy. ACS Nano. 2017; 11:249-257. DOI | PubMed
Fonte
Claus J, Patel G, Autore F, Colomba A, Weitsman G, et al. () Inhibitor-induced HER2-HER3 heterodimerisation promotes proliferation through a novel dimer interface. eLife 7e32271. https://doi.org/10.7554/eLife.32271





