Abstract
Introduzione
L’identificazione dei tratti che sono sotto selezione da parte degli ospiti è fondamentale per comprendere i processi che regolano le nascenti interazioni simbiotiche tra animali e microbi. La notevole efficienza con cui alcuni batteri evolvono la variazione che migliora l’accesso a nuove nicchie di ospiti indica che l’adattabilità può essere un attributo di alcuni genomi batterici. L’evoluzione adattativa verso una nuova nicchia, come ad esempio un nuovo ospite, può comportare la riconciliazione dei vincoli imposti dal contenuto genomico, dalla regolazione conflittuale e dalla pleiotropia(Morley et al., 2015; Bedhomme et al., 2012). In questo contesto, i regolatori globali potrebbero servire come obiettivi efficaci di selezione che guidano i salti adattivi fatti dai microbi patogeni o mutualistici, a condizione che le vie metaboliche essenziali siano entrambe sufficientemente isolate dagli effetti dannosi della mutazione e disponibili per l’integrazione con funzioni accessorie (Davenportet al., 2015; Wolfe et al. , 2004; Jansen et al., 2015 ). Gli studi che utilizzano l’evoluzione sperimentale hanno spesso rivelato che l’evoluzione adattativa procede inizialmente attraverso cambiamenti normativi, ma pochi hanno identificato i meccanismi sottostanti che promuovono l’adattamento o che collegano questi processi ai sistemi simbiotici naturali(Morley et al., 2015; Bedhomme et al., 2012; Kawecki et al., 2012; Marchetti et al., 2010; Guan et al., 2013).
I membri del genere Vibrio, batteri alofili con un’ampia distribuzione in ambienti marini e salmastri, si sono ripetutamente evoluti per colonizzare varie nicchie di ospiti(Nishiguchi, 2002; Takemura et al., 2014; Guerrero-Ferreira e Nishiguchi, 2007), e come tali, il loro studio può fornire una comprensione dell’adattabilità all’associazione ospite. Il Vibrio fischeri bioluminescente si trova tra il plancton marino(Lee e Ruby, 1992), ma la specie è nota soprattutto per la sua simbiosi mutualistica di organi leggeri con le specie di calamari e pesci. V. fischeri è anche noto per il suo comportamento di rilevamento del quorum sociale, per cui le comunità di batteri utilizzano molecole di segnale feromonico diffusibile per sincronizzare l’espressione genica in risposta alla densità cellulare(Schuster et al., 2013; Verma e Miyashiro, 2013; Waters e Bassler , 2005). Nel calamaro-simbiotico V. fischeri, il rilevamento del quorum avviene attraverso l’attivazione sequenziale di due diversi segnali feromonici: il primo segnale (C8-HSL) ‘primes’ percezione sensibile del secondo segnale (3-oxo-C6-HSL) attraverso una maggiore attività LitR, che aumenta i livelli del sensore feromonico LuxR, abbassando così la soglia per la percezione del segnale(Fidopiastis et al., 2002; Lupp e Ruby, 2004; Miyashiro et al., 2010). A sua volta, quando il LuxR si lega al 3-oxo-C6-HSL, il LuxR omodimerizza e attiva direttamente l’espressione dell’operone di bioluminescenza lux per produrre luce, che i calamari usano per mimetizzarsi in controilluminazione durante il loro comportamento notturno di foraggiamento(Lupp et al., 2003; Jones e Nishiguchi, 2004).
L’associazione simbiotica tra V. fischeri e il calamaro Euprymna scolopes è diventato un potente sistema per interrogare i meccanismi alla base della colonizzazione batterica delle superfici mucose degli ospiti metazoici dove i coloni devono superare le difese dell’ospite che limitano l’infezione da batteri non simbiotici, compresi gli agenti patogeni (Figura 1A). Una volta nati di recente i batteri intrappolano i calamari nel muco vicino all’organo di luce, i simbionti si aggregano in questo muco e, in risposta agli attrattori dell’ospite, successivamente nuotano attraverso i pori all’ingresso degli organi di luce nascenti (Nyholmet al., 2000). Mentre i batteri V. fischeri nuotano lungo i condotti e nelle cripte, si trovano ad affrontare un “guanto di sfida” di difese che include specie ossidative derivate dall’ospite(Davidson et al., 2004; Weis et al., 1996; Small e McFall-Ngai, 1999), oltre a pattugliare gli emociti macrofagi che si attaccano ad altre specie di batteri marini con maggiore affinità, uccidendo successivamente queste cellule invasori(Nyholm et al., 2009; Nyholm e McFall-Ngai, 1998; Koropatnick et al., 2007). Queste barriere garantiscono che solo il partner simbiotico corretto abbia accesso alle cripte dove i nutrienti forniti dall’ospite supportano la crescita batterica(Graf e Ruby, 1998; Heath-Heckman e McFall-Ngai, 2011). I parallelismi tra la colonizzazione benefica di V. fischeri e l’infezione patogena suggeriscono che le pressioni selettive esercitate dagli ospiti animali possono agire su un repertorio comune di tratti batterici utilizzati per aggirare gli ostacoli difensivi dell’ospite(Nyholm e McFall-Ngai, 2004).10.7554/eLife.24414.003Figure 1.Host meccanismi di selezione che modellano l’evoluzione adattiva di V. fischeri.(A) Vista dorsale dell’ospite giovanile E. scolopes (a sinistra) con riquadro che indica la posizione relativa dell’organo luminoso simbiotico ventrale. A destra, uno schema che illustra le fasi in cui avviene la selezione dell’ospite durante il calamaro-V. fischeri simbiosi: reclutamento dell’ospite (intrappolamento del muco, aggregazione ai pori degli organi leggeri), inizio della simbiosi (difese dell’ospite, compreso l’inghiottimento degli emociti e lo stress ossidativo), e colonizzazione e mantenimento (approvvigionamento di nutrienti, sanzionamento dei truffatori non luminosi, pattugliamento continuo degli emociti e spurgo quotidiano).(B) La crescita della popolazione simbionte modellato per un singolo passaggio sulla base delle dinamiche di crescita di V. fischeri ES114. Le popolazioni di organi leggeri sono iniziate con un minimo di ~ 10 cellule (Wollenberge Ruby, 2009; Altura et al., 2013) o fino all’1% dell’inoculo, ma sono ridotte del 95% in seguito alla ventilazione dell’organo leggero all’alba (ogni 24 ore) (Boettcheret al., 1996). Le zone d’ombra rappresentano i periodi notturni, mentre le zone di luce rappresentano la luce del giorno, che induce il comportamento di ventilazione.(C) Evoluzione sperimentale di V. fischeri sotto selezione dell’ospite come descritto in Schuster et al. (2010). Ogni popolazione ancestrale V. fischeri è stata preparata recuperando le cellule da cinque colonie, facendole crescere fino alla fase di mid-log, e sub-culturarle in 100 mL di acqua di mare filtrata ad una concentrazione sufficiente a colonizzare i calamari (≤ 20.000 CFU / ml). Il primo giorno, dieci calamari giovani non colonizzati (non luminosi) sono stati inoculati in comune da incubazione durante la notte, durante la quale i batteri sono stati sottoposti al primo collo di bottiglia host-selettivo. A seguito di ventilazione di ~ 95% della popolazione di organi leggeri, i calamari sono stati separati in lignaggi isolati in singoli pozzi di una piastra di 24 pozzetti di polistirolo contenente acqua di mare filtrata con file di calamari intervenuti da una coorte di controllo non inoculato, il controllo aposymbiotc (“controllo apo”). Si noti che solo due delle dieci popolazioni di calamari di passaggio sono mostrate. Nei giorni 2, 3 e 4, dopo lo sfiato, i calamari sono stati risciacquati e trasferiti in 2 mL di acqua di mare fresca filtrata. La luminescenza è stata misurata a vari intervalli per ogni calamaro per monitorare la colonizzazione e l’assenza di contaminazione nel controllo apossimbiotico calamaro. Il quarto giorno, il calamaro e metà del ventato sono stati congelati a -80°C per preservare i batteri, e il restante 1 mL di ventato è stato combinato con 1 mL di acqua di mare fresca filtrata, e utilizzato per inoculare un nuovo calamaro giovane non colonizzato di 24 ore. Il processo è continuato per 15 calamari solo per quei lignaggi in cui i calamari erano rilevabilmente luminosi a 48 ore dopo l’inoculazione.DOI:http://dx.doi.org/10.7554/eLife.24414.003
Non tutti i lignaggi di V. fischeri eccellono in simbiosi; questa osservazione riflette i vari regimi selettivi che modellano sia la variazione genetica che il potenziale adattivo come simbiotici (Leee Ruby, 1994a; Nishiguchi et al., 1998). Negli habitat in cui sono presenti ospiti calamari, essi influenzano le popolazioni locali di V. fischeri arricchendo la comunità planctonica con i ceppi più abili alla simbiosi (Lee e Ruby,1994b). I calamari reclutano piccole popolazioni di fondatori (~10 batteri) e le sottopongono a cicli giornalieri di espulsione (“venting”) e ricrescita del 95% delle popolazioni di organi leggeri a >105 batteri (Wollenberg e Ruby, 2009) (Figura 1B), aumentandocosì l’abbondanza relativa dei loro abitanti di organi leggeri nell’acqua di mare circostante (Lee e Ruby,1994b). Le strozzature all’interno del ciclo di ventilazione limitano la diversità microbica degli organi leggeri, comprese le variazioni che ostacolano la simbiosi, come i “truffatori” che non contribuiscono al mutualismo ma beneficiano dell’associazione simbiotica(Wollenberg e Ruby, 2009; Ruby e McFall-Ngai, 1999; Visick e McFall-Ngai, 2000). Tuttavia, la selezione imposta dall’ospite che spinge l’evoluzione di alcuni lignaggi verso una colonizzazione efficiente potrebbe ostacolare l’adattamento futuro e comportare compromessi di fitness in altri ambienti (Sotoet al., 2014; Caley e Munday, 2003). Così, al contrario, i ceppi planctonici V. fischeri che risiedono in habitat senza ospiti, o che non sono in grado di competere per le nicchie di ospiti primari, possono mantenere una maggiore adattabilità pur essendo inefficaci come simbionti (Takemura et al., 2014). La carenza nella colonizzazione dei calamari è correlata a una luminescenza insufficiente o eccessiva o a una produzione inadeguata di un polisaccaride simbiotico (noto come Syp), che è controllato da un attivatore acquisito orizzontalmente (RscS) nel ceppo nativo dei calamari ES114 (Nishiguchiet al., 1998; Yip et al., 2006; Mandel et al., 2009). Tuttavia, l’assenza del gene rscS in alcuni ceppi V. fischeri simbioticamente abile (e allo stesso modo, la presenza di rscS in ceppi carenti) indica che questo regolatore da solo non determina rigorosamente la capacità di colonizzazione dei calamari(Figura 1A, Figura 2-figure supplement 1). La somiglianza genomica tra ceppi strettamente correlati ma ecologicamente diversi ha oscurato le differenze funzionali rilevanti che a volte non sono rilevabili se non nel contesto simbiotico(Yip et al., 2006; Mandel et al., 2009; Travisano e Shaw , 2013).
Per questo studio, abbiamo condotto una serie di esperimenti evolutivi in cui i calamaro covacei selezionano tra le popolazioni di V. fischeri per i mutanti che sono in grado di avviare la simbiosi, di persistere nell’organo di luce e di colonizzare nuovi calamari quando vengono eliminati dall’organo di luce (Schusteret al., 2010). Questo ciclo di selezione dell’ospite è stato progettato per identificare i tratti alla base dell’evoluzione adattativa simbiotica e per rivelare le dinamiche evolutive e genomiche di questo processo. Abbiamo scelto come antenati dei nostri lignaggi sperimentali cinque ceppi V. fischeri che avevano attitudini variabili per la simbiosi dei calamari e che erano isolati da diverse nicchie, tra cui gli organi di luce dei calamari e dei pesci, e vari ambienti acquatici planctonici, tra cui uno senza ospiti noti (Tabella 1). Dopo aver sperimentalmente evoluto replicare popolazioni derivate da ogni antenato in parallelo, abbiamo valutato i cambiamenti genetici e fenotipici che si sono verificati sotto la selezione degli ospiti per esaminare come il fitness di partenza e la storia evolutiva passata ha influenzato l’adattabilità alla simbiosi calamaro. Per delineare gli effetti della selezione dell’ospite dall’accumulo di mutazioni neutre, abbiamo anche sottoposto V. fischeri all’evoluzione di laboratorio in mezzi di acqua di mare minima. In precedenza, abbiamo dimostrato che la luminescenza alterata era associata a diversi isolati dopo 15 passaggi seriali dell’ospite(Schuster et al., 2010). Qui riportiamo le basi genetiche di questo adattamento così come le dinamiche di popolazione dei simbionti in fase di selezione dell’ospite. È importante notare che identifichiamo anche i tratti precisi della selezione che hanno permesso a questi mutanti che hanno iniziato a spazzare via le barriere principali imposte dagli host.10.7554/eLife.24414.004Table 1.Strains e plasmidi utilizzati in questo studio.DOI:http://dx.doi.org/10.7554/eLife.24414.004StrainnomeDescrizione*Riferimento/fonteVibrio fischeri ceppi† ES114Isolatoda Euprymna scolopes(Boettchere Ruby, 1990)MJ11Isolato da Monocentris japonica light-organ(Haygoodet al., 1984)EM17Isolato da Euprymna morseii light-organ(Ruby e Lee, 1998)H905Isolato da plancton hawaiano(Lee e Ruby, 1992)WH1Isolato da plancton del Massachusetts(Lee, 1994)RF1A4MJ11 ∆binK::ermB; EmRQuesto studioRF1A5MJ11 ∆sypK::aphA1; KmRQuesto studioRF1A6MJ11 ∆binK: :ermB ∆sypK::aphA1; EmRKmRQuesto studioRF1A7MJ11 binK1 ∆sypK: :afA1; KmRQuesto studioMJ11EP2-3-2MJ11 binK4QuestostudioMJ11EP2-3-3-3MJ11 binK4QuestostudioMJ11EP2-3-4MJ11 binK4QuestostudioMJ11EP2-3-4MJ11 binK4Questo studioMJ11EP2-3-5MJ11 binK4QuestostudioMJ11EP2-3-6MJ11 binK4QuestostudioMJ11EP2-3-7MJ11 binK4QuestostudioMJ11EP2-3-8MJ11 binK4QuestostudioMJ11EP15-3-1MJ11 binK4QuestostudioMJ11EP15-3-3MJ11 binK4Questo studioMJ11EP15-3-3MJ11 binK4QuestostudioMJ11EP15-3-4MJ11 binK4QuestostudioMJ11EP15-3-7MJ11 binK4QuestostudioMJ11EP15-3-8MJ11 binK4QuestostudioMJ11EP2-4-1MJ11 binK1QuestostudioMJ11EP2-4-3MJ11 binK1QuestostudioMJ11EP2-4-4MJ11 binK1QuestostudioMJ11EP2-4-5MJ11 binK1QuestostudioMJ11EP2-4-6MJ11 binK1QuestostudioMJ11EP15-4-1MJ11 binK1 tadC1G593T(Schuster et al., 2010)MJ11EP15-4-6MJ11 binK1QuestostudioMJ11EP15-4-7MJ11 binK1QuestostudioMJ11EP15-4-8MJ11 binK1QuestostudioMJ11EP2-5-2MJ11 binK3QuestostudioMJ11EP2-5-3MJ11 binK3QuestostudioMJ11EP2-5-4MJ11 binK3Questo studioMJ11EP2-5-5-5MJ11 binK3QuestostudioMJ11EP2-5-5MJ11 binK3QuestostudioMJ11EP2-5-6MJ11 binK3QuestostudioMJ11EP15-5-2MJ11 binK4QuestostudioMJ11EP15-5-3MJ11 binK3QuestostudioMJ11EP15-5-4MJ11 binK3QuestostudioMJ11EP15-5-5-5MJ11 binK3Questo studioMJ11EP15-5-5MJ11 binK3QuestostudioMJ11EP2-6-1MJ11 binK2QuestostudioMJ11EP15-6-1MJ11 binK2(Schuster et al., 2010)MJ11EP15-6-2MJ11 binK2QuestostudioMJ11EP15-6-3MJ11 binK2QuestostudioMJ11EP15-6-4MJ11 binK2QuestostudioMJ11EP15-6-4MJ11 binK2Questo studioMJ11EP15-6-5MJ11 binK2QuestostudioMJ11CE4-1MJ11 fliAG80DQuesto studioMJ11CE5-1MJ11∆476QuestostudioNome dello strascicoDescrizione*Riferimento/fonteEscherichia coli ceppiDH5αF-recA1endA1hsdR17 supE44 thi-1 gyrA96relA1Δ(argF-lacZYA) U169φ80lacZΔM15λ– Gibco-BRLDH5αλpirsupE44 ΔlacU169(ϕlacZΔM15) recA1 endA1 hsdR17thi-1 gyrA96 relA1;λpir phage lysogen(Koltere Helinski, 1978)CC118λ pirΔ(arg-leu) araD ΔlacX74 galE galK phoA20thi-1 rpsErpoB argE(Am) recA1,lysogenized con λ pir dam dcm(Martín-Mora et al., 2016)NEB 10-betaΔ(ara-leu)7697araD139 fhuA Δ lacX74 galK16galK16 galE15 e14- Φ80dlacZΔ M15recA1relA1endA1 nupGrpsL(SmR) rph spoT1 Δ(mrr-hsdRMS-mcrBC)New England Biolabs, Ipswich, MATOP10F- mcrA Δ(mrr-hsdRMS-mcrBC) Φ80lacZΔM15Δ lacX74 recA1araD139Δ(ara-leu)7697 galU galKrpsL (SmR) endA1 nupGInvitrogen, Carlsbad,CAPlasmidspCR2.1-TOPOCommerciale vettore di clonazione; ApR KmRInvitrogen, Carlsbad, CApVSV105Mobilizable vettore; ChR(Dunn et al., 2006)pRAD2E1pVSV105 con binK wild-type; ChRQuesto studypRF2A2pVSV105 con binK1; ChRQuesto studypCLD48pVSV105 con ES114 sypE; ChR(Hussa et al.,2008)pRF2A3pVSVSV105 che trasporta il binA MJ11; ChRQuesto vettore di VSVSV104Mobilizable; KmR(Stabb and Ruby, 2002)pRF2A1pVSV104 che trasporta sypE; KmRQuesto vettore di VSVSV104 che trasporta il binA; KmRQuesto vettore di VVSV111Mobilizable che contiene gfp; ChR(Nyholm et al.., 2000)pRF2B7pCR2.1-TOPO contenente MJ11 ∆sypK::aph1 frammentoSOE; KmRQuesto studypVSV103Vettore mobilizzabile contenente lacZ ; KmR(Dunnet al.,2006)pCAW7B1pVSV103 contenente lacZ∆147-1080 bp; KmRQuesto studio*ApR, resistenza all’ampicillina; ChR, resistenza al cloramfenicolo; EmR, resistenza all’eritromicina; KmR, resistenza alla kanamicina; SmR, resistenza alla streptomicina.†I ceppi sperimentalmenteevoluti sono designati ‘MJ11EP#-#-#-#’, dove il primo e il secondo numero dopo la ‘P’ indica il passaggio dei calamari e la popolazione da cui il ceppo è stato isolato, e il terzo numero indica il numero isolato; i ceppi derivati dall’evoluzione in cultura sono designati ‘MJ11CE’.
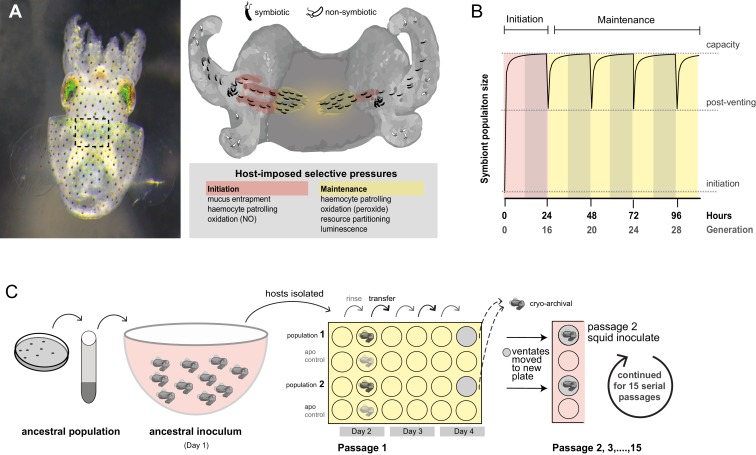
Figura 1.Meccanismi di selezione dell’ospite che modellano l’evoluzione adattiva di V. fischeri.(A) Vista dorsale dell’ospite giovanile E. scolopes (a sinistra) con riquadro che indica la posizione relativa dell’organo luminoso simbiotico ventrale. A destra, uno schema che illustra le fasi in cui avviene la selezione dell’ospite durante il calamaro-V. fischeri simbiosi: reclutamento dell’ospite (intrappolamento del muco, aggregazione ai pori degli organi leggeri), inizio della simbiosi (difese dell’ospite, compreso l’inghiottimento degli emociti e lo stress ossidativo), e colonizzazione e mantenimento (approvvigionamento di nutrienti, sanzionamento dei truffatori non luminosi, pattugliamento continuo degli emociti e spurgo quotidiano).(B) La crescita della popolazione simbionte modellato per un singolo passaggio sulla base delle dinamiche di crescita di V. fischeri ES114. Le popolazioni di organi leggeri sono iniziate con un minimo di ~ 10 cellule (Wollenberge Ruby, 2009; Altura et al., 2013) o fino all’1% dell’inoculo, ma sono ridotte del 95% in seguito alla ventilazione dell’organo leggero all’alba (ogni 24 ore) (Boettcheret al., 1996). Le zone d’ombra rappresentano i periodi notturni, mentre le zone di luce rappresentano la luce del giorno, che induce il comportamento di ventilazione.(C) Evoluzione sperimentale di V. fischeri sotto selezione dell’ospite come descritto in Schuster et al. (2010). Ogni popolazione ancestrale V. fischeri è stata preparata recuperando le cellule da cinque colonie, facendole crescere fino alla fase di mid-log, e sub-culturarle in 100 mL di acqua di mare filtrata ad una concentrazione sufficiente a colonizzare i calamari (≤ 20.000 CFU / ml). Il primo giorno, dieci calamari giovani non colonizzati (non luminosi) sono stati inoculati in comune da incubazione durante la notte, durante la quale i batteri sono stati sottoposti al primo collo di bottiglia host-selettivo. A seguito di ventilazione di ~ 95% della popolazione di organi leggeri, i calamari sono stati separati in lignaggi isolati in singoli pozzi di una piastra di 24 pozzetti di polistirolo contenente acqua di mare filtrata con file di calamari intervenuti da una coorte di controllo non inoculato, il controllo aposymbiotc (“controllo apo”). Si noti che solo due delle dieci popolazioni di calamari di passaggio sono mostrate. Nei giorni 2, 3 e 4, dopo lo sfiato, i calamari sono stati risciacquati e trasferiti in 2 mL di acqua di mare fresca filtrata. La luminescenza è stata misurata a vari intervalli per ogni calamaro per monitorare la colonizzazione e l’assenza di contaminazione nel controllo apossimbiotico calamaro. Il quarto giorno, il calamaro e metà del ventato sono stati congelati a -80°C per preservare i batteri, e il restante 1 mL di ventato è stato combinato con 1 mL di acqua di mare fresca filtrata, e utilizzato per inoculare un nuovo calamaro giovane non colonizzato di 24 ore. Il processo è continuato per 15 calamari solo per quei lignaggi in cui i calamari erano rilevabilmente luminosi a 48 ore dopo l’inoculazione.DOI:
http://dx.doi.org/10.7554/eLife.24414.003
Risultati
Calamaro evoluzione sperimentale sperimentale di ecologicamente diversificata V. fischeri ripetutamente prodotto mutazioni adattive nel gene della chinasi del sensore binK
Per studiare il processo dinamico di adattamento durante la simbiosi, abbiamo capitalizzato il processo di reclutamento naturale del calamaro per trovare popolazioni parallele di V. fischeri, e abbiamo utilizzato il comportamento quotidiano di sfogo del calamaro per limitare e ricrescere le popolazioni batteriche, che sono state passate attraverso 15 calamari in serie, che comprendono 60 eventi di bottlenecking e una stima di 290-360 generazioni (Figura 1C) (Schusteret al., 2010). Più popolazioni sono state derivate in parallelo da ciascuno dei cinque ceppi ancestrali utilizzando inoculi ad alta densità, fino a 10 volte la concentrazione necessaria per la colonizzazione del ceppo nativo, al fine di superare le carenze di colonizzazione dei ceppi mal adattati ai calamari(Figura 2A e Materiali e metodi).10.7554/eLife.24414.005Figure 2.Experimental evolution of Vibrio fischeri prodotto alleli multipli nel sensore chinasi BinK.(A). Relazione filogenetica, capacità simbiotica e mutazioni maturate durante l’evoluzione sperimentale dei ceppi di Vibrio fis cheri ecologicamente diversi. I rapporti di ceppo sono stati dedotti con la massima probabilità utilizzando genomi interi con RealPhy(Bertels et al., 2014) e con supporti dei nodi calcolati a partire da 1.000 bootstraps. I simboli grafici per le nicchie ecologiche rappresentano la fonte di isolamento. Le capacità simbiotiche intrinseche dei calamari dei cinque ceppi evoluti sperimentalmente, determinate dalla concentrazione minima di inoculo necessaria per una colonizzazione riuscita del 90% dei calamari con un inoculo di 3 ore (ES114, EM17 e WH1) o di notte (H905 e MJ11), sono rappresentate dallo spettro dei colori. Genomi di consenso per ciascuna delle popolazioni parallele V. fischeri evoluti attraverso E. scolopes sonomostrati a destra, con varianti indicate da cerchi. I dettagli della mutazione sono mostrati nella Tabella 2. Le mutazioni che sono state selezionate nelle popolazioni passate attraverso l’ospite hanno migliorato la capacità simbiotica piuttosto che il vigore generale.(B) BinK mutazioni BinK derivanti in popolazioni di calamari evoluti di MJ11 si è verificato nei domini HAMP e HATPaseC. Un modello strutturale omo-dimero per BinK che utilizza TMPRed e la modellazione ibrida del dominio della istidina chinasi (Anantharamane Aravind, 2000; Stewart e Chen, 2010) prevede che il dominio accessorio Cache1 sensoriale localizza al periplasma, mentre i restanti quattro domini funzionali (accessorio HAMP, e conservati HisKA, HATPaseC, e i domini phosphorelay REC) sono citoplasmatici (mostrati come banda grigia). Un’analisi della matrice di punteggio specifica della posizione (PSSM) per ciascuna delle posizioni BinK evolute del calamaro indica se un dato amminoacido è più (positivo) o meno (negativo) che può essere funzionalmente neutro. I punteggi per le sostituzioni effettuate in questi siti sono indicati in grassetto. Si prega di fare riferimento alla Figura 2-figure supplement 1 per una valutazione filogenetica dell’ortologia di BinK attraverso i ceppi Aliivibrio e V. fischeri.DOI:http://dx.doi.org/10.7554/eLife.24414.00510.7554/eLife.24414.006Figureintegratore a 2 cifre supplemento 1.BinK ortologia, domini conservati e calamari alleli binK adattati. (A)Unrooted maximum-likelihood (ML) filogenesisenza radici(ML) di tutti gli ibridi istidina chinasi identificati in V. genomi fis cheri. Le famiglie di geni sono state filogeneticamente annotate utilizzando, ove possibile, i riferimenti di Escherichia coli (non mostrati), altrimenti utilizzando il tag del locus ES114.DOI:http://dx.doi.org/10.7554/eLife.24414.006
Il sequenziamento del genoma degli isolati evoluti ha rivelato che, anche se poche mutazioni rilevabili sono emerse durante il passaggio dei calamari, la maggior parte delle mutazioni che sono emerse ad una frequenza rilevabile convergono in un gene conservato (locus VF_A0360 in V. fischeri ES114)(Figura 2A-B, Figura 2-figure supplement 1, Tabella 2) , che è stato recentemente identificato come una biofilminibizione chinasi (binK) nel ceppo nativo simbiotico ES114 (Brookse Mandel, 2016). Nove mutazioni indipendenti mappatura al locus binK, il più delle volte senza altre mutazioni che si ripetono, hanno dominato più popolazioni evolute parallele dei due ceppi inizialmente più compromesse in simbiosi con i calamari: MJ11 e H905(Figura 2A, Tabella 2). Dato che MJ11 è un pesce simbionte che manca di rscS, e H905 è un planctonico isolato dall’habitat dei calamari che è un povero colonizzatore di calamari nonostante la presenza di rscS, l’inizio dell’attività fisica ha meglio previsto il percorso evolutivo rispetto al contenuto di rscS o alla storia evolutiva passata, come dedotto sia dal lignaggio che dallo stile di vita(Figura 2A, Figura 2-figure supplement 1) (Mandel etal., 2009; Lee e Ruby, 1994a). Al contrario, pochissime mutazioni, tutte a loci unici, si sono verificate in isolati rappresentativi derivati da ceppi EM17 (un simbionte di calamaro Euprymna morsei ) e WH1 (un ceppo planctonico proveniente da un ambiente senza ospiti noti)(Figura 2A, Tabella 2). Entrambi questi ceppi hanno un fitness di partenza relativamente maggiore di MJ11 e H905, dimostrando ulteriormente che il fitness del simbionte di partenza influenza il suo percorso evolutivo(Wang et al., 2016). Infine, non sono state rilevate mutazioni in nessuno degli isolati rappresentativi del simbionte calamaro nativo ES114(Figura 2A, Tabella 2), anche se sono note diverse mutazioni che ne migliorano la dominanza competitiva(Fidopiastis et al., 2002; Brooks e Mandel, 2016). Laboratorio-cultura evoluzione del ceppo MJ11 che imitato le dinamiche di popolazione di colli di bottiglia indotta da calamari prodotto poche mutazioni ad eccezione di quelle localizzate a geni flagellari fliA e fliP(Tabella 2).10.7554/eLife.24414.007Tabella 2.Sommario delle mutazioni rilevate a seguito dell’evoluzione sperimentale di V. fischeri utilizzando Illumina genoma sequenziamento del genoma e sequenziamento Sanger mirati. Per le popolazioni evolute della cultura di V. fischeri MJ11, cinque isolati da ogni popolazione evoluta sono stati combinati per generare cinque metagenomi. Per le popolazioni evolute dei calamari di MJ11, EM17, WH1 e H905, i singoli isolati sono stati sequenziati da lignaggi che alla fine sono sopravvissuti a 15 passaggi dell’ospite. Gli isolati salvati dai primi punti temporali evolutivi (passaggio host 2) sono mostrati insieme ai genomi isolati dal punto finale (passaggio host 15). La profondità media di lettura e la copertura del genoma per gli isolati analizzati con WGS sono anche forniti.DOI:http://dx.doi.org/10.7554/eLife.24414.007AncestorEvolvedPassage (EP)PopulationIsolate† Mutazioni rilevate‡Statistiche di sequenziamento delle lucibinK(VFMJ11_A0397)tadC1(MJ11_0520) ; mutation (reads)Tutte le altre mutazioni rilevate dal gene WGS (locus); mutation (reads)Reads% Mapped to ancestorCoverageallele/mutationMethod (reads)§ ChIChIIMJ1121111binK3/S311LWGS(35)–375335299.5135.2118MJ11213binK3/S311LWGS(32)–371708899.6134.2113.5MJ111514binK3/S311LWGS(17)–171614499.546.842.5MJ11233binK4/N292TPCR/SSn.d.n.d.MJ11234binK4/N292TPCR/SSn.d.n.d.MJ11235binK4/N292TPCR/SSn.d.n.d.MJ11236binK4/N292TPCR/SSn.d.n.d.MJ11237binK4/N292TPCR/SSn.d.n.d.MJ11238binK4/N292TPCR/SSn.d.n.d.MJ111531binK4/N292TWGS(42)–303114998.9104.393.5MJ111533binK4/N292TWGS(63)–377771499.4114.6105.2MJ111534binK4/N292TWGS(42)–342021299.5106.497.1MJ111537binK4/N292TWGS(41)–330489199.590.382.5MJ111538binK4/N292TWGS(63)–294874399.685.581.2MJ11241binK1/R537CWGS(62)–2511256998478MJ11243binK1/R537CPCR/SSn.d.n.d.MJ11244binK1/R537CPCR/SSn.d.n.d.MJ11245binK1/R537CPCR/SSn.d.n.d.MJ11246binK1/R537CPCR/SSn.d.n.d.MJ11247binK1/R537CPCRn.d.n.d.MJ11248binK1/R537CPCRn.d.n.d.MJ11249binK1/R537CPCRn.d.n.d.MJ112410binK1/R537CPCRn.d.n.d.MJ112411binK1/R537CPCRn.d.n.d.MJ112412binK1/R537CPCRn.d.n.d.MJ112413binK1/R537CPCRn.d.n.d.MJ112414binK1/R537CPCR/SSn.d.n.d.MJ112415binK1/R537CPCR/SSn.d.n.d.MJ112416binK1/R537CPCR/SSn.d.n.d.MJ111541binK1/R537CWGS(131)G198V (85)-412614999.4117.8106.1MJ111546binK1/R537CWGS (61)G198V (55)-226682199.260.852.5MJ111547binK1/R537CWGS (89)G198V (93)-307443799.69283.6MJ111548binK1/R537CWGS (47)G198V (96)-290297799.58477.5MJ11252binK3/S311LWGS(26)–377104899.6132.4123.7MJ11253binK3/S311LWGS(46)–259551899.684.283.7MJ11254binK3/S311LWGS(20)–178571399.560.657.2MJ11255binK3/S311LWGS(62)–364134699.6117.4113.1MJ11256binK3/S311LWGS(81)–412875199.6141.1134.8MJ111552binK4/N292TWGS(89)–443082399.1152.3138.4MJ111553binK3/S311LWGS(10)–324858099.38881.1MJ111554binK3/S311LWGS(59)–360938299.5106.897.1MJ111555binK3/S311LWGS(28)–291557099.587.482.6MJ11261binK2/K482NWGS(104)–474856999.1164.6147MJ11262binK2/K482NPCR/SSn.d.n.d.MJ111561binK2/K482NWGS(75)–276491099.483.275.5MJ111562binK2/K482NWGS(63)–324096899.28872.6MJ111563binK2/K482NWGS(93)–381436799.5108.1101.7MJ111564binK2/K482NWGS(108)–371463899.5121.485.7MJ111565binK2/K482NWGS(90)–300636299.485.572MJ1115Culture1mg—1031929198272.8237.8MJ1115Culture3mg—749684798.2196.7195MJ1115Culture4mg– fliA (VF_1834); G80D(63 )289416098.376.667.4MJ1115Culture5mg– fliP (VF_1842); ∆1 @ 476/870nt (110)557143997.9148.5132.1MJ1115Culture2mg—541103298144.2129.4WH11541—727324498.6257.8251.1WH11542—214438199.661.465.1WH11543—226023299.662.166.6WH11544—234142899.761.665WH11551–NADH ossidasi (VF_A0027); A402T (62)173210699.560.864.7WH11552–NADH ossidasi (VF_A0027); A402T (61)173709599.461.964.9WH11553–NADH oxidase (VF_A0027); A402T (80)21948479660.863.4WH11554—219198699.861.964.9WH11561—925654799.3212.6220.3WH11562—213114499.66264.7WH11563—190885799.562.460.5EM171562—261160999.693.389.3EM171571—669013798.6225.8227.1EM171574—297742999.583.482.1EM171575– icmF (VF_0992);S171N, ( 72)241428899.571.671.5EM171581—317798199.597.594.6EM171582—313817599.592.492.3EM171583—281009999.581.280EM171585—523041199.6144.9143.2EM171591—802293599.4184.2173.5EM171592—334621699.6113.7106.9EM171593– gdh2 (VF_1284); E732D (72)348418899.595.793.2EM171595—244575899.572.872.6H9051511(∆37168 bp/25 geni)WGS (230)- IlvY (VF_2529); M25I (233)764550894.2250.4222.1H9051512(∆37168 bp/25 geni)WGS (167)- IlvY (VF_2529); M25I (112)353111496.8117.5104.4H9051513(∆37168 bp/25 geni)WGS (175)- IlvY (VF_2529);M25I (97)359668997122.3109.1H9051522∆16 bp@ 498/2595WGS (75)- fusa (VF_1572); N71T (60)281938797.691.479.6H9051524∆16 bp@ 498/2595WGS (94)- fusa (VF_1572);N71T (52)299297896.9103.391.4H9051525∆16 bp@ 498/2595WGS (90)- fusa (VF_1572); N71T (95)384483096.3123.6109H905231– tadF2 (VF_A0228); G21D (68)339361190.799.592.2H9051531– tadF2 (VF_A0228); G21D (140)797477391.5147.9143.9H9051532T195IWGS (65)- tadF2 (VF_A0228); G21D (28)198987595.565.458.2H9051533– tadF2 (VF_A0228); G21D (77)325389996.7103.894.4H9051534– tadF2 (VF_A0228); G21D (58)324274997.1103.394.7H9051535– tadF2 (VF_A0228); G21D (25)219077195.967.559H9051541E43*WGS (102)–665138592125.1130H9051543E43*WGS (111)–403237396.4135.9120.4H9051544E43*WGS (187)–612216895.8203.4179.4H9051545E43*WGS (90)–317781796.7100.890.6H9051551∆1 bp @ 2325/2595ntWGS (113)–716687090.4134.5130.9H9051552∆1 bp @ 2325/2595ntWGS (94)–370394696.7118.6108.3H9051553∆1 bp @ 2325/2595ntWGS (66)–282810297.498.690.4H9051554∆1 bp @ 2325/2595ntWGS (109)–472157597158.9143.8H905261T195IWGS (105)- tadF2 (VF_A0228); G21D (28)27436939483.373.6H9051563T195IWGS (142)- tadF2 (VF_A0228 ); G21D (49)559477197.5191.7175.3H9051564T195IWGS (105)- tadF2 (VF_A0228 ); G21D (37)336120696115.9101.4† I singoli nomi di raccolta dei ceppi caratterizzati assegnati agli isolati sono stati derivati dal loro lignaggio ancestrale (e.p.es. MJ11), dal passaggio evoluto (p.es. EP2), dalla popolazione (p.es. 1), e dal numero di isolati (p.es. 1), che nell’esempio precedente darebbero origine al nome di raccolta dei ceppi MJ11EP2-1-1-1. Isolati in grassetto servito come rappresentanti allelici binK per ulteriori saggi. mg: sequenziamento del metagenoma mettendo in comune cinque isolati da una popolazione.‡ Lapresenza di mutazioni è stata determinata da Illumina breve lettura (100PE) sequenziamento del genoma intero (WGS), da allele-specifica PCR (PCR), e / o da locus PCR-amplificazione PCR, seguita da Sanger sequenziamento (SS). ‘-‘ indica che non sono state identificate mutazioni in questo locus da breseq (Deatherage e Barrick, 2014) in questo isolato utilizzando il WGS. ‘n.d.’ indica che la presenza di mutazioni in questo locus non è stata determinata. § Viene fornito il numero di letture a supporto della chiamata della mutazione dai dati WGS. Le mutazioni sono state chiamate per siti con una copertura minima di 20 letture mappabili. Le mutazioni identificate dal sequenziamento Sanger (SS) degli ampliconi generati dalla PCR sono state confermate da allineamenti di entrambe le letture in avanti e indietro. Riferimento dei geni di codifica V. fischeri ES114 locus tags.
Per esaminare più a fondo il processo evolutivo che ha dato origine alle mutazioni binK convergenti, ci siamo concentrati su lignaggi derivati dal simbionte pesce MJ11 completamente sequenziato e relativamente ben caratterizzato. Solo cinque su dieci calamari esposti alla stessa popolazione di inoculo hanno trasmesso con successo i simbionti al secondo calamaro ricevente, e ogni lignaggio di successo ha ospitato le varianti binK(Tabella 2). Tra questi vi erano quattro alleli unici in cui le sostituzioni acquisite mappate a due dei cinque domini funzionali conservati della proteina BinK dedotta(Figura 2B, Tabella 2). Nonostante la variazione in piedi in binK attraverso i ceppi V. fischeri, le quattro mutazioni puntiformi nei lignaggi MJ11 sperimentalmente evoluti si sono verificati in posizioni che, con l’eccezione di binK3 (S311L), sono invarianti nei ceppi naturali e quindi sono suscettibili di rappresentare nuove varianti alleliche che non sono convergenti con il simbionte nativo (Figura 2B). Ulteriori analisi delle mutazioni acquisite utilizzando una matrice di punteggio specifica della posizione (PSSM) ha anche fornito la prova che le mutazioni in binK1 (R537C ), binK2 (K482N ) e binK3 (S311L ) influenzerebbero la funzione proteica (Figura 2B). In ciascuno dei cinque lignaggi di successo di MJ11, le varianti binK hanno dominato le popolazioni di organi leggeri da parte del terzo calamaro sperimentale(Tabella 2). Se le varianti benefiche in questo o qualsiasi altro locus erano tra le rimanenti cinque popolazioni di organi leggeri, il loro fallimento di colonizzare il secondo calamaro sperimentale ammontava a estinzione precoce di questi lignaggi.
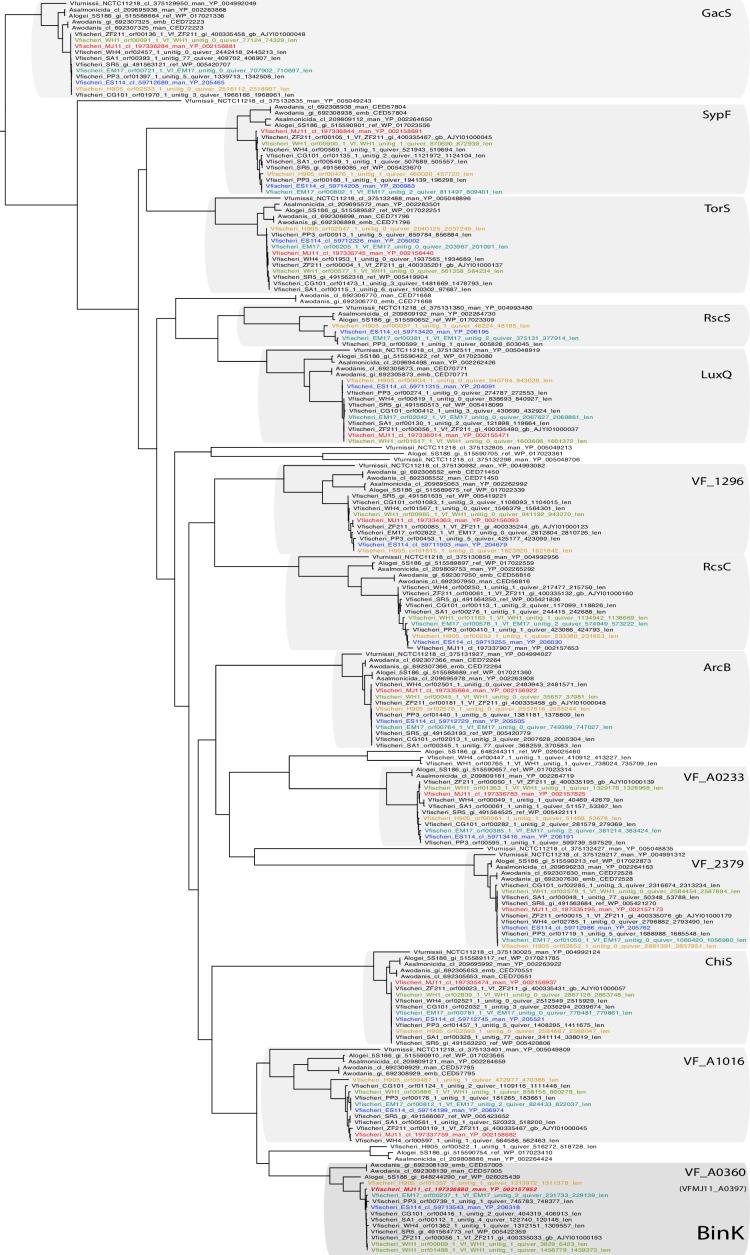
Figura 2-figure supplemento 1.Evoluzione sperimentale di Vibrio fischeri prodotto alleli multipli nel sensore chinasi BinK.BinK ortologia BinK.BinK, domini conservati e calamari adattati binK alleli.(A). Relazione filogenetica, capacità simbiotica, e mutazioni maturate durante l’evoluzione sperimentale dei calamari durante l’evoluzione sperimentale dei ceppi di Vibrio fischeri ecologicamente diversi. Le relazioni dei ceppi sono state dedotte con la massima probabilità utilizzando genomi interi con RealPhy(Bertels et al., 2014) e con supporti dei nodi calcolati a partire da 1.000 bootstraps. I simboli grafici per le nicchie ecologiche rappresentano la fonte di isolamento. Le capacità simbiotiche intrinseche dei calamari dei cinque ceppi evoluti sperimentalmente, determinate dalla concentrazione minima di inoculo necessaria per una colonizzazione riuscita del 90% dei calamari con un inoculo di 3 ore (ES114, EM17 e WH1) o di notte (H905 e MJ11), sono rappresentate dallo spettro dei colori. Genomi di consenso per ciascuna delle popolazioni parallele V. fischeri evoluti attraverso E. scolopes sonomostrati a destra, con varianti indicate da cerchi. I dettagli della mutazione sono mostrati nella Tabella 2. Le mutazioni che sono state selezionate nelle popolazioni passate attraverso l’ospite hanno migliorato la capacità simbiotica piuttosto che il vigore generale.(B) BinK mutazioni BinK derivanti in popolazioni di calamari evoluti di MJ11 si è verificato nei domini HAMP e HATPaseC. Un modello strutturale omo-dimero per BinK che utilizza TMPRed e la modellazione ibrida del dominio della istidina chinasi (Anantharamane Aravind, 2000; Stewart e Chen, 2010) prevede che il dominio accessorio Cache1 sensoriale localizza al periplasma, mentre i restanti quattro domini funzionali (accessorio HAMP, e conservati HisKA, HATPaseC, e i domini phosphorelay REC) sono citoplasmatici (mostrati come banda grigia). Un’analisi della matrice di punteggio specifica della posizione (PSSM) per ciascuna delle posizioni BinK evolute del calamaro indica se un dato amminoacido è più (positivo) o meno (negativo) che può essere funzionalmente neutro. I punteggi per le sostituzioni effettuate in questi siti sono indicati in grassetto. Si prega di fare riferimento alla Figura 2-figure supplement 1 per una valutazione filogenetica dell’ortologia di BinK attraverso i ceppi Aliivibrio e V. fischeri.DOI:
http://dx.doi.org/10.7554/eLife.24414.005(A) La filogenesi senza radici della massima probabilità (ML) di tutte le istidina chinasi ibride identificate nei genomi di V. fischeri. Le famiglie di geni sono state filogeneticamente annotate utilizzando, ove possibile, i riferimenti di Escherichia coli (non mostrati), altrimenti utilizzando il tag del locus ES114.DOI:
http://dx.doi.org/10.7554/eLife.24414.006

Figura 2-figure supplement 1.Ortologia BinK, domini conservati e alleli binK adattati ai calamari.(A) Unrooted maximum-likelihood (ML) filogenesi di tutti gli ibridi istidina chinasi identificati in V. fischeri genomi. Le famiglie di geni sono state filogeneticamente annotate utilizzando, ove possibile, i riferimenti di Escherichia coli (non mostrati), altrimenti utilizzando il tag del locus ES114.DOI:
http://dx.doi.org/10.7554/eLife.24414.006
Il grande vantaggio selettivo conferito dal binK adattato ai calamari ha migliorato la forma fisica sia durante la fase di avvio che durante la fase di mantenimento della simbiosi, in linea con le previsioni teoriche
Le ripetute spazzate di mutazioni binK romanzo che si sono verificati durante l’evoluzione dei calamari, ma non durante l’evoluzione della cultura di laboratorio, ha suggerito che le varianti binK erano calamari-adattabile (Tabella 2)(Dillon etal., 2017). Per valutare il contributo degli alleli binK evoluti specificamente per migliorare la colonizzazione simbiotica, abbiamo valutato l’efficienza di colonizzazione degli isolati evoluti dei calamari e dell’antenato utilizzando dosi di inoculo tipicamente utilizzate per il ceppo nativo simbionte ES114 (Figura 2A). Ogni variante binK evoluta dai calamari ha migliorato notevolmente l’efficienza di colonizzazione(Figura 3A), ma non erano significativamente più adatti alla coltura di laboratorio (il che sarebbe indicativo di mutanti che aumentano il vigore generale) rispetto all’ancestrale MJ11 (Figura 3B). Inoltre, mentre due delle cinque popolazioni di MJ11 evolute in cultura si sono evolute in mutazioni flagellari che hanno migliorato l’idoneità in cultura(Figure 2A e 3B, Tabella 2), nessuna mutazione binK maturata(Tabella 2) o migliorata come simbionti di calamari (Figura3A). Gli isolati evoluti che hanno mappato le mutazioni in diversi domini binK erano competitivamente indistinguibili l’uno dall’altro in termini di fitness simbiotico (test di permutazione t-test, p=0,348) (Figura 3-figure supplement 1), nonostante l’evidenza che l’allele binK1 (che codifica una sostituzione R537C, Figura 2B, Tabella 2) è apparso leggermente più efficiente nella colonizzazione dei calamari quando è stato inoculato singolarmente (Figura 3A).10.7554/eLife.24414.008Figure 3.Evolved binK alleles migliorata colonizzazione host e conferito un compromesso di fitness in ambienti non-host.(A) Efficienza di colonizzazione simbiotica di MJ11 e derivati in calamaro. Percentuale di calamari colonizzati da cultura evoluta (c1-c5) e calamari evoluti (binK1-binK4, isolati in grassetto nella Tabella 2) derivati di MJ11. Tre ore dopo una coorte di 10-20 calamari sono stati inoculati con 3000 CFU/mL di ogni ceppo MJ11, i calamari sono stati separati in fiale individuali, e percentuali di colonizzazione determinate dalla luminescenza rilevabile a 24 ore Bars: 95% CI.(B) Tassi di crescita di MJ11 e ceppi evoluti durante la competizione nella cultura di laboratorio. Tassi di crescita medi (parametri malthusiani realizzati) di ΔbinK, binK evoluti da calamari e mutanti flagellari evoluti in coltura ( varianti fliAe fliP, vedi Tabella 2) a seguito di una competizione di cultura in vitro in media minimi con binK ancestrale+ MJ11, stimato utilizzando i rendimenti CFU di ogni concorrente recuperati a intervalli regolari. Barre: 95% CI. La linea diagonale indica una crescita 1:1. Si prega di fare riferimento alla Figura 3-figure supplement 1 per i dati sulle capacità competitive di binK1 e binK3 durante la colonizzazione. Si prega di fare riferimento alla Figura 3-figure supplement 2 per i rendimenti simbiotici (CFU) dei ceppi ES114 e MJ11 dopo 24 e 48 ore.DOI:http://dx.doi.org/10.7554/eLife.24414.00810.7554/eLife.24414.009Figure3-figure supplement 1.Relative capacità competitiva delle varianti binK1 e binK3 per colonizzare i calamari. competizioni in vivo suggeriscono nessun vantaggio competitivo nella colonizzazione dei calamari tra evoluti V. fischeri MJ11 varianti MJ11 che trasportano mutazioni di dominio HAMP o HATPaseC. Gli indici competitivi relativi per le varianti binK1 e binK3 MJ11 (che portano mutazioni di dominio HATPaseC e HAMP, rispettivamente) utilizzati per co-inoculare i calamari attraverso una gamma di densità di inoculo. I punti sopra o sotto lo zero rappresentano gli organi leggeri dei calamari che sono dominati da bink3 o bink1 , rispettivamente.DOI:http://dx.doi.org/10.7554/eLife.24414.00910.7554/eLife.24414.010Figuresupplemento a 3 cifre 2.Crescita del ceppo ES114 e del ceppo MJ11 e delle sue varianti binK negli organi leggeri dei calamari 24 o 48 ore dopo l’inoculazione.Rendimenti di simbiotici determinati placcando diluizioni seriali di omogeneizzato di calamari come descritto in precedenza (Whistlere Ruby, 2003). Nota: l’asse Y è in scala log. Barre: 95% CI.DOI:http://dx.doi.org/10.7554/eLife.24414.010
Quantificare empiricamente il vantaggio selettivo (coefficiente selettivo: s) conferito da un allele binK rappresentativo che è sorto per la dominanza precoce prima delle mutazioni co-occorrenti, abbiamo co-inoculato il calamaro con MJ11 e basse densità di una variante binK1 ( un calamaro di secondo passaggio isolato completamente sequenziato che abbiamo chiamato MJ11EP2-4-1, vedi Tabelle 1 e 2), simulando le condizioni in cui prevediamo le varianti evolute dato il basso tasso di mutazione di V. fischeri(Dillonet al., 2017) (Figura 4A-B). Questi esperimenti hanno rivelato che anche a una frequenza estremamente bassa(ad esempio, una variante binK1 per 10.000 batteri MJ11 wild-type, che ammontava a soli 50 cellule variante binK1 in un inoculo 104 CFU-ml-1 per 10 calamari), la variante binK1 colonizzato calamari multipli (Figura 4-figuresupplemento 1). Il vantaggio selettivo stimato, basato sui rapporti dei tassi di crescita (una misura di competitività relativa) dei batteri di tipo selvatico e della variante binK1 nelle popolazioni di calamari colonizzati da organi leggeri, era indipendente dalle frequenze alleliche iniziali nell’inoculo, coerente con un modello di selezione dura (Figura 4B, Figura 4-figure supplement 1) (Saccherie Hanski, 2006). Il vantaggio selettivo stimato dell’allele binK1, che si adatta ai calamari, ha continuato ad aumentare di oltre il 60% tra le 24 e le 48 ore del calamaro (24 ore: 1,1; 48 ore: 1,8)(Figura 4B). Il vantaggio competitivo conferito da binK1 si è quindi esteso oltre gli eventi di colonizzazione iniziale (la “fase di iniziazione” durante le prime 24 ore) per includere il periodo di ricrescita competitiva dopo lo sfogo giornaliero del 95% della popolazione batterica (la “fase di mantenimento”), quando sono implicate diverse sanzioni di ospiti diversi (Figura 1A-B; Figura 4A-B). Al contrario, gli alleli binK adattativi ai calamari riducono l’idoneità rispetto al tipo selvaggio(binK+) in coltura planctonica di laboratorio (-0.18 > s > -1 ), dimostrando un modesto costo di fitness per alcuni alleli in assenza di ospiti (Figura 3B).10.7554/eLife.24414.011Figure 4.Stime empiriche e modellate del vantaggio selettivo nell’evoluzione delle popolazioni simbionti di V. fischeri.(A) Panoramica concettuale della dinamica delle popolazioni simbionti durante la crescita dell’inoculo e la successiva colonizzazione dell’ospite (linea nera), incluse le strozzature giornaliere imposte dall’ospite.(B) Confronto dei coefficienti di selezione conferiti da binK1 nel ceppo MJ11EP2-4-1 (che non ospita altre mutazioni) rispetto a binK+ da organi leggeri di calamari co-inoculati dopo 24 o 48 ore. Il vantaggio selettivo (cioè la competitività relativa) dell’allele evoluto è aumentato significativamente durante questo periodo da 1,1 a 1,8 (test di permutazione Fisher-Pitman, **p=0,0011). Ogni cerchio rappresenta il vantaggio selettivo di ogni deformazione misurata dai rapporti di deformazione recuperati in un singolo tratteggio. Si prega di fare riferimento alla Figura 4-figure supplement 1 per l ‘effetto delle frequenze di partenza del binK1 e delle densità dell’inoculo sulle stime del vantaggio selettivo.(C) Probabilità di sopravvivenza modellate per nuovi alleli benefici che sorgono in una crescente popolazione di simbionti che si trova ad affrontare colli di bottiglia imposti dall’ospite. Le curve sfumate grigie stimano la probabilità di sopravvivenza di nuovi mutanti a seguito del successivo collo di bottiglia della popolazione, che dipende sia dalla generazione di crescita dell’inoculo o dell’ospite in cui sorgono (asse delle x) sia dal vantaggio selettivo (s) conferito dalla mutazione (sfumatura grigia). In particolare, le varianti benefiche che si presentano all’inizio della coltura dell’inoculo sono suscettibili di sopravvivere all’estinzione al successivo collo di bottiglia, e questa probabilità di sopravvivenza diminuisce rapidamente anche quando conferisce un grande coefficiente selettivo. Sulla base di questo modello, ad esempio, una mutazione che conferisce un grande vantaggio selettivo(s~2) avrebbe meno del 10% di probabilità di sopravvivere al successivo collo di bottiglia di colonizzazione se si presentasse durante la decima generazione di crescita dell’inoculo (linea rossa).DOI:http://dx.doi.org/10.7554/eLife.24414.01110.7554/eLife.24414.012FigureSupplemento a 4 cifre 1.Stima del vantaggio selettivo dell’allele binK1 durante la colonizzazione dei calamari attraverso una gamma di frequenze di partenza e densità dell’inoculo.confronto dei coefficienti di selezione conferiti dal binK1 nel ceppo MJ11EP2-4-1 (‘Evo’) (che non contiene altre mutazioni) rispetto al binK+ (‘Anc’) da organi leggeri di calamari co-inoculati. Ogni punto rappresenta il vantaggio selettivo di ogni deformazione misurata dai rapporti di deformazione recuperati in un singolo tratteggio. Il vantaggio selettivo stimato conferito dall’allele binK1 evoluto non è stato influenzato dalla frequenza di partenza (A) (R2 = 0,025, pfrequenza = 0,62),ma è stato marginalmente influenzato dalla densità (B) (R2 = 0,025, pdensità = 0,03), basata su un’analisi di regressione multipla.DOI:http://dx.doi.org/10.7554/eLife.24414.012
Anche dato l’estremo vantaggio di fitness raggiunto dalla variante binK1 che cresce all’interno dei calamari(Figura 4B), il reclutamento ripetuto di varianti binK tra le poche cellule che hanno dato inizio alla simbiosi è notevole. Non solo le mutazioni devono conferire vantaggi eccezionali, ma queste rare varianti devono anche sopravvivere all’estinzione (cioè alla perdita della popolazione a causa della deriva genetica) durante i ripetuti collo di bottiglia imposti dall’ospite (Nyholme McFall-Ngai, 2004; Wollenberg e Ruby, 2009). Per esaminare come i tempi di mutazione, la forza del vantaggio selettivo e la dimensione della popolazione abbiano influenzato la capacità di rare varianti benefiche di raggiungere un’alta frequenza nelle popolazioni passate tra i calamari, abbiamo modellato le dinamiche evolutive e la probabilità di sopravvivenza delle singole varianti all’interno di una popolazione che sperimenta reclutamento, crescita e cicli ripetuti di colli di bottiglia all’interno di un singolo calamaro su una gamma teorica di coefficienti di selezione, applicando parametri generalizzati di popolazione e di crescita derivati dal ceppo nativo ES114 nella simbiosi calamaro-Vibrio (Wollenberge Ruby, 2009; Altura et al., 2013; Wahl e Gerrish, 2001) (vedi Materiali e metodi)(Figura 4C). Il modello prevede che, affinché la variazione benefica per garantire la sopravvivenza durante l’estremo collo di bottiglia imposto dall’ospite durante il reclutamento iniziale, i mutanti dovrebbero sorgere precocemente durante l’espansione della popolazione e conferire s ~6. Al contrario, le varianti benefiche che si presentano negli organi leggeri durante il mantenimento della simbiosi, che è caratterizzata da colli di bottiglia giornalieri di sfogo e ricrescita, hanno aumentato le probabilità di sopravvivenza anche se conferiscono un vantaggio selettivo inferiore, ma la probabilità della loro comparsa è ridotta a causa delle piccole dimensioni effettive della popolazione (Materiali e metodi e Figura 4C). Così, il modello suggerisce che i mutanti erano molto probabilmente presenti nell’inoculo di partenza e sono stati reclutati in simbiosi dai membri della prima coorte di calamari. Utilizzando una misura ad alta risoluzione del tasso di mutazione genomica V. fischeri ES114(Dillon et al., 2017), si prevede che ben 185 mutazioni individuali potrebbero essere sorti spontaneamente in binK (vedi Materiali e metodi) durante la crescita dell’inoculo (Figura 4A). Nonostante la loro bassa frequenza iniziale, qualsiasi nuovo allele che sia sorto dalla decima generazione di crescita dell’inoculo e che alla fine abbia conferito un elevato vantaggio selettivo nei calamari (cioè, s > 1 ) dovrebbe sopravvivere al primo passaggio dell’ospite per circa il 10% del tempo (Figura 4C, linearossa). Per inciso, la sopravvivenza osservata di ogni allele binK ammontava a 1 o 2 su 10 calamari sperimentali. Così, le stime empiriche del vantaggio selettivo conferito dal binK1 nell’ambiente simbiotico sono supportate da stime teoriche derivate da un modello di selezione straordinariamente forte durante i ripetuti colli di bottiglia(Wahl e Gerrish, 2001).
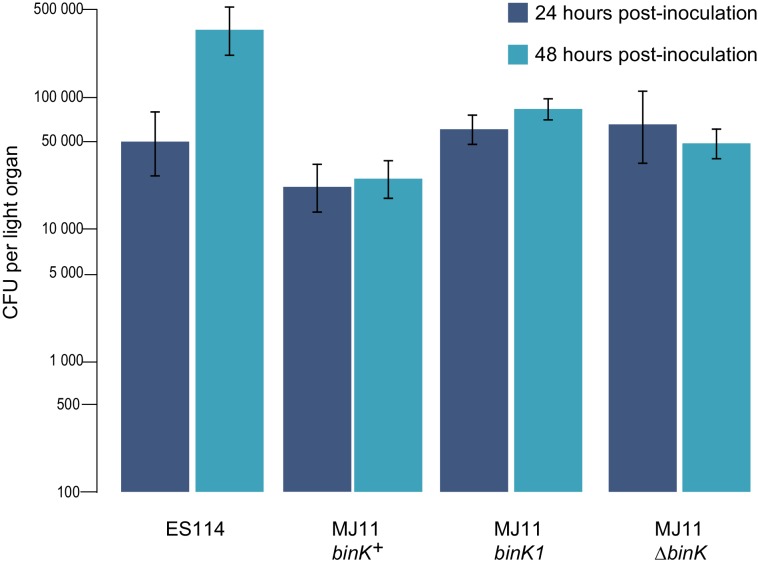
Figura 3-figure supplement 2.La crescita del ceppo ES114 e del ceppo MJ11 e le sue varianti binK negli organi leggeri dei calamari 24 o 48 ore dopo l’inoculazione.(A) Efficienza di colonizzazione simbiotica di MJ11 e derivati in calamari. Percentuale di calamaro colonizzato da cultura-evoluto (c1-c5) e calamaro-evoluto (binK1-binK4, isolati in grassetto nella tabella 2) derivati di MJ11. Tre ore dopo una coorte di 10-20 calamari sono stati inoculati con 3000 CFU/mL di ogni ceppo MJ11, i calamari sono stati separati in fiale individuali, e percentuali di colonizzazione determinate dalla luminescenza rilevabile a 24 ore Bars: 95% CI.(B) Tassi di crescita di MJ11 e ceppi evoluti durante la competizione nella cultura di laboratorio. Tassi di crescita medi (parametri malthusiani realizzati) di ΔbinK, binK evoluti dai calamari e mutanti flagellari evoluti dalla cultura ( varianti fliAe fliP, vedi Tabella 2) a seguito di una competizione di cultura in vitro in media minimi con binK ancestrale+ MJ11, stimato utilizzando i rendimenti CFU di ogni concorrente recuperati a intervalli regolari. Barre: 95% CI. La linea diagonale indica una crescita 1:1. Si prega di fare riferimento alla Figura 3-figure supplement 1 per i dati sulle capacità competitive di binK1 e binK3 durante la colonizzazione. Si prega di fare riferimento alla Figura 3-figure supplement 2 per i rendimenti simbiotici (CFU) dei ceppi ES114 e MJ11 dopo 24 e 48 ore.DOI:
http://dx.doi.org/10.7554/eLife.24414.008Le competizioni in vivo non suggeriscono alcun vantaggio competitivo nella colonizzazione dei calamari tra le varianti evolute V. fischeri MJ11 che portano mutazioni del dominio HAMP o HATPaseC. Gli indici competitivi relativi per le varianti binK1 e binK3 MJ11 (che portano mutazioni di dominio HATPaseC e HAMP, rispettivamente) utilizzati per co-inoculare i calamari attraverso una gamma di densità di inoculo. I punti sopra o sotto lo zero rappresentano gli organi leggeri dei calamari che sono dominati da bink3 o bink1 , rispettivamente.DOI:
http://dx.doi.org/10.7554/eLife.24414.009Rendimenti di simbiotici determinati placcando diluizioni seriali di omogeneizzato di calamari come descritto in precedenza(Whistler e Ruby, 2003). Nota: l’asse Y è in scala log. Barre: 95% CI.DOI:
http://dx.doi.org/10.7554/eLife.24414.010
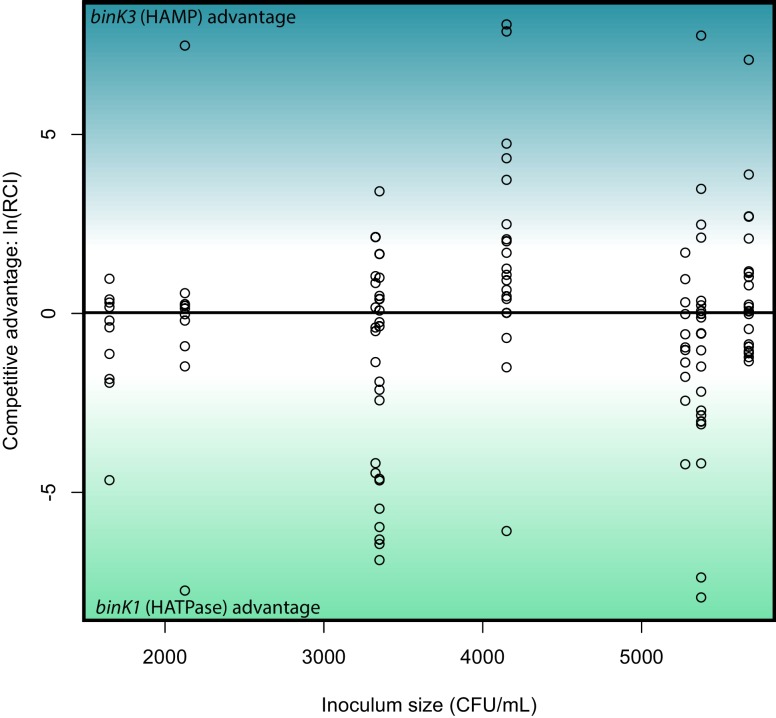
Figura 3-figure supplement 1.Capacità competitiva relativa delle varianti binK1 e binK3 di colonizzare i calamari.Le competizioni in vivo non suggeriscono alcun vantaggio competitivo nella colonizzazione dei calamari tra le varianti evolute V. fischeri MJ11 che portano mutazioni di dominio HAMP o HATPaseC. Indici competitivi relativi per le varianti binK1 e binK3 MJ11 (che portano mutazioni di dominio HATPaseC e HAMP, rispettivamente) utilizzati per co-inoculare i calamari attraverso una gamma di densità di inoculo. I punti sopra o sotto lo zero rappresentano gli organi leggeri dei calamari che sono dominati da bink3 o bink1 , rispettivamente.DOI:
http://dx.doi.org/10.7554/eLife.24414.009

Figura 3-figure supplement 2.Crescita del ceppo ES114 e del ceppo MJ11 e delle sue varianti binK negli organi leggeri dei calamari 24 o 48 ore dopo l’inoculazione.Rendimenti di simbiotici determinati placcando diluizioni seriali di omogeneizzato di calamari come descritto in precedenza(Whistler e Ruby, 2003). Nota: l’asse Y è in scala log. Barre: 95% CI.DOI:
http://dx.doi.org/10.7554/eLife.24414.010
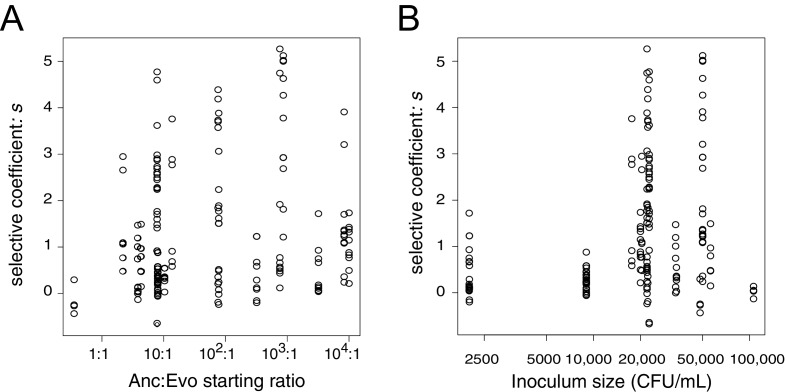
Figura 4-figure supplement 1.Stime empiriche e modellate del vantaggio selettivo nelle popolazioni di simbionti V. fischeri in evoluzione.stime del vantaggio selettivo dell’allele binK1 durante la colonizzazione dei calamari attraverso una gamma di frequenze di partenza e densità dell’inoculo.(A) Panoramica concettuale delle dinamiche della popolazione simbiotica durante la crescita dell’inoculo e la successiva colonizzazione dell’ospite (linea nera), comprese le strozzature giornaliere imposte dall’ospite.(B) Confronto dei coefficienti di selezione conferiti da binK1 nel ceppo MJ11EP2-4-1 (che non ospita altre mutazioni) rispetto a binK+ da organi leggeri di calamari co-inoculati dopo 24 o 48 ore. Il vantaggio selettivo (cioè la competitività relativa) dell’allele evoluto è aumentato significativamente durante questo periodo da 1,1 a 1,8 (test di permutazione Fisher-Pitman, **p=0,0011). Ogni cerchio rappresenta il vantaggio selettivo di ogni deformazione misurata dai rapporti di deformazione recuperati in un singolo tratteggio. Si prega di fare riferimento alla Figura 4-figure supplement 1 per l ‘effetto delle frequenze di partenza del binK1 e delle densità dell’inoculo sulle stime del vantaggio selettivo.(C) Probabilità di sopravvivenza modellate per nuovi alleli benefici che sorgono in una crescente popolazione di simbionti che si trova ad affrontare colli di bottiglia imposti dall’ospite. Le curve sfumate grigie stimano la probabilità di sopravvivenza di nuovi mutanti a seguito del successivo collo di bottiglia della popolazione, che dipende sia dalla generazione di crescita dell’inoculo o dell’ospite in cui sorgono (asse delle x) sia dal vantaggio selettivo (s) conferito dalla mutazione (sfumatura grigia). In particolare, le varianti benefiche che si presentano all’inizio della coltura dell’inoculo sono suscettibili di sopravvivere all’estinzione al successivo collo di bottiglia, e questa probabilità di sopravvivenza diminuisce rapidamente anche quando conferisce un grande coefficiente selettivo. Sulla base di questo modello, ad esempio, una mutazione che conferisce un grande vantaggio selettivo(s~2) avrebbe meno del 10% di probabilità di sopravvivere al successivo collo di bottiglia di colonizzazione se si presentasse durante la decima generazione di crescita dell’inoculo (linea rossa).DOI:
http://dx.doi.org/10.7554/eLife.24414.011Confronto dei coefficienti di selezione conferiti dal binK1 nel ceppo MJ11EP2-4-1 (‘Evo’) (che non contiene altre mutazioni) rispetto al binK+ (“Anc”) da organi leggeri di calamari co-inoculati. Ogni punto rappresenta il vantaggio selettivo di ogni deformazione misurata dai rapporti di deformazione recuperati in un singolo tratteggio. Il vantaggio selettivo stimato conferito dall’allele binK1 evoluto non è stato influenzato dalla frequenza di partenza (A) (R2 = 0,025, pfrequenza = 0,62),ma è stato marginalmente influenzato dalla densità (B) (R2 = 0,025, pdensità = 0,03), basata su un’analisi di regressione multipla.DOI:
http://dx.doi.org/10.7554/eLife.24414.012

Figura 4-figure supplement 1.Stime del vantaggio selettivo dell’allele binK1 durante la colonizzazione dei calamari attraverso una gamma di frequenze di partenza e densità dell’inoculo.Confronto dei coefficienti di selezione conferiti dal binK1 nella deformazione MJ11EP2-4-1 (“Evo”) (che non contiene altre mutazioni) rispetto al binK+ (“Anc”) da organi leggeri di calamari co-inoculati. Ogni punto rappresenta il vantaggio selettivo di ogni deformazione misurata dai rapporti di deformazione recuperati in un singolo tratteggio. Il vantaggio selettivo stimato conferito dall’allele binK1 evoluto non è stato influenzato dalla frequenza di partenza (A) (R2 = 0,025, pfrequenza = 0,62),ma è stato marginalmente influenzato dalla densità (B) (R2 = 0,025, pdensità = 0,03), basata su un’analisi di regressione multipla.DOI:
http://dx.doi.org/10.7554/eLife.24414.012
Il binK adattato all’ospite ha migliorato il comportamento di colonizzazione precoce, la sopravvivenza all’ossidazione e l’evasione dell’immunità dell’ospite attraverso una maggiore produzione di matrici associate alle cellule
Il sostanziale guadagno di fitness conferito dall’allele binK1 entro le prime 24 ore di colonizzazione(Figura 4B) ha suggerito che esso ha migliorato i comportamenti di colonizzazione precoce di MJ11 (Figura 1Ae B) (Nyholm e McFall-Ngai, 2004). Syp media l’aggregazione del ceppo nativo ES114 nel muco del calamaro e la sua sovrapproduzione aumenta la capacità di aggregazione di questo stesso ceppo (Brookse Mandel, 2016; Nyholm e McFall-Ngai, 2003; Shibata et al., 2012). Pertanto, abbiamo valutato se l’aggregazione della variante binK1 evoluta dai calamari è stata modificata. binK1 ha migliorato l’aggregazione all’ingresso degli organi di luce rispetto al wild-type MJ11 (Figura 5A,Figura 5-figure supplement 1). Al contrario, non ha causato il raggrinzimento delle colonie (dati non mostrati), un proxy per la produzione di biofilm Syp-mediata dal ceppo ES114(Brooks e Mandel, 2016; Shibata et al., 2012). Tuttavia, il binK1 ha aumentato notevolmente la produzione di biofilm in vitro rispetto al MJ11, come determinato dall’aderenza superficiale (Figura 5B), forse riflettendo la presenza di matrici di biofilm più complesse come la cellulosa la cui espressione è stata potenziata dalle mutazioni bink1 e ∆binK (Figura 5-figuresupplement 2, Appendice 1)(Shibata et al.,2012; Darnell et al. , 2008; Bassis e Visick , 2010). Per indagare le basi dell’aumento della formazione di biofilm da parte della variante binK1, abbiamo sovraespresso i geni che codificano un repressore di Syp, sypE (Morrise Visick, 2013), e di cellulosa, binA (Figura 5-figure supplement 3) (Bassise Visick, 2010). Ogni regolatore ha abolito il fenotipo migliorato del biofilm della variante binK1, indicando che entrambi i substrati della matrice hanno contribuito a questo tratto (Figura 5B). Per testare direttamente il ruolo del Syp, abbiamo anche introdotto una mutazione ∆sypK, che elimina funzionalmente la produzione di biofilm Syp dal ceppo ES114 (Shibataet al., 2012). La mutazione ha ridotto il biofilm dalla variante binK1, indicando che il miglioramento della produzione di biofilm della variante ha coinvolto la produzione di Syp (Figura 5B).10.7554/eLife.24414.013Figure 5.Host-adapted binK1 ha migliorato i fenotipi di inizio attraverso il miglioramento del biofilm.(A) V. fischeriMJ11 formazione di aggregati vicino ai condotti degli organi leggeri. Tessuto ospite colorato con CellTracker Orange. I simbionti portano plasmidi GFP (pKV111) (Nyholmet al., 2000). Le micrografie mostrano aggregati rappresentativi di V. fischeri a seguito della dissezione di 30 animali appena nati incubati con ogni ceppo. Gli aggregati sono stati visualizzati tra 2 e 3 ore dopo l’inoculazione con un microscopio confocale a scansione laser Zeiss LSM 510 Meta. Si prega di fare riferimento alla Figura 5-figure supplemento 1 per ulteriori visualizzazioni di formazione di aggregati.(B) Produzione di biofilm (colorazione violetta di cristallo rispetto a MJ11) da wild-type MJ11(binK+), varianti binK1 e ∆binK per calamari e varianti ∆binK in presenza di vettore vuoto (EV, pVSV105) (riempimento bianco), Syp biofilm repressor sypE ( pCLD48 ) (riempimento tratteggiato), o cellulosa repressor binA (pRF2A3 ) (riempimento grigio). n = 12-16 repliche biologiche. Vedere la Figura 5-figure supplement 2 per la prova dell’aumento di cellulosa nelle varianti binK, e la Figura 5-figure supplement 3 per lo schema del biofilm repressor repressor. Seguito dall’influenza di una delezione sypK sulla produzione di biofilm di varianti MJ11 e binK. n = 10 repliche biologiche.(C) Media binomiale di sopravvivenza a seguito di esposizione al perossido di idrogeno di tipo selvaggio MJ11(binK+), varianti binK1 e ∆binK per i calamari in presenza di vettori vuoti (EV, pVSV105) (riempimento bianco), sypE ( pCLD48 ) (riempimento tratteggiato), o binA ( pRF2A3 ) (riempimento grigio). n = 20-50 repliche biologiche. Seguito dall’influenza di una delezione sypK (sovrapposizione di linee diagonali) sulla sopravvivenza della popolazione delle varianti MJ11 e binK (riempimento a colori). n = 15-106 repliche biologiche. Barre di errore 95% CI. Valori p significativi (p<0,05) sono indicati sopra ogni confronto. *p<2.2e-16. Sebbene gli effetti della sovraespressione di binA e della delezione di sypK sulla resistenza ossidativa nella variante ∆binK abbiano seguito le stesse tendenze di questi geni in binK1 , le riduzioni sono state solo marginalmente significative (p=0,051 e 0,15, rispettivamente). Si prega di fare riferimento alla Figura 5-figure supplement 2 per l’ evidenza trascrittomica della riduzione dell’espressione di due loci di cellulosa nel ∆binK mutante. Uno schema dell’impatto dei repressori BinA e SypE sui substrati biofilm è disponibile come Figura 5-figure supplemento 3.DOI:http://dx.doi.org/10.7554/eLife.24414.01310.7554/eLife.24414.014Figure5-figure supplemento 1.In vivo cambiamenti comportamentali di aggregazione conferite da evoluto variante binK1. (A-B) Aggregazione di ancestrale (A) e si è evoluto (B) MJ11 su epitelio mucosale ospite prima della colonizzazione. Tessuto ospite colorato con CellTracker Orange. I simbionti portano plasmidi GFP (pKV111) (Nyholmet al., 2000). Le micrografie mostrano aggregati rappresentativi di V. fischeri a seguito della dissezione di 30 animali appena nati incubati con ogni ceppo. Gli aggregati sono stati visualizzati tra 2 e 3 ore dopo l’inoculazione con un microscopio confocale a scansione laser Zeiss LSM 510 Meta. Barre di scala: 24 μm.DOI:http://dx.doi.org/10.7554/eLife.24414.01410.7554/eLife.24414.015FigureSupplemento a 5 cifre 2.Spostamenti trascrizionali associati alle varianti binK.Differenze trascriptomiche tra i tipi wild-type MJ11 (binK+), MJ11 binK1 adattato ai calamari, e MJ11 ∆binK per i loci di codifica nel genoma MJ11 come determinato da RNA-Seq. Le varianti sono state campionate durante la crescita precoce dei log (OD600 ~ 0,25) in rich media (SWTO) prima della produzione di biofilm rilevabile da quattro repliche biologiche per ogni ceppo. Il verde indica un aumento dell’espressione; il rosso indica un’espressione ridotta rispetto all’espressione media per locus (cioè, conteggi letti in scala z rispetto alla media logCPM). La mappa termica mostra solo i loci per i quali l’espressione media in una variante binK differisce da quella in MJ11 con una soglia di significatività FDR di 0,05 (Tabella 3). Le etichette colorate si riferiscono a composti il cui metabolismo, trasporto o sintesi sono influenzati dall’espressione di questi geni. I geni coinvolti nella sintesi della cellulosa sono indicati con le frecce (VFMJ11_A1000- proteina C dell’operone della sintasi della cellulosa, e VFMJ11_A1007- proteina YhjU dell’operone della sintasi della cellulosa) e il log fold change (logFC) relativo al wild-type è indicato per binK1 e poi ∆binK.DOI:http://dx.doi.org/10.7554/eLife.24414.01510.7554/eLife.24414.016FigureSupplemento a 5 cifre 3.Schema di regolazione da parte dei biofilm repressori SypE e BinA.SypE reprime la produzione di biofilm Syp dopo la trascrizione (Morrise Visick, 2013). BinA reprime la cellulosa, ma non il Syp, la formazione di biofilm aumentando l’attività della fosfodiesterasi (Bassise Visick, 2010). Le linee nere indicano una regolazione negativa. Le frecce grigie indicano trascrizione/traslazione. Si noti che, poiché il binA è espresso da un promotore del locus syp, l’attivazione del locus syp che porta alla produzione di Syp porta anche alla repressione della cellulosa.DOI:http://dx.doi.org/10.7554/eLife.24414.016
Anche se l’aumento dell’aggregazione potrebbe conferire un guadagno in termini di fitness alle varianti binK durante la fase di avvio della simbiosi, l’aggregazione è un tratto abbastanza variabile da mettere in discussione la possibilità di spiegare la dominanza delle varianti binK. Una migliore aggregazione da sola non causerebbe l’aumento del 60% di fitness osservato durante il mantenimento della simbiosi(Figura 4B, Figura 3-figure supplement 2). Inoltre, a quanto ci risulta, nessuno studio ha ancora valutato se il biofilm conferisca una forma fisica simbiotica al di là dell’aggregazione. A causa del potenziale che il biofilm potrebbe conferire la sopravvivenza di fronte agli insulti ambientali, abbiamo valutato se il binK1 ha influito sulla sopravvivenza all’esposizione al perossido, in quanto l’ossidazione è tra l’arsenale difensivo dell’ospite (Small e McFall-Ngai, 1999; Visick e Ruby, 1998) (Figura 1A). Le varianti binK1 e ∆binK sono sopravvissute all’ossidazione meglio di MJ11, e la sovraespressione del syp repressor sypE o del repressor binA della cellulosa ha diminuito la sopravvivenza (Figura 5C). Anche la soppressione del sypK nelle varianti binK ha ridotto ulteriormente la sopravvivenza, sostenendo la conclusione che la produzione di Syp conferisce resistenza all’ossidazione (Figura 5C). L’aumento della produzione di biofilm e la sopravvivenza dopo l’esposizione al perossido sono correlati, suggerendo che il Syp e il biofilm di cellulosa contribuiscono alla resistenza all’ossidazione conferita dalle varianti binK .
Durante la migrazione e al raggiungimento dell’organo luminoso del calamaro, i potenziali simbionti devono confrontarsi con gli emociti fagocitici dell’ospite, simili a macrofagi, che legano, inghiottono e distruggono i batteri(Figura 1A)(Nyholm e McFall-Ngai, 1998). La capacità degli emociti di calamari di legarsi preferibilmente a specie batteriche non simbiotiche è ben stabilita, ma il riconoscimento differenziale tra V. fischeri è stato riportato solo per il ceppo nativo ES114 e le sue varianti genetiche (Nyholmet al., 2009). Pertanto, abbiamo valutato se gli emociti di calamaro bersagliano preferibilmente gli emociti di MJ11 non simbiotici e se l’alterata capacità del biofilm conferita dal binK1 ha favorito l’evasione del sistema immunitario innato dell’ospite (Figura 6, Figura 6-figure supplement 1). Gli emociti dei calamari giovanili hanno legato l’MJ11 wild-type MJ11 in misura maggiore rispetto al ceppo nativo ES114, e questo legame era paragonabile a quello osservato con altre specie di batteri, come V. harveyi(Figura 6). Al contrario, la variante binK1 ha resistito al legame degli emociti dell’ospite ad un livello paragonabile al ceppo nativo dei calamari ES114(Figura 6). La sovraespressione di sypE o binA ha ridotto l’evasione immunitaria da parte di ES114, e sypE ha anche ridotto in modo significativo l’evasione immunitaria da parte della variante binK1, che dimostra che la produzione di Syp e di matrici extracellulari di cellulosa hanno mediato questo tratto. Questi risultati forniscono la prima prova sperimentale che la produzione di Syp e di cellulosa da parte di ceppi di V. fischeri nativi e non nativi contribuisce alla risposta degli emociti ospiti. Inoltre, questi risultati dimostrano che, alterando la produzione del substrato del biofilm, binK1 potrebbe migliorare la sopravvivenza di MJ11 durante i punti di controllo selettivi imposti dall’ospite.10.7554/eLife.24414.017Figure 6.Biofilm produzione di biofilm da varianti binK1 calamaretta-adattabile media l’evasione degli emociti. (A) Efficienza relativa del legame degli emociti di calamaro di ceppi V. fischeri etichettati GFP tra cui: simbionte nativo di calamari ES114, binK+ MJ11, ∆binKMJ11 (RF1A4), binK1 MJ11, e l’agente patogeno dei molluschi V. harveyi B392.(B) Efficienza relativa del legame degli emociti di calamari del simbionte nativo dei calamari ES114 e del bink1 MJ11 adattato ai calamari che trasporta il vettore vuoto (pVSV104), sypE (pRF2A1) o binA (pRF2A4). N = 30-52 emociti quantificati per ceppo. Barre di errore: 95% CI. I valori p significativi (p<0,05) sono indicati sopra ogni confronto. Si prega di fare riferimento alla Figura 6-figure supplement 1 per le micrografie delle interazioni tra Vibrio-emocita.DOI:http://dx.doi.org/10.7554/eLife.24414.01710.7554/eLife.24414.018Figure 6-figure supplement 1.Risposta in vitro degli emociti di calamaro a Vibrio selvaggio, evoluto da calamaro e mutante .Le micrografie mostrano esempi di cellule non simbiotiche legate ad emociti (A:Vibrio harveyi), calamaro-simbiotico (B: V. fischeri ES114 ), calamaro-naive (C: V. fischeri MJ11 binK+ ) e calamaro-adattato (D: MJ11 binK1 ). Il numero medio di cellule Vibrio marcate GFP legate da emociti è stato quantificato rispetto alla carica batterica totale in un raggio di 60 µm utilizzando la microscopia confocale con ingrandimento 63X, dopo un’ora di esposizione batterica. Emociti di calamari in rosso (CellTracker Orange), Vibrio in verde (GFP). Barre di scala: 12 μm.DOI:http://dx.doi.org/10.7554/eLife.24414.018
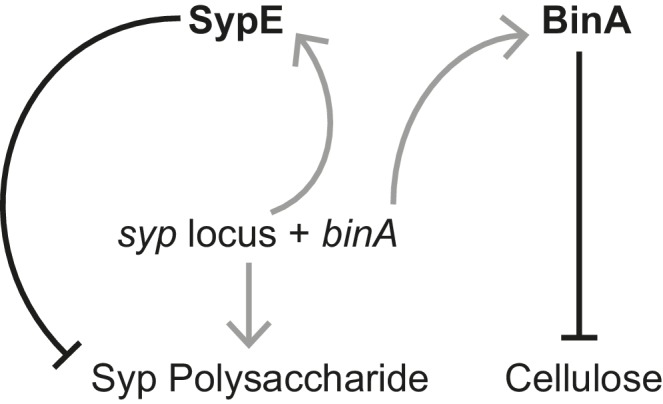
Figura 5-figure supplement 3.Figura 5— figura 3. Fenotipi di iniziazione migliorata binK1 adattata all’ospite attraverso il miglioramento del biofilm.cambiamenti comportamentali di aggregazione in vivo conferita da evoluti binK1 variante.spostamenti trascrizionali associati con varianti binK.schema di regolazione da parte dei repressori biofilm SypE e BinA.(A) V. fischeri MJ11 formazione di aggregati in prossimità di condotti di organi leggeri. Tessuto ospite colorato con CellTracker Orange. I simbionti portano plasmidi GFP (pKV111) (Nyholmet al., 2000). Le micrografie mostrano aggregati rappresentativi di V. fischeri a seguito della dissezione di 30 animali appena nati incubati con ogni ceppo. Gli aggregati sono stati visualizzati tra 2 e 3 ore dopo l’inoculazione con un microscopio confocale a scansione laser Zeiss LSM 510 Meta. Si prega di fare riferimento alla Figura 5-figure supplemento 1 per ulteriori visualizzazioni di formazione di aggregati.(B) Produzione di biofilm (colorazione violetta di cristallo rispetto a MJ11) da wild-type MJ11(binK+), varianti binK1 e ∆binK per calamari e varianti ∆binK in presenza di vettore vuoto (EV, pVSV105) (riempimento bianco), Syp biofilm repressor sypE ( pCLD48 ) (riempimento tratteggiato), o cellulosa repressor binA (pRF2A3 ) (riempimento grigio). n = 12-16 repliche biologiche. Vedere la Figura 5-figure supplement 2 per la prova dell’aumento di cellulosa nelle varianti binK, e la Figura 5-figure supplement 3 per lo schema del biofilm repressor repressor. Seguito dall’influenza di una delezione sypK sulla produzione di biofilm di varianti MJ11 e binK. n = 10 repliche biologiche.(C) Media binomiale di sopravvivenza a seguito di esposizione al perossido di idrogeno di tipo selvaggio MJ11(binK+), varianti binK1 e ∆binK per i calamari in presenza di vettori vuoti (EV, pVSV105) (riempimento bianco), sypE ( pCLD48 ) (riempimento tratteggiato), o binA ( pRF2A3 ) (riempimento grigio). n = 20-50 repliche biologiche. Seguito dall’influenza di una delezione sypK (sovrapposizione di linee diagonali) sulla sopravvivenza della popolazione delle varianti MJ11 e binK (riempimento a colori). n = 15-106 repliche biologiche. Barre di errore 95% CI. Valori p significativi (p<0,05) sono indicati sopra ogni confronto. *p<2.2e-16. Sebbene gli effetti della sovraespressione di binA e della delezione di sypK sulla resistenza ossidativa nella variante ∆binK abbiano seguito le stesse tendenze di questi geni in binK1 , le riduzioni sono state solo marginalmente significative (p=0,051 e 0,15, rispettivamente). Si prega di fare riferimento alla Figura 5-figure supplement 2 per l’ evidenza trascrittomica della riduzione dell’espressione di due loci di cellulosa nel ∆binK mutante. Uno schema dell’impatto dei repressori BinA e SypE sui substrati del biofilm è disponibile come Figura 5-figure supplement 3.DOI:
http://dx.doi.org/10.7554/eLife.24414.013(A-B) Aggregazione di ancestrali (A) ed evoluti (B) MJ11 sull’epitelio della mucosa ospite prima della colonizzazione. Tessuto ospite colorato con CellTracker Orange. I simbionti portano plasmidi GFP (pKV111) (Nyholmet al., 2000). Le micrografie mostrano aggregati rappresentativi di V. fischeri a seguito della dissezione di 30 animali appena nati incubati con ogni ceppo. Gli aggregati sono stati visualizzati tra 2 e 3 ore dopo l’inoculazione con un microscopio confocale a scansione laser Zeiss LSM 510 Meta. Barre di scala: 24 μm.DOI:
http://dx.doi.org/10.7554/eLife.24414.014Differenze trascriptomiche tra wild-type MJ11(binK+), MJ11 binK1 adattato ai calamari, e MJ11 ∆binK per i loci di codifica nel genoma MJ11 come determinato da RNA-Seq. Le varianti sono state campionate durante la crescita precoce dei log (OD600 ~ 0,25) in rich media (SWTO) prima della produzione di biofilm rilevabile da quattro repliche biologiche per ogni ceppo. Il verde indica un aumento dell’espressione; il rosso indica un’espressione ridotta rispetto all’espressione media per locus (cioè, conteggi letti in scala z rispetto alla media logCPM). La mappa termica mostra solo i loci per i quali l’espressione media in una variante binK differisce da quella in MJ11 con una soglia di significatività FDR di 0,05 (Tabella 3). Le etichette colorate si riferiscono a composti il cui metabolismo, trasporto o sintesi sono influenzati dall’espressione di questi geni. I geni coinvolti nella sintesi della cellulosa sono indicati con le frecce (VFMJ11_A1000- proteina C dell’operone della sintasi della cellulosa, e VFMJ11_A1007- proteina YhjU dell’operone della sintasi della cellulosa) e il log fold change (logFC) relativo al wild-type è indicato per binK1 e poi ∆binK.DOI:
http://dx.doi.org/10.7554/eLife.24414.015SypE reprime la produzione di biofilm Syp dopo la trascrizione(Morris e Visick, 2013). BinA reprime la cellulosa, ma non il Syp, formazione di biofilm aumentando l’attività della fosfodiesterasi (Bassise Visick, 2010). Le linee nere indicano una regolazione negativa. Le frecce grigie indicano trascrizione/traslazione. Si noti che, poiché il binA è espresso da un promotore del locus syp, l’attivazione del locus syp che porta alla produzione di Syp porta anche alla repressione della cellulosa.DOI:
http://dx.doi.org/10.7554/eLife.24414.016
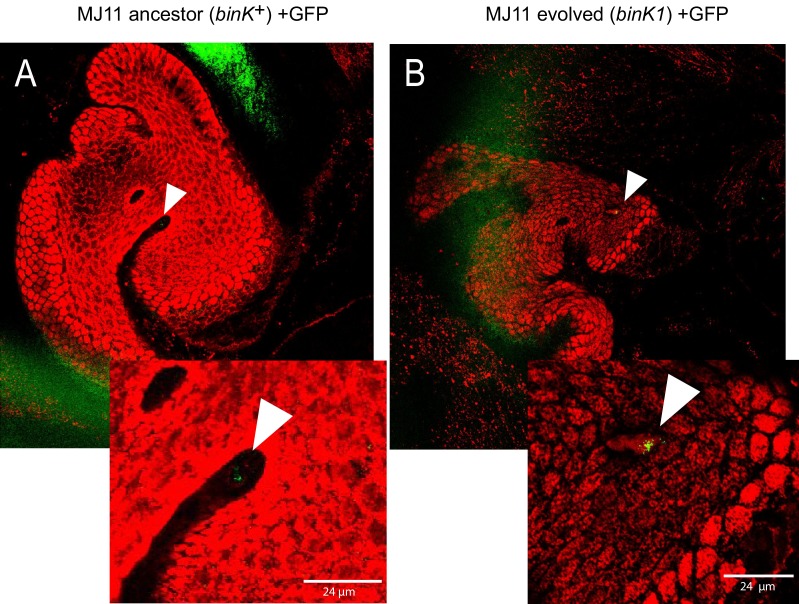
Figura 5-figure supplement 1.Cambiamenti comportamentali in vivo di aggregazione conferita dalla variante evoluta binK1.(A-B) Aggregazione di ancestrale (A) e si è evoluto (B) MJ11 su epitelio mucosale ospite prima della colonizzazione. Tessuto ospite colorato con CellTracker Orange. I simbionti portano plasmidi GFP (pKV111) (Nyholmet al., 2000). Le micrografie mostrano aggregati rappresentativi di V. fischeri a seguito della dissezione di 30 animali appena nati incubati con ogni ceppo. Gli aggregati sono stati visualizzati tra 2 e 3 ore dopo l’inoculazione con un microscopio confocale a scansione laser Zeiss LSM 510 Meta. Barre di scala: 24 μm.DOI:
http://dx.doi.org/10.7554/eLife.24414.014
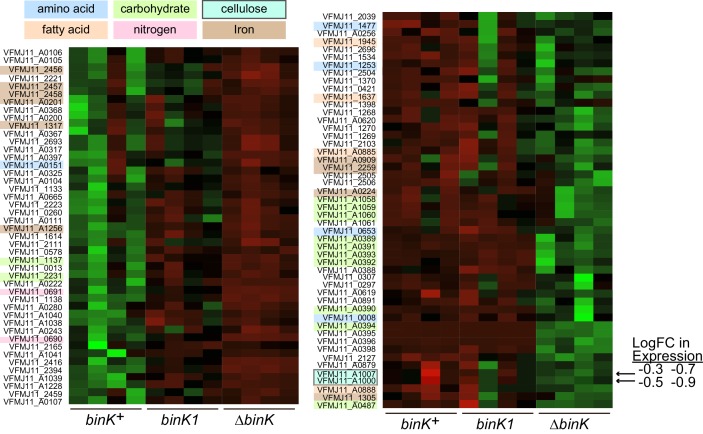
Figura 5-figure supplement 2.Spostamenti di trascrizione associati alle varianti binK.Differenze trascriptomiche tra wild-type MJ11(binK+), MJ11 binK1 adattato ai calamari, e MJ11 ∆binK per i loci di codifica nel genoma MJ11 come determinato da RNA-Seq. Le varianti sono state campionate durante la crescita precoce dei log (OD600 ~ 0,25) in rich media (SWTO) prima della produzione di biofilm rilevabile da quattro repliche biologiche per ogni ceppo. Il verde indica un aumento dell’espressione; il rosso indica un’espressione ridotta rispetto all’espressione media per locus (cioè, conteggi letti in scala z rispetto alla media logCPM). La mappa termica mostra solo i loci per i quali l’espressione media in una variante binK differisce da quella in MJ11 con una soglia di significatività FDR di 0,05 (Tabella 3). Le etichette colorate si riferiscono a composti il cui metabolismo, trasporto o sintesi sono influenzati dall’espressione di questi geni. I geni coinvolti nella sintesi della cellulosa sono indicati con le frecce (VFMJ11_A1000- proteina C dell’operone della sintasi della cellulosa, e VFMJ11_A1007- proteina YhjU dell’operone della sintasi della cellulosa) e il log fold change (logFC) relativo al wild-type è indicato per binK1 e poi ∆binK.DOI:
http://dx.doi.org/10.7554/eLife.24414.015

Figura 5-figure supplement 3.Schema di regolazione da parte dei repressori biofilm SypE e BinA.SypE reprime la produzione di biofilm Syp dopo la trascrizione(Morris e Visick, 2013). BinA reprime la cellulosa, ma non il Syp, formazione di biofilm aumentando l’attività della fosfodiesterasi (Bassise Visick, 2010). Le linee nere indicano una regolazione negativa. Le frecce grigie indicano trascrizione/traslazione. Si noti che, poiché il binA è espresso da un promotore del locus syp, l’attivazione del locus syp che porta alla produzione di Syp porta anche alla repressione della cellulosa.DOI:
http://dx.doi.org/10.7554/eLife.24414.016
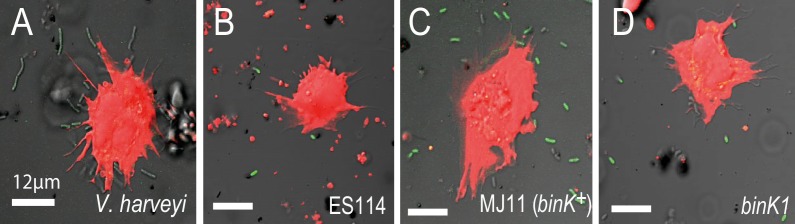
Figura 6-figure supplement 1.La produzione di biofilm da parte delle varianti binK1 che si adattano ai calamari media l’evasione degli emociti.la risposta in vitro degli emociti di calamari al Vibrio selvaggio, evoluto dai calamari e mutante.A) Efficienza relativa del legame degli emociti di calamaro di ceppi V. fischeri marcati GFP, tra cui: simbionte nativo del calamaro ES114, binK+ MJ11, ∆binKMJ11 (RF1A4), binK1 MJ11, e l’agente patogeno dei molluschi V. harveyi B392.(B) Efficienza relativa del legame degli emociti di calamari del simbionte nativo dei calamari ES114 e del bink1 MJ11 adattato ai calamari che trasporta il vettore vuoto (pVSV104), sypE (pRF2A1) o binA (pRF2A4). N = 30-52 emociti quantificati per ceppo. Barre di errore: 95% CI. I valori p significativi (p<0,05) sono indicati sopra ogni confronto. Si prega di fare riferimento alla Figura 6-figure supplement 1 per le micrografie delle interazioni tra Vibrio-emocita.DOI:
http://dx.doi.org/10.7554/eLife.24414.017Le micrografie mostrano esempi di cellule non simbiotiche legate all’emocito (A:Vibrio harveyi), calamaro-simbiotico (B: V. fischeri ES114 ), calamaro-naive (C: V. fischeri MJ11 binK+ ) e calamaro-adattato (D: MJ11 binK1 ). Il numero medio di cellule Vibrio marcate GFP legate da emociti è stato quantificato rispetto alla carica batterica totale in un raggio di 60 µm utilizzando la microscopia confocale con ingrandimento 63X, dopo un’ora di esposizione batterica. Emociti di calamari in rosso (CellTracker Orange), Vibrio in verde (GFP). Barre di scala: 12 μm.DOI:
http://dx.doi.org/10.7554/eLife.24414.018

Figura 6-figure supplement 1.Risposta in vitro di emociti di calamari a Vibrio selvaggio, evoluto da calamari e mutante.Le micrografie mostrano esempi di cellule non simbiotiche legate ad emociti (A:Vibrio harveyi), calamaro-simbiotico (B: V. fischeri ES114 ), calamaro-naive (C: V. fischeri MJ11 binK+ ) e calamaro-adattato (D: MJ11 binK1 ). Il numero medio di cellule Vibrio marcate GFP legate da emociti è stato quantificato rispetto alla carica batterica totale in un raggio di 60 µm utilizzando la microscopia confocale con ingrandimento 63X, dopo un’ora di esposizione batterica. Emociti di calamari in rosso (CellTracker Orange), Vibrio in verde (GFP). Barre di scala: 12 μm.DOI:
http://dx.doi.org/10.7554/eLife.24414.018
Sia il Syp che la cellulosa hanno contribuito a migliorare l’efficienza della colonizzazione dei calamari con le varianti binK
Data l’importanza dimostrata del Syp per l’inizio della colonizzazione(Shibata et al., 2012), abbiamo previsto che l’aumento della produzione di Syp da parte dei derivati binK ha migliorato la colonizzazione(Figura 3A). Sebbene sia il Syp che la cellulosa abbiano conferito diversi fenotipi importanti per la simbiosi (Figure 5B,C e 6), un ruolo per la cellulosa durante i processi di colonizzazione non è ancora stato dimostrato. Qui, la repressione del Syp (attraverso l’espressione del sypE) o della cellulosa (attraverso l’espressione del binA) ha ridotto significativamente l’efficienza della colonizzazione da parte di MJ11 e dei suoi derivati binK(Figura 7A). Tuttavia, la colonizzazione di sypE compromessa da ∆binK in misura maggiore rispetto al binA. Questo ci ha suggerito che il syp può giocare un ruolo maggiore della cellulosa nella colonizzazione, in accordo con i risultati dell’evasione degli emociti (Figura 6B). In alternativa, il sypE potrebbe produrre altri effetti normativi(Shibata et al., 2012; Bassis e Visick, 2010; Ray et al., 2015; Miyashiro et al., 2014). Per affrontare in modo più diretto il contributo di Syp al miglioramento della colonizzazione, abbiamo valutato l’impatto di un’eliminazione di sypK, che elimina la colonizzazione da parte del simbionte nativo (Shibataet al., 2012). La perdita di sypK non ha avuto alcun effetto percepibile sulla colonizzazione di MJ11, presumibilmente perché Syp è già sotto-prodotto(Mandel et al., 2009), ma come previsto, ha ridotto significativamente la colonizzazione di entrambe le varianti binK1 e ∆binK (Figura 7B). In particolare, la cancellazione della colonizzazione di sypK solo modestamente compromessa (riduzione del 25%) dalla variante binK1, suggerendo che Syp non è l’unico contributore della sua colonizzazione potenziata. L’eliminazione di sypK ha avuto un impatto maggiore sulla colonizzazione da parte del mutante ∆binK rispetto alla variante binK1, riducendo la sua colonizzazione a livelli wild-type, che potrebbe riflettere il maggior costo di fitness associato all’allele ∆binK (Figura 3Ae B) o potrebbe alludere a funzioni uniche associate all’allele binK1 evoluto. Insieme, questi risultati suggeriscono che sia il Syp che la cellulosa contribuiscono ad una maggiore efficienza di colonizzazione nelle varianti binK1 e ∆binK.10.7554/eLife.24414.019Cifra 7.Contributo del Syp e della cellulosa ad una migliore colonizzazione dei calamari con le varianti binK.(A) Efficienza di colonizzazione (% di calamari colonizzati a 24 ore) con il tipo selvaggio MJ11 (binK+), le varianti binK1 e ∆binK per i calamari in presenza del vettore vuoto (EV, pVSV105) (riempimento bianco), il Syp repressor sypE ( pCLD48 ) (riempimento a tratteggio), o il binA del repressore della cellulosa ( pRF2A3 ) (riempimento grigio). n = 15-20 repliche biologiche.(B) Influenza di una delezione sypK sull’efficienza di colonizzazione delle varianti MJ11 e binK. n = 31-52 repliche biologiche. Barre di errore: 95% CI. I valori p significativi (p<0,05) sono indicati sopra ogni confronto. *p<2.2e-16.DOI:http://dx.doi.org/10.7554/eLife.24414.019
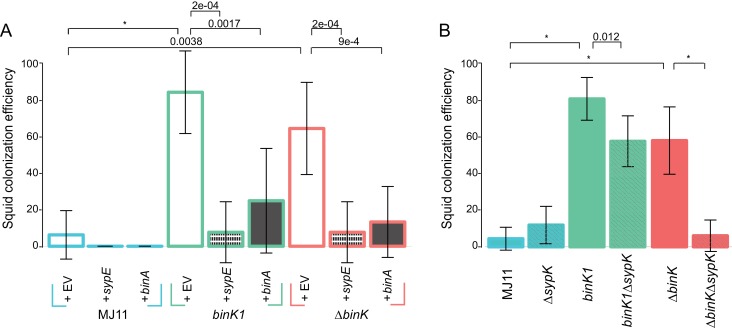
Figura 7.Contributo del Syp e della cellulosa al miglioramento della colonizzazione dei calamari da parte delle varianti binK.(A) Efficienza di colonizzazione (% di calamari colonizzati a 24 ore) da wild-type MJ11(binK+), le varianti binK1 e ∆binK per i calamari in presenza del vettore vuoto (EV, pVSV105) (riempimento bianco), il Syp repressor sypE ( pCLD48 ) (riempimento a tratteggio), o il binA del repressore della cellulosa ( pRF2A3 ) (riempimento grigio). n = 15-20 repliche biologiche.(B) Influenza di una delezione sypK sull’efficienza di colonizzazione delle varianti MJ11 e binK. n = 31-52 repliche biologiche. Barre di errore: 95% CI. I valori p significativi (p<0,05) sono indicati sopra ogni confronto. *p<2.2e-16.DOI:
http://dx.doi.org/10.7554/eLife.24414.019
BinK adattato ai calamari ha ridotto la luminescenza attenuando il rilevamento del quorum
La bioluminescenza serve come moneta di questa simbiosi, eppure la correlazione di un’eccessiva bioluminescenza con una scarsa capacità simbiotica suggerisce che l’intensità della luminescenza è un fenotipo modellato dalla selezione dell’ospite (Leee Ruby, 1994a; Nishiguchi et al., 1998; Visick et al., 2000). I derivati adattati ai calamari di MJ11 – dove l’antenato wild-type è ≥1.000 volte più luminoso del ceppo simbionte nativo ES114 (Schusteret al., 2010) – hanno sviluppato un ritardo nell’induzione della luminescenza rispetto ai loro antenati. Per determinare se le soglie di quorum-sensing erano state alterate dalle mutazioni binK, abbiamo quantificato la produzione dei segnali AinS-sintetizzati C8-HSL e LuxI-sintetizzati C8-HSL e LuxI-sintetizzati 3-oxo-C6-HSL e la produzione simultanea di luminescenza da parte del wild-type MJ11 e binK1, e le varianti ∆binK durante il periodo di induzione (OD600 1.1) (Figura 8). Per tutti e tre i ceppi, luminescenza correlata con 3-oxo-C6-HSL concentrazione(Figura 8A)(r2= 0,857, p=6,4×10-13) e non concentrazione C8-HSL (r2= 0.105, p=0.1). Rispetto al tipo wild-type, sia la variante binK1 che la variante ΔbinK degli alleli binK hanno ridotto la produzione di 3-oxo-C6-HSL e la corrispondente luminescenza di un ordine di grandezza (Figura 8). Queste differenze significative non sono state causate da MJ11 di raggiungere una maggiore densità cellulare (2,0 × 108 CFU-ml-1-OD600-1), comesia il binK1 e ΔbinK derivati prodotto CFU leggermente superiore (Figura 8B) (3.2 × 108 CFU-ml-1-OD600-1 e 3,7× 108 CFU-ml-1-OD600-1, rispettivamente)(Figura 8B). Anche se c’è stato un modesto (<2 volte) aumento della concentrazione molare di C8-HSL in supernatanti mutanti ΔbinK, che potrebbe inibire la produzione di luce attraverso l’inibizione competitiva di LuxR-binding al suo cognato 3-oxo-C6-HSL segnale (Kuo et al.,1996; Schaefer et al., 1996), non vi era alcuna differenza rilevabile nella produzione di C8-HSL quando si controlla per i conteggi delle cellule più elevate prodotte dal mutante ΔbinK rispetto al wild-type MJ11 (p = 0,82) (Figura 8B). Questi risultati sono in accordo con i precedenti saggi biologici e dimostrano che la mutazione binK1 altera il rilevamento del quorum e aumenta la soglia per l’attivazione del quorum-sensing della luminescenza (Schusteret al., 2010).10.7554/eLife.24414.020Figure 8.Host-adapted binK1 attenua la regolazione del quorum-sensing di luminescenza.(A) Supernatant concentrazioni (nM/OD600) di N-(3-oxohexanoyl) omoserina lattone (C6-HSL), come quantificato contro standard sintetici(Schaefer et al., 2000; Pearson et al., 1994; Duerkop et al., 2007) e la luminescenza corrispondente (Lum/OD600) di 10 colture indipendenti ciascuna per i derivati wild-type MJ11, binK1 e ΔbinK durante il quorum-sensing induzione della luminescenza determinata da colture cresciute a log precoce (media OD600 1,1, range 0,9-1,4,).(B) Densità cellulare media come misurato da assorbanza (OD600), unità di formazione di colonia (CFU)/mL/OD600, N-(3-oxohexanoyl) lattone omoserina (C6) concentrazione nM, N-octanoyl lattone omoserina (C8) concentrazione nM, e luminescenza (Lum)/1 mL cultura per dieci repliche biologiche di ogni variante rispetto al wild-type MJ11. Barre di errore: 95% CI. Significativi p-valori p (p<0,05) sono indicati sopra ogni confronto. *p<2.2e-16.DOI:http://dx.doi.org/10.7554/eLife.24414.020
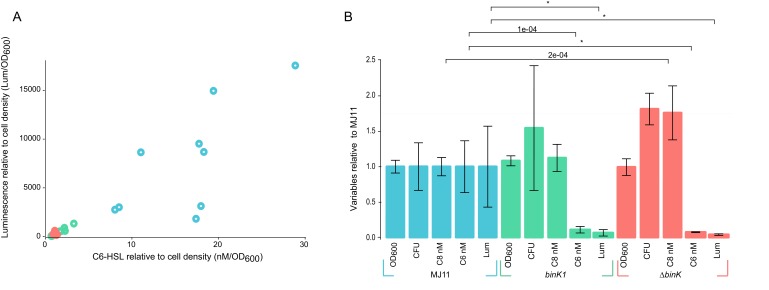
Figura 8.Il binK1 adattato all’host attenua la regolazione del quorum di rilevazione della luminescenza.(A) Le concentrazioni dei surnatanti (nM/OD600) di N-(3-oxohexanoyl) omoserina lattone (C6-HSL), quantificate rispetto a standard sintetici(Schaefer et al., 2000; Pearson et al., 1994; Duerkop et al., 2007) e la corrispondente luminescenza (Lum/OD600) di 10 colture indipendenti ciascuna per i derivati wild-type MJ11, binK1 e ΔbinK durante il quorum-sensing induzione della luminescenza determinata da colture cresciute a log precoce (media OD600 1.1, range 0.9-1.4, ).(B) Densità cellulare media come misurato da assorbanza (OD600), unità di formazione di colonia (CFU)/mL/OD600, N-(3-oxohexanoyl) lattone omoserina (C6) concentrazione nM, N-octanoyl lattone omoserina (C8) concentrazione nM, e luminescenza (Lum)/1 mL cultura per dieci repliche biologiche di ogni variante rispetto al wild-type MJ11. Barre di errore: 95% CI. Significativi p-valori p (p<0,05) sono indicati sopra ogni confronto. *p<2.2e-16.DOI:
http://dx.doi.org/10.7554/eLife.24414.020
L’adattamento dell’ospite ha prodotto alleli binK dominanti che suggeriscono una funzione alterata
I confronti tra la variante binK1 evoluta dai calamari e la variante ΔbinK mutante, esemplificata in particolare dall’efficienza di colonizzazione (Figura 7B), dai profili trascrizionali e dai cambiamenti nell’attività metabolica che erano convergenti con il simbionte nativo (Figura 5-figure supplement 2, Appendice 1, Appendice 2), suggeriscono che la selezione dei calamari non ha favorito la perdita della funzione BinK in MJ11. Le varianti evolute binK1 e null ∆binK non differivano in modo significativo nella produzione di biofilm ed esibivano simili tratti di sopravvivenza all’ossidazione e all’evasione degli emociti legati al biofilm (Figura 5C e 6). Tuttavia, la variante binK1 adattata ai calamari ha superato significativamente il mutante nullo nella competizione culturale con il binK+(Figura 3B). Questo miglioramento della forma fisica potrebbe essere dovuto al mantenimento di una funzione parziale o ad effetti regolatori che sono unici per l’allele evoluto. Per indagare ulteriormente su questo aspetto, abbiamo valutato l’impatto dell’espressione multi-copia di alleli wild-type e binK1. Il binK+ancestrale ha integrato i comportamenti adattativi conferiti dai mutanti binK1 e ∆binK, comprese le capacità di formare biofilm e di colonizzare i calamari, come ci si aspetterebbe se la funzione binK wild-type BinK compromettesse questi tratti (Figura 9). L’espressione multi-copia di binK1 ha ridotto modestamente la produzione di biofilm da parte del mutante ∆binK, suggerendo che la funzione parziale è stata mantenuta da questo allele, ma ha anche migliorato inaspettatamente la produzione di biofilm da parte di MJ11, implicando una funzione alterata (Figura 5Be 11). Infine, il binK1 ha significativamente migliorato la colonizzazione di tutte le varianti, anche in presenza di una singola copia genomica dell’allele wild-type, dimostrando che il binK1 è dominante e coerente con la sua funzione alterata. Anche se la ridotta attività di binK è stata sufficiente a conferire alcuni tratti adattativi(Figure 5-8), questi risultati suggeriscono che una migliore simbiosi potrebbe anche sorgere attraverso fenotipi conferiti dall’alterazione della sua funzione (Figure9-11).10.7554/eLife.24414.021Figure 9.Effetto del binK sulla colonizzazione dei calamari e sulla produzione di biofilm.(A) Miglioramento della colonizzazione tramite multi-copia nell’espressione trans dell’allele evoluto binK1 e diminuzione della colonizzazione tramite l’espressione dell’allele ancestrale binK+ allele. Colonizzazione valutata in percentuale di calamari luminosi dopo 24 ore. Barre di errore: 95% CI. N = 15–25.(B) Aumento della produzione di biofilm risultante dall’espressione trans dell’allele binK1 e diminuzione della produzione di biofilm risultante dall’espressione dell’allele binK+ ancestrale. I confronti tra la produzione di biofilm nei plasmidi di controllo (pVSV105= EV) e quella dei plasmidi multi-copia che portano il binK suggeriscono un ruolo inibitorio per il BinK nella produzione di biofilm, presumibilmente alleviato dalla dominanza dell’allele binK1 . La produzione di biofilm è stata quantificata dall’assorbanza del violetto cristallino a A550. Il colore di sfondo rappresenta lo sfondo del ceppo in cui sono stati misurati gli effetti del plasmide multicopia, rispecchiando quelli usati in tutti i casi in cui il blu è il tipo selvaggio MJ11, il verde è la variante evoluta del binK1 ed il salmone è il derivato ∆binK. Barre di errore: 95% CI; la non sovrapposizione indica un significato. N = 7–8. I valori p significativi (p<0,05) sono indicati sopra ogni confronto. *p<0.05, **p<0.005, ***p<0.005.DOI:http://dx.doi.org/10.7554/eLife.24414.02110.7554/eLife.24414.022Figure10.Modello di regolazione BinK dei tratti adattativi durante la simbiosi dei calamari.Le frecce che provengono da BinK puntano a caratteristiche che sono attivate o potenziate, e le linee bloccate puntano a quelle che sono represse o bloccate da BinK. Le linee tratteggiate indicano i polisaccaridi che contribuiscono al biofilm.DOI:http://dx.doi.org/10.7554/eLife.24414.022
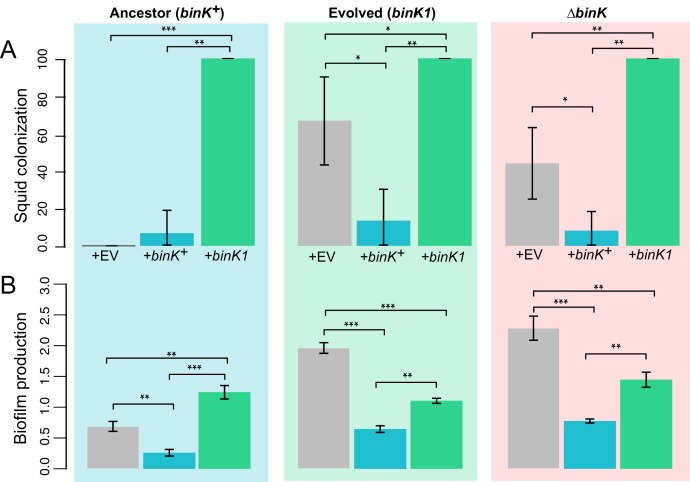
Figura 9.Effetto di binK sulla colonizzazione dei calamari e sulla produzione di biofilm.(A) Miglioramento della colonizzazione tramite multi-copia nell’ espressione trans dell’allele evoluto binK1 e diminuzione della colonizzazione tramite l’espressione dell’allele ancestrale binK+ allele. Colonizzazione valutata in percentuale di calamari luminosi dopo 24 ore. Barre di errore: 95% CI. N = 15–25.(B) Aumento della produzione di biofilm risultante dall’espressione trans dell’allele binK1 e diminuzione della produzione di biofilm risultante dall’espressione dell’allele binK+ ancestrale. I confronti tra la produzione di biofilm nei plasmidi di controllo (pVSV105= EV) e quella dei plasmidi multi-copia che portano il binK suggeriscono un ruolo inibitorio per il BinK nella produzione di biofilm, presumibilmente alleviato dalla dominanza dell’allele binK1 . La produzione di biofilm è stata quantificata dall’assorbanza del violetto cristallino a A550. Il colore di sfondo rappresenta lo sfondo del ceppo in cui sono stati misurati gli effetti del plasmide multicopia, rispecchiando quelli usati in tutti i casi in cui il blu è il tipo selvaggio MJ11, il verde è la variante evoluta del binK1 ed il salmone è il derivato ∆binK. Barre di errore: 95% CI; la non sovrapposizione indica un significato. N = 7–8. I valori p significativi (p<0,05) sono indicati sopra ogni confronto. *p<0,05, **p<0,005, ***p<0,005.DOI:
http://dx.doi.org/10.7554/eLife.24414.021
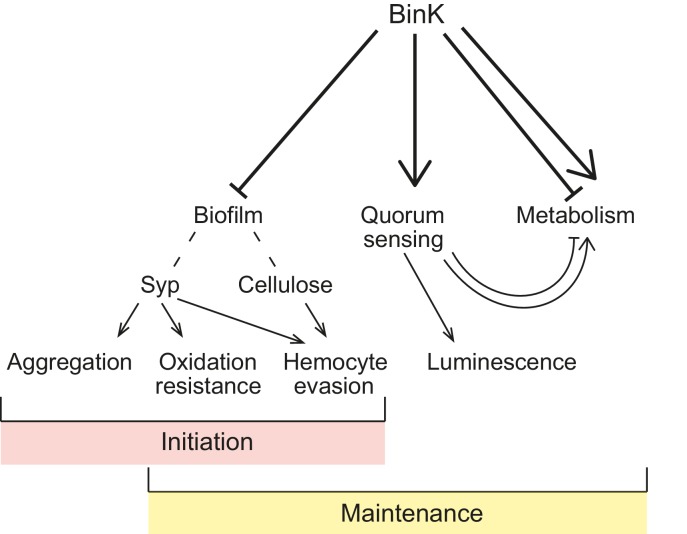
Figura 10.Modello di regolazione BinK dei tratti adattativi durante la simbiosi dei calamari.Le frecce che hanno origine da BinK puntano a caratteristiche che vengono attivate o potenziate, e le linee bloccate puntano a quelle che vengono represse o bloccate da BinK. Le linee tratteggiate indicano i polisaccaridi che contribuiscono al biofilm.DOI:
http://dx.doi.org/10.7554/eLife.24414.022
Discussione
In teoria, le grandi dimensioni della popolazione e la diversità genetica all’interno delle specie batteriche possono consentire uno stile di vita simbiotico con gli ospiti eucarioti di evolvere rapidamente (Fisher, 1930). Mentre i processi che portano all’emergenza dei patogeni sono stati intensamente studiati, molto meno si sa sui cambiamenti genetici che guidano l’adattamento a nuove nicchie di ospiti in batteri non patogeni(Jansen et al., 2015; Ochman e Moran, 2001; Kwong e Moran, 2015; Guan et al., 2013). Negli agenti patogeni, gli elementi mobili codificati su isole di patogenicità sono spesso citati come causa della ripetuta e rapida evoluzione delle associazioni di ospiti, ma questi elementi da soli raramente forniscono ai batteri la capacità di colonizzare gli ospiti (Reuteret al., 2014). Inoltre, le pressioni selettive esercitate dai nuovi ospiti possono richiedere cambiamenti fenotipici sincronizzati, limitando il numero di “soluzioni” adattative disponibili per un genoma microbico che è vincolato dalla struttura normativa. Qui, rapido adattamento alla simbiosi calamaro si è verificato in molteplici lignaggi sperimentali paralleli attraverso mutazioni convergenti in un singolo gene, il binK sensor kinase. Queste mutazioni hanno alterato funzioni multiple che sono note per contribuire alla simbiosi nativa tra il ceppo ES114 e il calamaro (Figura 10), suggerendo che i circuiti regolatori di V. fischeri possono essere stati precablati per coordinare diversi tratti simbiotici. Molti dei comportamenti regolati da BinK hanno stabilito ruoli cruciali nell’associazione simbiotica, tra cui l’attivazione del quorum di bioluminescenza e l’aggregazione mediata da Syp, (Nishiguchiet al., 1998; Brooks e Mandel, 2016; Nyholm e McFall-Ngai, 2003; Shibata et al., 2012; Visick et al., 2000; Yip et al., 2005), ma noi forniamo la prima prova sperimentale che due diverse sostanze a matrice cellulare a regolazione binK, Syp e cellulosa, modulano le interazioni immunitarie innate dell’ospite che potrebbero contribuire alla discriminazione del ceppo durante la selezione dei partner simbiotici.
I percorsi convergenti di adattamento intrapresi da lignaggi indipendenti che si evolvono sperimentalmente attraverso il calamaro rivelano che gli ospiti del calamaro esercitano una dura selezione sui batteri colonizzatori, guidando l’evoluzione dei genotipi simbiotici, che sono alla base dell’evoluzione dei genotipi simbiotici. Un modello della popolazione – la dinamica genetica della colonizzazione batterica suggerisce che per sopravvivere all’estinzione durante le strozzature imposte dall’ospite, gli alleli binK devono conferire un enorme vantaggio selettivo nell’associazione simbiotica e devono emergere precocemente durante la crescita della popolazione, molto probabilmente – prima del reclutamento dell’ospite – più tardi durante il mantenimento della simbiosi (Figura 4A eC). Questa previsione è coerente con la migliorata capacità di iniziazione delle varianti evolute(Figure 1, 3A, 7 e 9) e spiega il loro rilevamento nei primi passaggi dei calamari(Tabella 2). Non ci si aspetterebbe che questi mutanti aumentino a frequenza rilevabile considerando che gli alleli che conferiscono una maggiore idoneità nei calamari sono deleteri nella coltura del brodo (Figura 3B). Il successo delle mutazioni binK, passando da una frequenza non rilevabile nell’inoculo ancestrale alla fissazione in meno di ~ 50 generazioni, è stato realizzato solo in presenza di una forte selezione dell’ospite del calamaro. Coefficienti selettivi stimati per l’allele binK1 di MJ11 variava fino a s = 5,3 quando determinato empiricamente, simile alle stime ottenute dalla modellazione della popolazione (s ~ 6) (vedi Materiali e metodi, Figura 4). Coefficienti selettivi superiori a uno sono raramente riportati dalla natura; tuttavia, questi sono coerenti con le rigorose pressioni di selezione imposte agli agenti patogeni mentre colonizzano nuovi ospiti(Morley et al., 2015; Bedhomme et al., 2012; Thurman e Barrett, 2016). Questo enorme vantaggio selettivo è anche coerente con l’osservazione che le popolazioni ancestrali con una forma fisica media inferiore (come i ceppi MJ11 e H905) hanno maggiori probabilità di fare un grande salto di adattamento rispetto alle popolazioni più in forma (come WH1, EM17 ed ES114) (Lenskie Travisano, 1994). Cioè, a causa della loro distanza dalla forma fisica ottimale (ad esempio, colonizzazione al 100%), gli antenati meno in forma sono pronti a beneficiare maggiormente delle mutazioni di maggiore vantaggio selettivo(Orr, 2000, 2003; Wielgoss et al., 2013). Quindi, anche se l’eliminazione della funzione BinK aumenta anche l’idoneità competitiva di ES114(Brooks e Mandel, 2016), non sorprende che le mutazioni binK non si siano evolute in popolazioni derivate da antenati con una maggiore idoneità iniziale, come EM17, WH1, ed ES114, poiché è improbabile che queste mutazioni possano conferire un vantaggio selettivo sufficiente per sopravvivere all’estinzione (Figura 2A e 4C, Tabella 2). Gli elevati vantaggi selettivi previsti deimutanti binK si sono evoluti da ceppi MJ11 e H905 mal adattati ai calamari, supportano la teoria che l’adattamento da antenati non idonei può inizialmente procedere con grandi balzi, in contrapposizione a cambiamenti incrementali di piccolo effetto (Wiseret al., 2013).
Requisito per una simbiosi di successo con i calamari è la capacità dei batteri di bypassare le barriere dell’ospite durante l’iniziazione: i simbionti prima si aggregano e poi migrano attraverso i condotti che sono sorvegliati da emociti e alla fine raggiungono l’interno dell’organo luminoso ossidativo (Figura 1)(Nyholm e McFall-Ngai, 2004).La scarsa capacità di colonizzazione di MJ11 è stata attribuita alla sua mancanza di rscS, un regolatore acquisito orizzontalmente nella stessa famiglia ibrida istidina chinasi della famiglia binK ( Figura 2-figuresupplement 1). RscS che attiva il polisaccaride Syp e permette al simbionte nativo ES114 di superare la barriera di iniziazione dei calamari(Figura 1A)(Yip et al., 2006; Mandel et al., 2009). Nonostante la sua funzione conservata come repressore del Syp in ES114(Brooks e Mandel, 2016), BinK non ostacola la simbiosi in quel ceppo, forse a causa dell’attività di RscS (Yipet al., 2006). Ma, in particolare, il ceppo H905, un ceppo vicino a ES114 isolato dall’habitat dei calamari e contenente rscS, è simbioticamente compromesso e ha anche evoluto mutazioni convergenti in binK durante i nostri esperimenti (Perry,2009) (Figura 2-figuresupplement 1, Tabella 2). Ciò suggerisce che la sua carenza di colonizzazione deriva da vincoli normativi sulla produzione di Syp, dall’integrazione inefficace del regolatore RscS acquisito orizzontalmente con i circuiti normativi esistenti, o dall’evoluzione degli attributi relativi ad uno stile di vita planctonico che compromettono la sua capacità di accedere agli organi leggeri dei calamari (Lee e Ruby,1994a). Qui, MJ11 adattato alla simbiosi sperimentale calamaro attraverso il potenziamento non solo di Syp(Figure 5-7),un meccanismo proposto per l’evoluzione simbiotica nel simbionte nativo ES114 (Mandelet al., 2009), ma anche producendo ulteriori componenti della matrice come la cellulosa (Figura 5-figuresupplemento 2, appendice 1), entrambi i quali hanno migliorato la colonizzazione (Figura 7).
La scoperta che i fenotipi del biofilm conferite dagli alleli binK evoluti hanno migliorato la sopravvivenza delle difese dell’ospite(Figura 5 e 6) amplia la nostra comprensione della natura della selezione dell’ospite e fornisce un contesto importante per il modo in cui il biofilm può conferire maggiore fitness agli individui. I microbi simbiotici comunemente secernono esopolisaccaridi o composti glicosilati per produrre capsule di biofilm che conferiscono protezione contro macrofagi, antibiotici o sostanze tossiche, e che promuovono l’adesione alle superfici epiteliali (Nizete Esko, 2009; Sengupta et al., 2013; Williams et al., 2013; Hsieh et al., 2003). Tuttavia, al di là del suo ruolo nella formazione di aggregati, non si sapeva se il biofilm avesse contribuito alla colonizzazione dei calamari(Yip et al., 2006). L’allele binK1 ha permesso l’evasione immunitaria riducendo l’attaccamento di emociti simili ai macrofagi dell’ospite ad un livello paragonabile a quello del ceppo ES114 nativo dei calamari e migliorando la sopravvivenza quando esposto all’ossidazione (Figura 5). Sia l’evasione immunitaria che la produzione di biofilm sono state soppresse dalla sovraespressione dei repressori sypE o binA, il che indica che questi tratti sono mediati dalla produzione di Syp e cellulosa (Figura 5). La risposta immunitaria dei calamari è mitigata da V. fischeri lipopolisaccaridi e altri pattern molecolari microbici associati (MAMP)(Nyholm et al., 2009; Nyholm e McFall-Ngai, 1998; Koropatkin et al., 2012; Koropatnick et al., 2004; Foster et al., 2000), ma questo studio fornisce la prima prova che il Syp contribuisce all’immunomodulazione ospite da parte di V. fischeri. I geni per Syp condividono poca somiglianza con quelli che codificano il polisaccaride capsulare comune alle specie di Vibrio immunomodulanti e ad altri patogeni (Shibataet al., 2012; Yildiz e Visick, 2009), ma il polisaccaride per il Syp può comunque svolgere un ruolo analogo a quello dei ligandi polisaccaridi dei recettori dei macrofagi dei mammiferi prodotti dai simbionti intestinali, che mostrano anche un’attività immunosoppressiva che riduce la risposta infiammatoria dell’ospite (Mazmanianet al., 2008; Chu e Mazmanian, 2013; Jones et al., 2014). Recenti evidenze nel Vibrio parahaemolyticus suggeriscono che l’uso del Syp è potenzialmente diffuso tra i Vibrio associati all’ospite , mediando la virulenza e la colonizzazione epiteliale (Ye et al.,2014) così come l’evasione dell’immunità innata dell’ospite (Hsieh et al.,2003; Vuong et al., 2004). Gli effetti pleiotropici del Syp sulla competenza simbiotica suggeriscono perché le singole mutazioni binK forniscono un tale beneficio al calamaro-naïve V. fischeri. Inoltre, rivelano un ruolo critico per i polisaccaridi associati alle cellule nell’interazione tra calamaro e bibrio, non solo mediando i comportamenti di gruppo che migliorano l’iniziazione, ma anche contribuendo alla selezione del partner su base cellulare individuale.
Non solo gli alleli binK evoluti aumentano la forma fisica durante le prime 24 ore di colonizzazione, ma migliorano ulteriormente la forma fisica tra le 24 e le 48 ore dopo la colonizzazione durante la fase di mantenimento della simbiosi(Figura 4B, Figura 3-figure supplement 2) quando il calamaro seleziona l’intensità della luminescenza simbionte e l’utilizzo delle risorse (Grafe Ruby, 1998; Soto et al., 2014; Schuster et al., 2010; Visick et al., 2000; Septer et al., 2013; Soto e Nishiguchi, 2014)(Figura 1A). Sebbene la luminescenza potrebbe essere direttamente sotto la selezione dell’ospite(Figura 8A) (Visicket al., 2000; Whistler e Ruby, 2003), la selezione potrebbe in alternativa favorire la soglia di quorum alterata alla base della luminescenza ridotta nelle varianti binK (Figura 8B). Le mutazioni in luxO e litR – che, come le varianti binK, attenuano il quorum di rilevamento – aumentano la capacità competitiva sia nella cultura che nei calamari (Fidopiastiset al., 2002; Kimbrough e Stabb, 2015). L’alterato rilevamento del quorum da parte di altre specie migliora anche la crescita competitiva a causa della successiva de-repressione delle funzioni metaboliche come l’assorbimento e l’utilizzo dei carboidrati e la perturbazione della biosintesi degli acidi grassi e dei carboidrati (Davenportet al., 2015; An et al., 2014). L’analisi trascrittomica ha indicato che cambiamenti simili si sono verificati nel Δ binK mutante( Figura 5-figuresupplement 2; Appendice 1). Le vie metaboliche regolate dal quorum che servono come “beni privati” potrebbero essere bersaglio di selezione se facilitano l’utilizzo di risorse fornite dall’ospite che supportano la crescita simbionte nei calamari giovani (Appendici 1 e 2) (Grafe Ruby, 1998; Pan et al., 2015; Wier et al., 2010; Miyashiro et al., 2011; Mandel et al., 2012; Dandekar et al., 2012), contribuendo così a un vantaggio selettivo duraturo dopo la colonizzazione iniziale(Figure 1A e 4B).
I cambiamenti sincronizzati ottenuti attraverso le sostituzioni di aminoacidi in una chinasi di sensore esistente evidenziano come i componenti conservati ma malleabili dei sistemi di trasduzione del segnale li rendono mediatori chiave dell’evoluzione adattiva(Figura 2C). Durante l’evoluzione batterica, i percorsi di trasduzione sensoriale possono servire come bersagli flessibili a causa della modularità dei loro componenti (Vogelet al., 2004; Pasek et al., 2006). I domini di phosphorelay e di accessori conservati (ad esempio la Figura 2C) sono condivisi attraverso numerosi percorsi e facilitano le interazioni flessibili con i partner, note come ‘cross-talk’(Capra e Laub, 2012). Le istidina chinasi dei sensori sono obiettivi efficaci di regimi selettivi in parte grazie alle loro capacità di doppia chinasi e fosfatasi, così come la loro capacità di aumentare le interazioni tra partner attraverso questi moduli condivisi che possono consentire un rapido ricablaggio delle reti (Capra e Laub, 2012; Tayloret al., 2015; Rowland e Deeds, 2014). L’array di fenotipi effettuati nelle varianti binK(Figure 5, 6 e 8) implica che la chinasi del sensore BinK può partecipare a più di un percorso di trasduzione del segnale(Nyholm e McFall-Ngai, 2004; Yip et al., 2006; Miyashiro e Ruby, 2012). I cambiamenti fenotipici potrebbero essere causati da un’interazione alterata con una serie di regolatori con moduli di fosforelay già descritti sia per il polisaccaride Syp (Brooks eMandel, 2016) che per il percorso di rilevamento del quorum che controlla la luminescenza (Miyashiroe Ruby, 2012; Whistler et al., 2007), anche se questo non elimina la possibilità che ci siano partner non identificati che mediano questi effetti. Indipendentemente da ciò, BinK evoluto ha messo in atto effetti globali intersecando i circuiti preesistenti, che presumibilmente sono stati modellati da interazioni variabili con gli ambienti, compresi gli host, durante l’evoluzione adattiva di V. fischeri(Gao e Stock, 2013; Mitrophanov e Groisman, 2008).
Questo studio dimostra che alcuni ceppi di V. fischeri possono evolvere con salti nella gamma di ospiti che risultano da singole mutazioni di grande effetto. Che semplici mutazioni puntiformi in un regolatore possono evocare conseguenze così ampie rivela che i tratti disparati che sono importanti per l’inizio e il mantenimento della simbiosi sono già co-regolati. Tale coordinamento preesistente è quasi certamente un’abilità evoluta, che riflette forse una storia di selezione e di “armeggiamento”, mentre fluttua tra gli ambienti non ospitali e quelli associati all’ospite in cui questi batteri risiedono naturalmente (Lee e Gelembiuk,2008; Jacob, 1977). Le immense popolazioni di specie di Vibrio dovrebbero, in teoria, permettere alla selezione naturale di affinare anche i tratti più sottili, promuovendo la capacità di adattarsi a condizioni incerte attraverso un’adeguata regolazione con notevole efficacia(Dillon et al., 2017). Visto in questa luce, questo studio suggerisce che l’eccezionale adattabilità di alcuni batteri come il Vibrio nel formare nuove associazioni intime con vari organismi ospiti può essere possibile in parte grazie alla struttura dei percorsi di regolazione esistenti che si sono formati durante migliaia di passate interazioni transitorie. Una tale parsimoniosa riconciliazione dei vincoli genomici con le pressioni di selezione dell’ospite è probabilmente fondamentale per la formazione di simbiosi emergenti.
Materiali e metodi
Ceppi batterici, plasmidi e condizioni di coltura
I ceppi e i plasmidi sono elencati nella Tabella 1. Vibrio fischeri wild-type Vibrio fischeri compreso il ceppo MJ11 (isolato dal pesce Monocentris japonica[Haygood et al., 1984]) e i suoi derivati, così come il simbionte calamaro ES114, sono stati abitualmente coltivati a 28°C sia in brodo liquido di acqua di mare-tryptone (SWT) che in brodo Luria Bertani con aggiunta di sale (LBS) con agitazione a 200 rpm, o su terreno LBS con 1,5% di agar (LBS agar) (Graf etal., 1994). I ceppi di Escherichia coli sono stati abitualmente coltivati in brodo Luria-Bertani (LB) (Sambrook et al., 1989) o in mezzo di infusione del cuore del cervello (BHI) (Difco) a 37°C. Quando necessario, i mezzi sono stati integrati con antibiotici alle seguenti concentrazioni: per V. fischeri, cloramfenicolo (Ch) a 2,5 μg/ml, kanamicina (Km) 100 μg/ml ed eritromicina (Em) a 5 μg/ml; per E. coli, Ch a 25μg/ml, Km a 50 μg/ml, ed Em a 150 μg/ml (per mezzi BHI). Per mantenere la selezione in acqua di mare, questi antibiotici sono stati utilizzati a metà di questa concentrazione. Quando applicabile, le piastre di agar sono state integrate con 40 mg di 5-bromo-4-cloro-3-indolil-β-galattopiranosidasi (X-gal)/ml per la visualizzazione dell’attività della β-galattosidasi. Per la quantificazione del biofilm, i batteri sono stati coltivati in brodo liquido di acqua di mare-tryptone con aggiunta di sale (SWTO) (Boseet al., 2007). Per generare biblioteche trascrittomica, i batteri sono stati coltivati in 3 mL SWTO integrato con 0,5 mM N-acetil-D-glucosamina. I batteri sono stati anche cresciuti in variazioni di HEPES minimo terreno (HMM) (Rubye Nealson, 1977), un terreno minimo definito a base di acqua di mare con 1x acqua di mare artificiale (ASW: 50 mM MgSO4, 10 mM CaCl2,300 mM NaCl, 10 mM KCl), 0.333 mM K2HPO4, 18,5 mM NH4Cl, e 0,0144% di casaminoacidi, tamponati con 10 mM Hepes con un’adeguata fonte di carbonio. Altri tamponi sono stati sostituiti e nutrienti supplementari sono stati integrati come segue: per la concorrenza in vitro, il mezzo è stato integrato con 0,53 mM di glucosio; per la valutazione sideroforo in condizioni di ferro ridotto(Payne, 1994a), il mezzo è stato tamponato con 100 mM Pipes (pH 6,8), i casaminoacidi sono stati aumentati a 0.3%, e il mezzo è stato integrato con 32,6 mM glicerolo; e per la rilevazione qualitativa di siderofori, questo mezzo è stato inoltre integrato con 1,5% Difco bacto-agar e 10% di cromo azurol S-hexadecyltrimethylammonium bromide soluzione di dosaggio (CAS -HDTMA) (Leee Ruby, 1994a; Payne , 1994a; Boettcher e Ruby, 1990; Graf e Ruby, 2000). I plasmidi sono stati coniugati tra E. coli e V. fischeri come descritto in precedenza(Stabb e Ruby, 2002).
Tecniche di ricombinazione del DNA e PCR
Integrated DNA Technologies (Coralville, IA) ha sintetizzato i primer oligonucleotidici elencati nella Tabella 4. La PCR di routine è stata eseguita utilizzando AccuStart II PCR Supermix (Quanta, Houston, TX). Phusion High Fidelity DNA polimerasi (New England Biolabs, Ipswich, MA) è stato utilizzato per la clonazione e per produrre modelli per il sequenziamento delle reazioni. La PCR ciclismo è stata eseguita secondo il protocollo del produttore in un Eppendorf Mastercycler o Master Cycler Nexus (Eppendorf, Amburgo, Germania). Le temperature di ricottura utilizzate per i primer sono state determinate sottraendo 2°C dalle temperature di fusione (Tm) determinate dal Netprimer di Premiere Biosoft. La più bassa temperatura di ricottura dei primer nella reazione è stata utilizzata durante la PCR(Tabella 4).10.7554/eLife.24414.023Tabella 3.Genomi utilizzati nelle analisi filogenetiche. Questa tabella elenca le adesioni GenBank per i genomi nucleotidici utilizzati nella filogenesi dei ceppi e la fonte per i modelli genici utilizzati nella filogenesi ibrida istidina chinasi.DOI:http://dx.doi.org/10.7554/eLife.24414.023StrainNCBIaccessione/de novoProkka/NCBI modelEscherichiacoliNC_000913NCBIAliivibriowodanisLN554846-51NCBIA. salmonicidaNC_011311-6NCBIA.logeiNZ_AJYJ000000000000ProkkaVibriofurnissiiNC_016602, NC_016628NCBIVibrioparahaemolyticusNC_004603-5NCBIVibriofischeri SR5NZ_AHIH00000000000000ProkkaVibriofischeriES114NC_006840-2NCBIVibriofischeriMJ11NC_011184-6NCBIVibrio fischeriEM17DenovoProkkaVibrio fischeriWH1DenovoProkkaVibrio fischeriZF211AJYI01ProkkaVibriofischeriWH4DenovoProkkaVibriofischeriSA1DenovoProkkaVibriofischeri CG101DenovoProkkaVibrio fischeriH905DenovoProkkaVibrio fischeriPP3DenovoProkkaVibrio fischeriVLS2DenovoProkka10.7554/eLife.24414.024Table 4.DNA oligonucleotide primer utilizzati in questo studio.DOI:http://dx.doi.org/10.7554/eLife.24414.024Nome del primerSequenzaDNA del primer (5′-3”)Temperatura di ricotturaFonte A0397 F5AAGAGAGTCATGGTATACATCGGG51°C Questo studioA0397 F5*TGTAGCTGATGGATGAGAGACTTTGCG56°CQuesto studio A0397 F8TCATTGAAAGGGTTTAATCGGGTGTGTGT57°CQuesto studio A0397 R11CACTTTATGGGATGATGATGATCTTCGGCT56°.CQuesto studio;A0397 F3GCTGATGGATGAGAGACTTTCGCTGCTC52°CQuesto studio;A0397 R4GGGCTGATTAGATTAGATCATCCTGC54°CQuesto studioA0397 F12CAGAGAAGCACCACTAAATCATGGATGTGAG52°CQuesto studio A0397 R9TCTGACACATGCCAATAATGCCATGCCAT59°CQuesto studio MJ11A0397 R KpnIGGTACCCCGAAATTAACGACCAT50°.C Questo studio MJ11A0397 F SalIGTCGACAAATAGAAAACACACTAACCAC50°C Questo studio HKSoeA F (SalI)GTCGACAATGTAGAGAAGTGGTAGAGAACGC50°.CIl presente studio HKSoeA2 RGTTTCCGCCCCCCATTGATTGGTGTGTGTGTGTGTTTCTCT350°C Il presente studio HKSoeB2 FAGAAACACACTAACCACAAAAAGAATGGCGGAAAC50°C Il presente studioHKSoeB2 RGCACCGACCGACACTCATCAATCAATTCGATCAATCAAGCT50°CQuesto studio HKSoeC2 FAGCTTGATGATCGAATTGGATGAGTGGGTGGTGGTGC50°CQuesto studioHKSoeC R (KpnI)GGTACCAGCGGGCAATAGAATCAATCAGTC50°CQuesto studio TnErm4AATGCCCTCTACCTGTTACCTGTTCC53°CQuesto studio TnErm5CATGCGTCTGACATCTGA55°CQuesto studioA0397 R13GTACACCCCCGAAATTAACGACGACCA59°CQuesto studio A0397 F10CAGAGTTATGGGGGGGGTTGGGGAGTG58°CQuesto studio A0397 WT+ RGTCCCACCAAATTGACG53°.C Questo studio;A0397 4+ RGTCCCACCAAATTGACAATTGACA53°C Questo studio di tipo RF F2GCAGGGTTATGTGCGAGG52°C Questo studio di tipo F1GCCGTAGTGTGTACTTCGAGCG55°.C31 gapA R1CCCATTACTCTCACCCTTGTTGTTTGG55°C31 PrRF9AAGCTTATTGGGGAATACGGGATACGGATACCTG53°C Questo studio PrRF10CATATGCACATCTTCTAACCATTGGG53°.CIl presente studio PrRF19TGTGTCAGTATCACTCCCCTTCTCAC55°C Il presente studio PrRF20AGCAGACAGAGAGTTTTTTATTGTTCATTGTATTGTTTCACCTACCTCATTTAA50°; CIl presente studio PrRF19TGTCAGTATCACTCCCCTTCTCATTTAA50CIl presente studio PrRF21TTAAATGGAGGGTGAAACAACAATGAACAATAAACTGTCTGGCTGCTCT50°C Il presente studio PrRF22TTTCCTGTGTTTGTTTTTTTTTTTTTTTAGAAAAACTCATCGAGCA50°; CIl presente studio PrRF21TTAAATGAGGTGAAACAACAACAACAATGAACAATAAACTGTCTCTGCT50CIl presente studio PrRF23TGGCTCGATGGAGTTTTTTTCTAAAAAAAAAAAAACAACAGGAAA50°CIl presente studio PrRF24GTTCCCCTTACAAGTCCTATTCCTATTCC53°CIl presente studioPrRF36ATCCATTGATTGTAATAGTGGCTGGC53°CQuesto studio PrRF52AATAAGTCCATTATTTCGTTGTTGGC54°CQuesto studio PrRF53AAGCGGAAGTAGGCGAAAAC54°CQuesto studioVSV105InFGCCTGGGGGGGGTGCCCCTAATG56°CQuesto studio KanINFATACAAGGGGGGTGTTATGAGCC55°CQuesto studio KanINRCAAGTCAGCGTAATGCTCTGC56°CQuesto studio
I metodi molecolari standard e i protocolli dei produttori sono stati utilizzati per le trasformazioni, i digest enzimatici di restrizione, le legature, l’elettroforesi in gel e la PCR. Gli enzimi di restrizione sono stati acquistati da New England Biolabs (Beverly, MA), e T4 DNA Ligase era da Invitrogen (Waltham, MA). L’isolamento del gel e l’estrazione del DNA dai digest di restrizione sono stati fatti utilizzando il Qiagen QIAquick Gel Extraction Kit (Qiagen, Valencia, CA). Plasmidi per il lavoro ricombinante e per il sequenziamento sono stati purificati utilizzando Zymo Research Zyppy Plasmid Mini Prep (Irvine, CA). Il DNA genomico utilizzato nelle reazioni PCR è stato isolato con il metodo di estrazione fenolo/cloroformio (Wilson,2001). Le amplificazioni PCR di routine sono state eseguite con AccuStart II (Quanta Bio, Beverly, MA).
Sequenziamento del genoma e analisi
Il DNA genomico è stato estratto da colture a metà del log coltivate in LBS utilizzando il kit di purificazione del DNA genomico di Promega Wizard (Madison, WI). I genomi di V. fischeri ceppi EM17, WH1 e H905 sono stati sequenziati de novo utilizzando il sequenziamento a singola molecola (Pacific Biosciences) e assemblati utilizzando HGAP presso la Icahn School of Medicine. I modelli genici per i genomi de novo sono stati previsti e annotati utilizzando Prokka con il ceppo ES114 che funge da riferimento (Seemann, 2014). Per tutti i ceppi derivati dall’evoluzione sperimentale (sia esperimenti su calamari che su colture), le biblioteche genomiche sono state preparate su cloni isogenici seguendo un protocollo di costruzione di biblioteche Nextera modificato ad alto rendimento (Baymet al., 2015) e sono state sequenziate utilizzando la piattaforma Illumina Hi-Seq 2500 presso la University of New Hampshire o il New York Genome Center. Le sequenze dell’adattatore PE Nextera sono state rimosse dalle letture grezze utilizzando Trimmomatic(Bolger et al., 2014) con le impostazioni della clip come segue: ILLUMINACLIP = 2:40:15 LEADING:2 TRAILING:2 TRAILING:2 MINLEN:25 (Macmanes,2014). Le letture elaborate sono state allineate e analizzate rispetto al rispettivo genoma di riferimento del ceppo (ancestrale) per identificare le mutazioni, utilizzando le impostazioni predefinite in breseq(Deatherage e Barrick, 2014) per i genomi a singolo isolato e utilizzando l’impostazione ‘-polimorfismo’ per le biblioteche costruite con gDNA isolato in pool. In media, il 99% delle letture elaborate da ogni isolato mappato sul loro genoma di riferimento, con una copertura cromosomica media di 95x per isolato(Tabella 2) per MJ11. Le mutazioni sono state chiamate solo per le regioni coperte da un minimo di 20 letture. Per identificare quali richiami di mutazione riflettevano il vero cambiamento evolutivo rispetto agli errori nel genoma di riferimento PacBio o NCBI, abbiamo confrontato ogni richiamo putativo su tutti i genomi derivati dallo stesso antenato. I potenziali richiami di mutazione per il ceppo ES114 sono stati confrontati con varianti note(Foxall et al., 2015). Ogni richiamo di mutazione che è stato condiviso tra almeno il 50% dei genomi di ceppi evoluti indipendentemente è stato assunto per riflettere il genotipo ancestrale e quindi scartato. Tutte le mutazioni nel locus binK identificate da breseq sono state successivamente confermate da amplificazione PCR mirata e sequenziamento Sanger utilizzando i primer A0397 F3 e A0397 R4 per l’amplificazione e per la sequenza binK1 e binK2, e i primer A0397 F8 e A0397 R9 per l’amplificazione e A0397 F3 e A0397 R6 per la sequenza binK3 e binK4 (UNH e GeneWiz).
Relazioni filogenetiche tra V. fischeri
Sequenza nucleotidica dei genomi pubblicati delle Vibrionaceae(Vibrio parahaemolyticus, Aliivibrio salmonicida, A. logei, e V.fischeri ceppi ES114, MJ11, SR5, ZF-211; Tabella 3) e dei genomi di nuova generazione (V. i ceppi difischeri H905, EM17, SA1, CG101, VLS2, PP3, WH1, WH4) sono stati analizzati in REALPHY e RAxML per dedurre la filogenesi del genoma intero con la massima probabilità di filogenesi sotto il modello GTRGAMMA di sostituzione nucleotidica(Bertels et al., 2014). Il supporto dei nodi è stato stimato eseguendo 1.000 analisi bootstrapped.
Colonizzazione dei calamari ed evoluzione sperimentale di V. fischeri
La colonizzazione dei calamari è stata condotta come descritto in precedenza(Whistler e Ruby, 2003). I calamari sono stati allevati da adulti raccolti da Maunalua Bay, HI con gli adulti originali raccolti e allevati nel dicembre 2006, e le coorti successive raccolte ad intermittenza dalla stessa località tra il 2007 e il 2016. I calamari sono stati tenuti di routine in 32 ppt Instant Ocean (IO) (Blacksburg, VA) in acqua diH2O. Per determinare l’efficienza di colonizzazione, una coorte di calamari è stato posto in inoculo batterico derivato da mid-log (OD600 0,2) colture di brodo SWT diluito in IO filtrato. La luminescenza dei calamari alloggiati individualmente in 4 mL di IO è stata monitorata giornalmente, e la colonizzazione batterica è stata determinata mediante diluizioni di placcatura di calamari omogeneizzati dopo il congelamento a -80°C. Per le misure di capacità di partenza, calamari sono stati esposti a inoculo per 3 ore (ES114, EM17, e WH1) o durante la notte (H905 e MJ11) a concentrazioni crescenti di batteri (da 3.000 a 20.000 CFU / ml), fino al 90% dei calamari sono stati colonizzati come determinato dalla rilevazione di luminescenza a 24 e 48 ore dopo la colonizzazione, e placcatura diretta di omogeneizzati di organi leggeri a 48 ore dopo la colonizzazione. Esperimenti di colonizzazione sono stati completati con almeno 10 replicare calamaro, incluso calamaro controllo aposymbiotic, e sono stati ripetuti un minimo di tre volte.
Ceppi MJ11, EM17, WH1, H905, ed ES114 sono stati evoluti utilizzando calamari ospiti come descritto in precedenza(Schuster et al., 2010). In breve, 10 calamari aposimbiotici sono stati inoculati in una popolazione ancestrale di ogni ceppo (20.000 CFU/ml in 50 ml di IO filtrato per H905 e MJ11, 6.000 CFU/ml per WH1, e 3.000 CFU/ml EM17 ed ES114). Dopo l’incubazione notturna, i calamari sono stati isolati e sciacquati in IO filtrato. Calamaro con luminescenza rilevabile dopo 48 ore è servito come il passaggio fondatore per ogni popolazione parallela replicare replicare. A 96 ore dopo l’inoculazione iniziale, i calamari ospiti sono stati conservati a -80°C mentre la loro acqua di mare contenente ventato è stata utilizzata per inoculare un nuovo passaggio di calamaro apossimbiotico. La metà del ventato è stata conservata congelando il 40% di glicerolo a -80°C. Passaggio seriale con 1 ml di ventato combinato con 1 mL di IO fresco è stato avviato con un calamaro schiusa tenutasi durante la notte per confermare che sono stati noncolonizzati sulla base di misurazioni di luminescenza. Passaggio continuato in questo modo per un totale di 15 calamari ospiti per ogni lignaggio sperimentale (vedi Figura 1C).
Isolati da vari passaggi delle evoluzioni sono stati recuperati e conservati dal ventato archiviato. Dieci microlitri del ventato sono stati placcati su agar SWT e incubati a 28 ° C, e le colonie rappresentative che erano fenotipicamente simili a V. fischeri erano quadranti striati per l’isolamento su agar LBS. Le colonie isolate sono state coltivate in mezzi liquidi LBS e conservate mediante congelamento in glicerolo al 40% a -80°C per analisi successive. Per gli isolati la cui identità come V. fischeri era sospetta a causa di differenze morfologiche, è stata misurata la luminescenza da colture SWT, e il gene diagnostico del ceppo gapA è stato amplificato e sequenziato utilizzando primer gapA F1 e gapA R1(Tabella 4) per conferma(Nishiguchi et al., 1998).
Ortologia BinK e filogenesi ibrida istidina chinasi
Per costruire un albero genetico per i geni ibridi di istidina chinasi attraverso i ceppi V. fischeri e i parenti Vibrio, ciascuno dei modelli di gene dei genomi completi elencati nella tabella 4 sono stati interrogati con i modelli PFAM Hidden Markov per i domini HATPase C (PF02518), HisKA (PF00512) e REC (PF00072) utilizzando hmmer. Sequenze contenenti tutti questi domini conservati sono stati poi allineati in MAFFT(Katoh et al., 2002). Una topologia di massima probabilità è stata dedotta usando RAxML(Stamatakis, 2006) sotto il modello PROTGAMMAWAG di sostituzione degli aminoacidi, seguendo la selezione del modello usando il Bayesian Information Criterion con IQ-TREE(Nguyen et al., 2015). Le famiglie di geni sono state annotate sulla base del consenso tra i ceppi ES114, Vibrio parahaemolyticus, e le annotazioni di E. col i identificate utilizzando l’algoritmo BLAST(Camacho et al., 2009).
Identificazione degli alleli
Gli isolati dal secondo sfiato dei calamari provenienti dalla popolazione di replicati MJ11 sono stati sottoposti a screening per gli alleli binK e binK1 utilizzando il primer forward A0397 F5* e i primer inversi allele-specifici A0397 WT+ R e A0397 4+ R per binK e binK1, rispettivamente(Tabella 4). La presenza o l’assenza di ampliconi è stata valutata rispetto ai controlli che includono MJ11 (binK+), la variante binK1 MJ11EP2-4-1 e la variante ∆binK RF1A4. L’amplificazione PCR è stata condotta a seguito di denaturazione a 95°C per 30 s seguita da ricottura a 53°C per 15 s e allungamento a 72°C per 50 s. Per confermare l’identità degli alleli, la regione binK in cinque isolati è stata amplificata mediante PCR usando A0397 F10 e A0397 R13, e i dNTP e i primer non consumati sono stati rimossi usando ExoSAP-IT (Affymetrix Santa Clara, CA) prima del Sanger-sequencing a Genewiz (Cambridge, MA) usando i primer A0397 F3 e A0397 R4 (Tabella 4). I risultati sono stati allineati con il riferimento MJ11_A0397 utilizzando i programmi Lasergene Software (DNASTAR, Inc. Madison, WI) e la presenza di binK1 negli isolati evoluti è stata confermata.
∆binK generazione di mutanti
Il ceppo MJ11 ∆binK::EmR(RF1A4 ) è stato generato dalla mutagenesi a scambio di marker usando un costrutto prodotto dalla PCR di Splicing e Overlap Extension PCR (Hortonet al., 1990). In breve, le coppie di primer HKSoeA F (SalI) e HKSoeA2 R, HKSoeB2 F e HKSoeB2 R, e HKSoeC2 F e HKSoeC R (KpnI), e la Phusion High Fidelity DNA polimerasi sono stati utilizzati per amplificare la regione genomica a monte e a valle del binK da DNA genomico MJ11, utilizzandoEmRcolonie e DNA plasmidico pEVS170 come modelli (Tabelle 1e 4) ( Lyell et al., 2008). Gli ampliconi purificati sono stati poi fusi usando la polimerasi Expand Long Template (Roche) dove il binK è stato sostituito da una cassetta EmR. Questo prodotto purificato è stato clonato in pCR2.1 TOPO e trasformato in cellule TOP10 (Invitrogen, Waltham, MA), seguendo il protocollo del produttore. Cloni putativi sono stati sequenziati con il metodo Sanger con primer M13 F, M13 R, TnErm4, e TnErm5(Tabella 4) presso l’Hubbard Center for Genome Studies della University of New Hampshire prima che il frammento è stato sub clonato nel vettore suicida pEVS79, che è stato utilizzato per lo scambio allelico(Stabb e Ruby, 2002). Il ri-sequenziamento dell’intero genoma (illumina HiSeq) ha confermato che il gene è stato sostituito nel mutante MJ11 RF1A4.
Generazione di mutanti ∆sypK
I ceppi mutanti ∆sypK::aphA1 RF1A5, RF1A6, e RF1A7 sono stati generati dalla mutagenesi a scambio di marker utilizzando un costrutto prodotto da Splicing e Overlap Extension PCR (Hortonet al., 1990). In breve, le coppie di primer PrRF19 e PrRF20, PrRF21 e PrRF22, e PrRF23 e PrRF24, e la Phusion High Fidelity DNA polimerasi sono state usate per amplificare la regione genomica a monte e a valle della sypK dal DNA genomico MJ11, e utilizzando come modello le colonie KmR e il DNA plasmidico pVSV103 (Tabelle 1 e 4) (Dunnet al., 2006). Gli ampliconi purificati sono stati poi fusi usando la polimerasi Expand Long Template (Roche) dove il sypK è stato sostituito da una cassetta KmR. Questo prodotto purificato è stato clonato in pCR2.1 TOPO e trasformato in cellule TOP10 (Invitrogen, Waltham, MA), seguendo il protocollo del produttore. I cloni putativi sono stati sequenziati con il metodo Sanger con primer M13 F, M13 R, KanINF, KanINR(Tabella 4) a Genewiz a South Plainfield, NJ prima che il costrutto, RF2B7, fosse utilizzato per lo scambio allelico con un protocollo di competenza chitinica modificato(Brooks et al., 2015). In breve, le cellule di V. fischeri sono state coltivate in mezzi minimi con un derivato della chitina (n-acetil glucosamina) fino a raggiungere OD600 0,2. Le colture sono state incubate con 10 µg/mL di pRF2B7 linearizzato da un massimo di cinque cicli di congelamento-scongelamento. Dopo l’incubazione con frammenti di DNA per lo scambio allelico, le cellule sono state recuperate, placcate su piastre LBS+Km e schermate con PCR per l’incorporazione di ∆sypK::aphA1 frammento utilizzando primer PrRF36 e KanINR2 (Tabella 4).
Sequenziamento e analisi del trascrittoma
Singole colonie di V. fischeri MJ11 e due dei suoi ceppi derivati, il ceppo binK1 evoluto dai calamari (MJ11EP2-4-1) e il mutante MJ11 ΔbinK (RF1A4 ), sono stati coltivati in quadruplicato fino ad avere un OD600 di 0.25 (Biofotometro; Eppendorf AG, Amburgo, Germania) al fine di catturare le popolazioni prima dell’attività biofilm rilevabile o flocculazione e per ridurre al minimo gli effetti della soppressione spontanea a causa di difetti di crescita delle varianti binK . Le cellule sono state pellettate e congelate in flash. L’RNA è stato estratto seguendo il protocollo per il kit Quick-RNA MiniPrep (Zymo, Irvine, CA). Ribosomal RNA è stato impoverito utilizzando il kit RiboZero (Illumina). librerie mRNA sono state costruite utilizzando il TruSeq Stranded mRNA kit di preparazione biblioteca mRNA (Illumina) e sequenziato utilizzando il 2500 HiSeq al New York Genome Center. Le letture di qualità sono state mappate sul genoma di riferimento MJ11 usando bowtie2 (Langmeade Salzberg, 2012) e quantificate usando RSEM (Li e Dewey, 2011). L’espressione differenziale tra i ceppi è stata valutata utilizzando edgeR (Robinsonet al., 2010) con una soglia di significatività di FDR < 0,05.
Costruzione in plasmide
Gli allelibinK e binK1 sono stati clonati in pVSV105(Dunn et al., 2006) a seguito dell’amplificazione del DNA genomico MJ11 e binK1 con primer forward MJ11A0397 F SalI e reverse MJ11A0397 R KpnI(Tabella 4). Il prodotto di 2,977 Kb è stato clonato in pCR2.1 TOPO (Invitrogen) seguendo le istruzioni del produttore. I costrutti sono stati sequenziati utilizzando M13F, M13R, A0397 F3, A0397 F5, A0397 F8, A0397 F12, A0397 R4, A0397 R9 e A0397 R11(Tabella 4) e allineati ai rispettivi riferimenti per garantire l’assenza di mutazioni. Gli inserti sono stati sub clonati da pCR2.1 TOPO in pVSV105 a seguito di digestione con gli enzimi di restrizione SalI e KpnI, e legatura con T4 DNA ligasi. Le reazioni di legatura sono state trasformate in cellule pir DH5αλ chimicamente competenti(Herrero etal., 1990). I lisati cellulari delle colonie ChR sono stati direttamente sottoposti a screening per la corretta inserzione di plasmidi che ospitano i plasmidi mediante PCR utilizzando M13F e A0397 R4. Cloni positivi ospitano pRAD2E1(binK+) e pRF2A2(binK1).
IlbinA è stato clonato in pVSV105(Dunn et al., 2006) a seguito dell’amplificazione del DNA genomico MJ11 con primer forward PrRF9 e reverse PrRF10(Tabella 4). Il prodotto da 2,053 Kb è stato clonato in pCR2.1 TOPO (Invitrogen) seguendo le istruzioni del produttore. I costrutti TOPO sono stati sequenziati utilizzando M13F, M13R, PrRF9, PrRF10, PrRF52 e PrRF53 (Tabella4), e allineati alla sequenza genomica in MJ11 utilizzando il pacchetto software DNA Star(https://www.dnastar.com/) per garantire che non siano state generate mutazioni durante la clonazione. Gli inserti sono stati sub clonati a seguito di digestioni con XhoI e NdeI e SalI e NdeI digestioni di pVSV105, e la legatura utilizzando T4 DNA ligasi. Le reazioni di legatura sono state trasformate in cellule pir DH5αλ chimicamente competenti. I lisati cellulari di ChR sono stati sottoposti direttamente a PCR per l’inserimento di plasmidi portatori di plasmidi da PrRF9 e VSV105InF(Tabella 4). Cloni positivi ospitati pRF2A3(binA+)(Tabella 1).
Per rendere i costrutti KmR compatibili con pKV111 per i saggi degli emociti, il frammento sypE SphI e SacI è stato subclonato da pCLD48 in SphI e SacI digerito pVSV104(Stabb and Ruby, 2002). A seguito della trasformazione in cellule pir piriche DH5αλ chimicamente competenti,i lisati cellulari delle colonie KmR sono stati sottoposti a screening diretto per l’inserimento di sypE utilizzando M13F e sypE RF F2 (Tabella 4). Cloni positivi ospitati pRF2A1(Tabella 1). Il frammento binA Sph1 e SacI è stato subclonato da TOPO 2.1 in pVSV104 digerito con SphI e SacI(Stabb and Ruby, 2002). I lisati cellulari delle colonie di KmR sono stati sottoposti a screening diretto per l’inserto binA utilizzando VSV105InF e PrRF9(Tabella 4). I cloni positivi ospitano pRF2A4(Tabella 1).
Per marcare i batteri per la competizione diretta, è stato usato il plasmide di espressione lacZ pVSV103(Dunn et al., 2006), che conferisce una colonia blu su supporti contenenti X-gal e conferisce resistenza alla kanamicina, insieme ad un derivato di questo plasmide (pCAW7B1) in cui la lacZ è stata inattivata dalla rimozione di un frammento interno di 624 bp tramite digestione con HpaI seguita da auto-legittimazione.
Concorrenza batterica in vivo
Le stime dei tassi di crescita e dell’idoneità di Malthusian per i ceppi MJ11 sono state calcolate misurando le abbondanze relative di ceppi marcati nei tratteggi dei calamari che sono stati co-inoculati con rapporti variabili di ciascun ceppo (Metodo del Rapporto di Partenza Alterato sensu[Wiser e Lenski, 2015]). I ceppi sono stati marcati con una versione intatta del plasmide pVSV103 (Dunnet al., 2006) o pCAW7B1 che contiene lacZ, che contiene una delezione di 200 aminoacidi che rende il LacZ incapace di produrre il pigmento blu nelle colonie (Tabella 1). Calamaro sono stati inoculati durante la notte in 50 ml di IO contenente 25 μg / ml Km e conservati a -80 ° C dopo 24 o 48 ore (n = 98 e 59, rispettivamente) dopo l’esposizione iniziale inoculo se rilevabilmente luminoso. Le inoculazioni hanno riguardato 17 esperimenti, che contenevano inoculi con ceppi reciprocamente marcati al fine di controllare i potenziali effetti plasmidici, che vanno sia nella densità totale delle cellule (da 1.600 a 26.600 CFU/mL) che nella frequenza di deformazione relativa (da ~ 1 binK1 per 10.000 binK+ fino a proporzioni approssimativamente uguali). Per stimare l’abbondanza di CFU per ogni ceppo negli organi leggeri dei calamari, abbiamo contato le colonie blu e bianche dopo 72 ore di placcatura degli omogeneizzati di calamari su piastre SWT contenenti 50 μg/ml Km e 1,5 mg/ml X-gal.
Per calcolare il coefficiente selettivo(s) associato alla variante evoluta durante la competizione con il genotipo ancestrale nel calamaro, usiamo la derivazione in Chevin (2011). In primo luogo, i tassi di crescita maltusiani (M)(Fisher, 1930) sono stati stimati prendendo il log naturale del rapporto della stima del CFU da ogni organo luminoso co-colonizzato alla concentrazione dell’inoculo di partenza (cioè la densità di partenza)(Lenski e Travisano, 1994; Lenski et al., 1991). Il metodo standard di placcatura per quantificare i simbionti degli organi leggeri dei calamari può rilevare solo 15 CFU(Ruby e Asato, 1993). Poi la differenza relativa del tasso di crescita(sGR) è stato utilizzato per calcolare il coefficiente di selezione:
Differenza relativa del tasso di crescita, sGR = (MEvo– – MAnc)/ MAnc
Coefficiente di selezione, s = sGR / ln2
I test di correlazione del rango di Spearman sono stati poi utilizzati per verificare le relazioni tra i tassi di crescita di Malthusian e la frequenza di partenza o la densità di partenza degli inoculi. Differenze significative nel tasso di crescita a 24 o 48 ore tra i ceppi ancestrali e quelli evoluti di binK1 sono state valutate utilizzando i test di permutazione esatti di Fisher-Pitman attraverso il metodo ‘oneway_test’ nel pacchetto R ‘coin’ (Hothorn et al.,2008). Differenze significative nella colonizzazione competitiva da varianti evolute binK1 e binK3 (mutazioni nei domini HATPaseC o HAMP, rispettivamente) sono state valutate con un t-test di permutazione nel pacchetto R ‘DAAG’ utilizzando il metodo ‘onet.permutation’ con 9.999 simulazioni (Maindonald eBraun, 2015).
Concorso batterico in vitro
I tassi di crescita di Malthusian sono stati stimati in modo simile alle competizioni in vivo in cui l’idoneità per i ceppi MJ11 è stata determinata a seguito di co-inoculazione di 150 μl con una singola colonia da ogni ceppo contrassegnato con pVSV103 (Dunn et al.,2006) o pCAW7B1. Le colture sono state coltivate staticamente a 28°C e, ad intervalli di 2 ore, è stata fondata una nuova coltura per diluizione seriale 1/10 in mezzi freschi in una micropiastra di polistirolo a 96 pozzetti (Corning). Ad ogni passaggio, 20 μl di ogni competizione è stato diluito, e placcato su piastre SWT contenenti 50 μg/ml Km e 1,5 mg/ml X-gal. Il numero totale di colonie blu e bianche apparente dopo 72 ore di crescita è stato determinato e utilizzato per il calcolo dei parametri maltusiani realizzati. Le gare di ceppo sono state condotte ciascuna con otto repliche e ripetute due volte. Le differenze nel tasso di crescita (parametro maltusiano, descritto sopra e in Fisher (1930) sono state valutate per la significatività utilizzando i test di permutazione esatti di Fisher-Pitman attraverso il metodo ‘oneway_test’ del pacchetto R ‘coin’ (Hothorn et al.,2008).
Stima teorica del vantaggio selettivo e della probabilità di mutazione in BinK
Modellazione del coefficiente di selezione
L’approssimazione analitica sviluppata in Wahl e Gerrish (2001) è stata utilizzata per stimare la gamma di coefficienti di selezione necessari per una nuova variante benefica per superare il rischio di estinzione in una popolazione esposta a frequenti strozzature:V(t,s)≅1-(ln22t-1sτ)
Dove, V(t,s) rappresenta la probabilità di estinzione dato il coefficiente (s) selettivo e la generazione (t) di crescita in cui la variante si presenta. Questo rischio è determinato dal numero di generazioni tra i colli di bottiglia (τ), il vantaggio (s) selettivo e la generazione di arrivo (t). Nel contesto della dinamica di colonizzazione del calamaro-Vibrio, i seguenti valori sono stati applicati a questi parametri: per il collo di bottiglia iniziale di colonizzazione dell’ospite a seguito della crescita dell’inoculo, τ è stato di 25 generazioni; per i successivi colli di bottiglia di sfogo sperimentati dalle popolazioni di simbionti, τ è stato di quattro generazioni.
Per stimare il tasso di selezione minimo(r) conferito da una nuova variante rara in grado di colonizzare con successo un ospite (cioè, comprendente una delle ~ 10 cellule iniziatrici[Wollenberg e Ruby, 2009; Altura et al., 2013]), per prima cosa abbiamo previsto il numero di mutazioni non sinonime che si sarebbero accumulate nel locus binK durante la crescita della popolazione ancestrale in evoluzione neutra utilizzando il tasso di mutazione stimato per V. fischeri (Dillon et al.,2017): questo è stato ~325 assumendo ~25 generazioni di divisione cellulare per formare una dimensione finale della popolazione di 2.4 × 108. Poi, usando il metodo di Lenski e Travisano (1994) per stimare le differenze di fitness nelle popolazioni in declino, i tassi di selezione sono stati stimati per la variante rara usando i parametri maltusiani(Fisher, 1930):M(variante rara)=ln(1/325)M(wild-type)=ln(9/2.4×108)r=M(variante rara)-M(wild-type)=5.6 tronchi naturali
Utilizzando queste approssimazioni, i coefficienti di selezione per le varianti che si presentano durante la fase di crescita dell’inoculo devono essere molto più grandi di uno per ottenere una ragionevole possibilità di sopravvivere al collo di bottiglia della colonizzazione. Al contrario, durante i periodi di ventilazione-ricrescita, anche se la probabilità che si verifichi una nuova mutazione è bassa, dato che relativamente poche generazioni si verificano durante la ricrescita giornaliera, alleli benefici con coefficienti così bassi a 0,5 possono regolarmente sopravvivere(Figura 3C).
Un avvertimento a questo approccio è che il modello applicato non ha incorporato dinamiche di sub-popolazione che potrebbero derivare da sfumature nella topologia dell’organo leggero di un singolo calamaro, piuttosto applica parametri generalizzati di popolazione e di crescita di una singola popolazione in evoluzione attraverso un calamaro sperimentale, utilizzando dati derivati dal ceppo nativo ES114 nella simbiosi calamaro-Vibrio (Wollenberge Ruby, 2009; Altura et al., 2013; Wahl e Gerrish, 2001). Mentre tale suddivisione della popolazione potrebbe potenzialmente facilitare la variazione genetica tra i simbionti, non influisce sul coefficiente selettivo stimato degli alleli evoluti.
Modellazione della probabilità di mutazione BinK
Per stimare la probabilità che si verifichi una mutazione neutra all’interno del locus binK durante la fase di crescita dell’inoculo o durante i cicli di crescita nell’ospite, sono stati utilizzati i seguenti parametri. I riferimenti sono forniti per tutti i parametri basati su stime pubblicate in precedenza.
ParameterEstimateStimateSourceGenome tasso di mutazione 2,08 × 10-8 bp-1division-1Dillon et al.(2017)Dimensione del genoma di MJ114,323,877 bpNCBIA disponibile posizioni binK non sinonimi (circa 2/3 delle posizioni codon)2,595 *2/3N0 (popolazione di partenza dell’inoculo)5 cellsNinoc (max. popolazione di inoculo prima della diluizione)2,4 × 108 cellsNinoc(V.fischeri founder population size)12 (2-3 cellule per cripta)Nyholm etal. (2000); Wollenberg e Ruby, (2009); Altura et al. (2013)Nhost (Juvenile light organ V. fischeri population capacity)5 × 105 cellsKoch et al. (2014)
Per collocare le osservazioni empiriche nel contesto delle aspettative utilizzando il modello di Wahl e Gerrish (2001), prevediamo che i mutanti portatori di un vantaggio selettivo di s ~ 2,8 avrebbero avuto origine all’interno delle prime 10 generazioni di crescita dell’inoculo, con la probabilità che qualsiasi mutazione non sinonima nel locus si verificasse all’interno delle prime 10 generazioni di crescita dell’inoculo sarebbe stata di 0,004 (sotto Poisson). Tuttavia, il recupero di quattro distinti alleli binK suggerisce che la selezione potrebbe essere molto più grande di questa stima empirica. Sebbene la quantificazione del vantaggio selettivo sia fondamentale per comprendere le dinamiche della selezione naturale durante l’evoluzione, ottenere stime accurate è reso più difficile dal fatto che i differenziali di fitness divergono e diventano estremi(Wiser e Lenski, 2015). Sospettiamo che le stime empiriche di s utilizzando co-inoculazioni competitive possano sottovalutare notevolmente la forza della selezione in questo sistema, non solo a causa del differenziale di fitness estremo e divergente tra ceppi antenati ed evoluti, ma anche per la difficoltà imposta dal recupero e le sfide di un’accurata enumerazione di genotipi rari.
Aggregazione batterica
La valutazione delle capacità di MJ11 e della variante binK1 di formare aggregati cellulari nel muco dei calamari prima dell’ingresso attraverso i condotti è stata condotta come descritto in precedenza (Nyholm eMcFall-Ngai, 2003). In breve, 1,5 ore dopo che i calamari appena nati sono stati inoculati con ~ 105 CFU / ml GFP etichettati ceppi di interesse (che ospitano pKV111[Nyholm et al., 2000]), i calamari sono stati incubati in 1 uM CellTracker Orange (Invitrogen) per 30 minuti, anestetizzati con cloruro di magnesio isotonico e sezionati rimuovendo il mantello per esporre l’organo leggero intatto. Gli animali sezionati sono stati poi prontamente ripresi a 20X e 40X utilizzando un microscopio confocale a scansione laser Zeiss 510. N = 15-20 calamari testati per ceppo.
Quantificazione del biofilm
La produzione di biofilm è stata quantificata utilizzando un test standard con piccole modifiche (O’Toole, 2011). In breve, una colonia di batteri da una piastra di agar è stata inoculata in 150 μl (in una piastra Costar a 96 pozzetti) o 2 mL (in un tubo di vetro da 15 mm) di SWTO e cresciuta agitando a 200 rpm per 17 ore a 28°C. Il biofilm che è rimasto dopo l’espulsione del liquido, il risciacquo, e la fissazione di calore a 80 ° C per 10 minuti è stato colorato con 0,1% di cristallo viola e poi decolorata in un volume di 200 µl per i saggi in piastra o 2 mL per i saggi in tubo. La produzione di biofilm è stata determinata mediante assorbanza a 550 nm utilizzando un lettore di piastre Tecan Infinite M200. Gli esperimenti sono stati eseguiti in triplice copia e conteneva 3-5 repliche biologiche per trattamento. Le differenze nei mezzi sono state valutate per la significatività utilizzando un test di permutazione Fisher-Pitman a due campioni condotto utilizzando l’esatta distribuzione con il metodo ‘oneway_test’ dalla moneta del pacchetto in R (Hothornet al., 2008).
Sopravvivenza del perossido di idrogeno
I ceppi sono stati coltivati in mezzi LBS a 28°C con scuotimento a 200 rpm fino a quando le colture raggiungono un OD600 tra 1 e 1,5, le colture sono state normalizzate a un OD600di 1,0 per diluizione e 5 μl è stato soggetto, in triplice copia, all’esposizione al perossido di idrogeno a diverse concentrazioni (che vanno da 0,02% a 0,18%) in 200 μl di mezzi LBS in una piastra di polistirolo Costar a 96 pozzetti. Le concentrazioni minime di perossido di idrogeno che hanno limitato tutta la crescita (MIC) di wild-type MJ11 e ES114 dopo l’incubazione durante la notte sono state determinate per ogni lotto di perossido di idrogeno. Le concentrazioni sperimentali variavano dallo 0,02% allo 0,18%. Differenze nella sopravvivenza del ceppo (risultati binomiali) di almeno tre esperimenti combinati che contenevano 106 repliche di ceppi senza plasmidi, 15 repliche di varianti ∆sypK che sono state saggiate insieme a ceppi di controllo privi della mutazione (MJ11, binK1 , ∆ binK) e 50 replicati di ceppi con plasmidi sono stati valutati per la significatività usando esattamente i test di permutazione di Fisher-Pitman con il metodo “oneway_test” del pacchetto R “coin” (Hothorn et al., 2008). Il plasmide che ospitava pRF2A3 (binA)è stato analizzato 20 volte nello stesso esperimento di ceppi di controllo che ospitavano pVSV105 e pCLD48 (sypE), che è stato valutato nello stesso modo.
Legame degli emociti ospitanti dei batteri
Calamaro macrofago-come emociti sono stati isolati da calamari aposymbiotic hatchling calamari utilizzando l’adesione di vetro e poi colorati con Cell Tracker Orange (Invitrogen) sospeso in Squid-Ringers, prima dell’esposizione a GFP etichettati V. fischeri cellule secondo un protocollo precedentemente dettagliato (Nyholm etal., 2009; Collins e Nyholm, 2010), con modifiche comunicate dal dottor Bethany Rader. Gli emociti sono stati esposti per un’ora ai ceppi di V. fischeri ES114, MJ11(binK+), MJ11EP2-4-1(binK1) o non simbiotico Vibrio harveyi B392, che trasporta il plasmide GFP pKV111 (Nyholmet al., 2000). Per testare l’effetto del biofilm Syp sul legame degli emociti, sono stati condotti ulteriori saggi utilizzando ceppi marcati GFP con plasmide di controllo (pVSV104), plasmide di espressione sypE (pRF2A1), o plasmide di espressione binA (pRF2A4) oltre al plasmide GFP (pKV111) (Nyholmet al., 2000)(Tabella1). Dopo l’esposizione, la risposta degli emociti ai batteri è stata visualizzata con un ingrandimento di 63x mediante microscopia confocale e contrasto di interferenza differenziale utilizzando uno Zeiss LSM 510. Il legame degli emociti è stato quantificato mediante l’enumerazione del Vibrio legato rispetto al Vibrio totale entro un raggio di 60 μm che circonda ogni cellula. Sono state quantificate almeno 30 interazioni di emociti per ceppo. Differenze significative nel legame proporzionale medio tra i ceppi sono state rilevate usando un test di indipendenza basato sulla permutazione nel pacchetto R “moneta” (metodo “independence_test”, usando la distribuzione esatta) (Hothorn et al.,2008).
Produzione Siderophore
Sideroforo è stato misurato qualitativamente come un alone arancione che appare intorno alle cellule coltivate su agar CAS(Graf e Ruby, 2000) o da supernatanti liberi da cellule dopo 17 ore di crescita in condizioni di ferro limitate utilizzando un test liquido crom-azurol S(Lee e Ruby, 1994a; Payne, 1994b). La riduzione colorimetrica in OD630 è stata misurata in un lettore di piastre Tecan Infinite M200 e le unità % siderohpore sono state calcolate e normalizzate dalla densità cellulare (Leee Ruby, 1994a). Le unità sideroforo erano al di sotto del limite di rilevazione per MJ11 e il suo derivato binK1 ma non ES114.
Luminescenza, lattone omoserico e determinazione della densità cellulare
Luminescenza, densità cellulare e lattoni omoserini sono stati quantificati da V. fischeri MJ11 e varianti cresciute in un volume iniziale di 15 mL di coltura di brodo SWT in un pallone da 125 ml, che è diminuito progressivamente in volume con il campionamento. La luminescenza prodotta dall’equivalente di 1 mL di coltura è stata quantificata su cellule diluite fino a 1:1000, per garantire che le misurazioni fossero entro il range di rilevazione, con un luminometro Turner 20/20 (Turner Designs, Sunnyvale, CA). Contemporaneamente, la densità ottica (OD600) è stata determinata con un Biofotometro (Eppendorf AG, Amburgo, Germania), con celle diluite in mezzo. In parallelo, le unità di formazione delle colonie sono state determinate mediante diluizione seriale standard e placcatura su agar LBS. Metodi pubblicati sono stati utilizzati per la purificazione e la quantificazione di N-(3-oxohexanoyl) lattone omoserina (3-oxo-C6-HSL) e N-octanoyl lattone omoserina (C8-HSL)(Schaefer et al., 2000; Duerkop et al., 2007). In breve, acyl-HSLs sono stati estratti due volte con un volume uguale di acetato di etile acidificato da supernatanti privi di cellule di MJ11 e derivati campionati a diversi livelli di OD600-rappresentazione mid-log (OD600~ 0.7 e 1,0), tardo-log (~ 1,7), stazionario precoce (~ 3,5), e fase stazionaria (~ 5,3-8)-per valutare il range dinamico di sintesi AHL per ogni derivato e per determinare l’OD600 ottimale durante l’induzione. AHL sono stati estratti e concentrati da 0,5 a 5 mL di MJ11 e le varianti erano rilevabili e all’interno del range lineare del test, identificando che un OD600di ~ 1,0 era ottimale. Esperimenti replicati sono stati eseguiti in cui OD600è stato monitorato a intervalli regolari, e AHL sono stati immediatamente estratti quando le colture hanno raggiunto un OD600 di 0,9-1,4. Qualsiasi coltura di derivati binK identificata come dominata da mutanti soppressori (cioè che mostravano un tasso di crescita anormalmente veloce accompagnato da una luminescenza maggiore di quella selvaggia e da un’alta percentuale di colonie di grandi dimensioni quando placcate) è stata scartata. I campioni estratti sono stati concentrati per evaporazione sotto azoto anidro prima dell’analisi. 3-oxo-C6-HSL è stato quantificato utilizzando il ceppo reporter E. coli VJS533 che ospita il plasmide pHV200I-, che risponde a 3-oxo-C6-HSL producendo luminescenza (Pearsonet al., 1994). C8-HSL è stato quantificato utilizzando il ceppo reporter E. coli MG4 che ospita pQF50(bmaI1-lacZ promotore di fusione derivato da Burkhoderia mallei) e pJN105 (un gene R arabino-inducibile), che esprime la lacZ specificamente in risposta al C8-HSL esogeno con bassa sensibilità al 3-oxo-C6-HSL(Duerkop et al., 2007). L’attività di lacZ è stata misurata con un saggio standard(Miller, 1972) e utilizzando il sistema di analisi genica Dual-Light Luciferase e β-Galattosidasi Reporter Gene Assay System (Applied Biosystems). Le quantità di 3-oxo-C6-HSL e C8-HSL sono state determinate confrontando l’attività misurata da una serie di diluizioni dei campioni estratti con il range lineare (R2 ≥0,98) di ogni curva standard generata da substrati sintetici (N-(ß-ketocaproyl)-L-homoserine lactone e N-octanoyl-L-homoserine lactone) (Cayman Chemical). Un totale di 10 colture per ogni derivato da cinque esperimenti combinati sono stati analizzati e riportati con l’eccezione del CFU, che era da tre colture. Differenze in CFU/mL/OD600, OD600, nM 3-oxo-C6, nM C8-HSL, e luminescenza (Lum) per 1 mL di coltura per ogni variante segnalata rispetto a MJ11 sono stati testati per la significatività utilizzando precisi test di permutazione Fisher-Pitman nel pacchetto R ‘moneta’ (metodo “oneway_test”) (Hothorn et al.,2008).
Profilazione metabolica
Fenotipo MicroArray (Biolog, Hayward, CA) PM1 e PM2A sono stati eseguiti secondo i protocolli dei produttori(Bochner et al., 2001) con poche modifiche per l’analisi V. fischeri, in particolare compresa l’integrazione di IF-0 con 1% di NaCl. In breve, per ogni ceppo, è stato preparato un inoculo sufficiente per due piastre di replicazione, recuperando e mescolando le colonie batteriche in 16 ml IF-0 per ottenere una sospensione uniforme a OD600 0,175 e mescolato con la miscela di colorante D (diluizioni 1:5). PM1 e PM2A duplicato (ES114, binK1- e ΔbinK-varianti) o triplicato (MJ11 e bianco) piastre sono stati inoculati con 100 microlitri di sospensione per pozzetto, e incubato a 28 ° C per 48 ore OD490 è statoregistrato da un lettore di micropiastre Tecan Infinite M200 ogni 4 ore per misurare le variazioni cinetiche di colore (stato redox) del colorante D. Per determinare quali substrati hanno suscitato diverse risposte cinetiche tra i ceppi, abbiamo eseguito un ANOVA sui valori di OD490 dopo la normalizzazione rispetto ai valori di controllo del bianco per ogni misurazione temporizzata. L’importanza delle differenze di attività della deformazione per qualsiasi substrato è stata determinata dopo la correzione per prove multiple usando un tasso di falso rilevamento di 0,05. Per quantificare il significato complessivo delle risposte metaboliche per MJ11 binK1 e MJ11 ∆binK convergenti con ES114 e divergenti da MJ11, abbiamo usato l’Exact Binomial Test nell’ipotesi nulla che solo il 12,5% dei substrati dovrebbe produrre un tale modello attraverso i quattro ceppi analizzati (2*0,54) con il metodo R ‘binom.test’.
Analisi statistiche
Se non diversamente specificato, le risposte differenziali alla colonizzazione e i saggi sperimentali per i diversi ceppi sono stati testati usando esattamente i test di permutazione Fisher-Pitman con il ‘oneway_test’ del pacchetto R ‘coin’ (Hothorn et al., 2008). I risultati degli esperimenti condotti in triplice copia sono stati combinati con l’inclusione di una variabile di blocco per tener conto di potenziali artefatti tecnici.
References
- Altura MA, Heath-Heckman EA, Gillette A, Kremer N, Krachler AM, Brennan C, Ruby EG, Orth K, McFall-Ngai MJ. The first engagement of partners in the Euprymna scolopes-Vibrio fischeri symbiosis is a two-step process initiated by a few environmental symbiont cells. Environmental Microbiology. 2013; 15:2937-2950. DOI | PubMed
- An JH, Goo E, Kim H, Seo YS, Hwang I. Bacterial quorum sensing and metabolic slowing in a cooperative population. PNAS. 2014; 111:14912-14917. DOI | PubMed
- Anantharaman V, Aravind L. Cache – a signaling domain common to animal ca(2+)-channel subunits and a class of prokaryotic chemotaxis receptors. Trends in Biochemical Sciences. 2000; 25:535-537. DOI | PubMed
- Bassis CM, Visick KL. The cyclic-di-GMP phosphodiesterase BinA negatively regulates cellulose-containing biofilms in Vibrio fischeri. Journal of Bacteriology. 2010; 192:1269-1278. DOI | PubMed
- Baym M, Kryazhimskiy S, Lieberman TD, Chung H, Desai MM, Kishony R. Inexpensive multiplexed library preparation for megabase-sized genomes. PLoS One. 2015; 10DOI | PubMed
- Bedhomme S, Lafforgue G, Elena SF. Multihost experimental evolution of a plant RNA virus reveals local adaptation and host-specific mutations. Molecular Biology and Evolution. 2012; 29:1481-1492. DOI | PubMed
- Bertels F, Silander OK, Pachkov M, Rainey PB, van Nimwegen E. Automated reconstruction of whole-genome phylogenies from short-sequence reads. Molecular Biology and Evolution. 2014; 31:1077-1088. DOI | PubMed
- Bochner BR, Gadzinski P, Panomitros E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Research. 2001; 11:1246-1255. DOI | PubMed
- Boettcher KJ, Ruby EG. Depressed light emission by symbiotic Vibrio fischeri of the sepiolid squid Euprymna scolopes. Journal of Bacteriology. 1990; 172:3701-3706. DOI | PubMed
- Boettcher KJ, Ruby EG, McFall-Ngai MJ. Bioluminescence in the symbiotic squid Euprymna scolopes is controlled by a daily biological rhythm. Journal of Comparative Physiology A. 1996; 179:65-73. DOI
- Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014; 30:2114-2120. DOI | PubMed
- Bose JL, Kim U, Bartkowski W, Gunsalus RP, Overley AM, Lyell NL, Visick KL, Stabb EV. Bioluminescence in Vibrio fischeri is controlled by the redox-responsive regulator ArcA. Molecular Microbiology. 2007; 65:538-553. DOI | PubMed
- Brooks JF, Gyllborg MC, Kocher AA, Markey LE, Mandel MJ. TfoX-based genetic mapping identifies Vibrio fischeri strain-level differences and reveals a common lineage of laboratory strains. Journal of Bacteriology. 2015; 197:1065-1074. DOI | PubMed
- Brooks JF, Mandel MJ. The Histidine kinase BinK is a negative regulator of biofilm formation and squid colonization. Journal of Bacteriology. 2016; 198:2596-2607. DOI | PubMed
- Caley MJ, Munday PL. Growth trades off with habitat specialization. Proceedings of the Royal Society B: Biological Sciences. 2003; 270 Suppl 2:S175-S177. DOI | PubMed
- Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, Madden TL. BLAST+: architecture and applications. BMC Bioinformatics. 2009; 10:421-429. DOI | PubMed
- Capra EJ, Laub MT. Evolution of two-component signal transduction systems. Annual Review of Microbiology. 2012; 66:325-347. DOI | PubMed
- Chevin LM. On measuring selection in experimental evolution. Biology Letters. 2011; 7:210-213. DOI | PubMed
- Chu H, Mazmanian SK. Innate immune recognition of the Microbiota promotes host-microbial symbiosis. Nature Immunology. 2013; 14:668-675. DOI | PubMed
- Collins AJ, Nyholm SV. Obtaining hemocytes from the hawaiian bobtail squid Euprymna scolopes and observing their adherence to symbiotic and non-symbiotic bacteria. Journal of Visualized Experiments. 2010. DOI | PubMed
- Dandekar AA, Chugani S, Greenberg EP. Bacterial quorum sensing and metabolic incentives to cooperate. Science. 2012; 338:264-266. DOI | PubMed
- Darnell CL, Hussa EA, Visick KL. The putative hybrid sensor kinase SypF coordinates biofilm formation in Vibrio fischeri by acting upstream of two response regulators, SypG and VpsR. Journal of Bacteriology. 2008; 190:4941-4950. DOI | PubMed
- Davenport PW, Griffin JL, Welch M. Quorum sensing is accompanied by global metabolic changes in the Opportunistic Human Pathogen Pseudomonas aeruginosa. Journal of Bacteriology. 2015; 197:2072-2082. DOI | PubMed
- Davidson SK, Koropatnick TA, Kossmehl R, Sycuro L, McFall-Ngai MJ. NO means ‘yes’ in the squid-vibrio symbiosis: nitric oxide (NO) during the initial stages of a beneficial association. Cellular Microbiology. 2004; 6:1139-1151. DOI | PubMed
- Deatherage DE, Barrick JE. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods in Molecular Biology. 2014; 1151:165-188. DOI | PubMed
- Dillon MM, Sung W, Sebra R, Lynch M, Cooper VS. Genome-Wide biases in the rate and molecular spectrum of spontaneous mutations in Vibrio cholerae and Vibrio fischeri. Molecular Biology and Evolution. 2017; 34:93-109. DOI | PubMed
- Duerkop BA, Ulrich RL, Greenberg EP. Octanoyl-homoserine lactone is the cognate signal for Burkholderia mallei BmaR1-BmaI1 quorum sensing. Journal of Bacteriology. 2007; 189:5034-5040. DOI | PubMed
- Dunn AK, Millikan DS, Adin DM, Bose JL, Stabb EV. New rfp- and pES213-derived tools for analyzing symbiotic Vibrio fischeri reveal patterns of infection and lux expression in situ. Applied and Environmental Microbiology. 2006; 72:802-810. DOI | PubMed
- Fidopiastis PM, Miyamoto CM, Jobling MG, Meighen EA, Ruby EG, LitR JMG. LitR, a new transcriptional activator in Vibrio fischeri, regulates luminescence and symbiotic light organ colonization. Molecular Microbiology. 2002; 45:131-143. DOI | PubMed
- Fisher RA. The Genetical Theory of Natural Selection — A Complete Variorum Edition. Oxford University Press; 1930.
- Foster JS, Apicella MA, McFall-Ngai MJ. Vibrio fischeri lipopolysaccharide induces developmental apoptosis, but not complete morphogenesis, of the Euprymna scolopes symbiotic light organ. Developmental Biology. 2000; 226:242-254. DOI | PubMed
- Foxall RL, Ballok AE, Avitabile A, Whistler CA. Spontaneous phenotypic suppression of GacA-defective Vibrio fischeri is achieved via mutation of csrA and ihfA. BMC Microbiology. 2015; 15DOI | PubMed
- Gao R, Stock AM. Evolutionary tuning of protein expression levels of a positively autoregulated two-component system. PLoS Genetics. 2013; 9DOI | PubMed
- Graf J, Dunlap PV, Ruby EG. Effect of transposon-induced motility mutations on colonization of the host light organ by Vibrio fischeri. Journal of Bacteriology. 1994; 176:6986-6991. DOI | PubMed
- Graf J, Ruby EG. Host-derived amino acids support the proliferation of symbiotic bacteria. PNAS. 1998; 95:1818-1822. DOI | PubMed
- Graf J, Ruby EG. Novel effects of a transposon insertion in the Vibrio fischeri glnD gene: defects in iron uptake and symbiotic persistence in addition to nitrogen utilization. Molecular Microbiology. 2000; 37:168-179. DOI | PubMed
- Guan SH, Gris C, Cruveiller S, Pouzet C, Tasse L, Leru A, Maillard A, Médigue C, Batut J, Masson-Boivin C, Capela D. Experimental evolution of nodule intracellular infection in legume symbionts. The ISME Journal. 2013; 7:1367-1377. DOI | PubMed
- Guerrero-Ferreira RC, Nishiguchi MK. Biodiversity among luminescent symbionts from squid of the genera Uroteuthis, Loliolus and Euprymna (Mollusca: Cephalopoda). Cladistics. 2007; 23:497-506. DOI | PubMed
- Haygood MG, Tebo BM, Nealson KH. Luminous bacteria of a monocentrid fish (Monocentris japonicus) and two anomalopid fishes (Photoblepharon palpebratus and kryptophanaron alfredi): population sizes and growth within the light organs, and rates of release into the seawater. Marine Biology. 1984; 78:249-254. DOI
- Heath-Heckman EA, McFall-Ngai MJ. The occurrence of chitin in the hemocytes of invertebrates. Zoology. 2011; 114:191-198. DOI | PubMed
- Herrero M, de Lorenzo V, Timmis KN. Transposon vectors containing non-antibiotic resistance selection markers for cloning and stable chromosomal insertion of foreign genes in gram-negative bacteria. Journal of Bacteriology. 1990; 172:6557-6567. DOI | PubMed
- Horton RM, Cai ZL, Ho SN, Pease LR. Gene splicing by overlap extension: tailor-made genes using the polymerase chain reaction. BioTechniques. 1990; 8:528-535. DOI | PubMed
- Hothorn T, Hornik K, van de Wiel MA, Zeileis A. Implementing a class of Permutation tests: the coin package. Journal of Statistical Software. American Statistical Association. 2008; 28:1-23. DOI
- Hsieh YC, Liang SM, Tsai WL, Chen YH, Liu TY, Liang CM. Study of capsular polysaccharide from Vibrio parahaemolyticus. Infection and Immunity. 2003; 71:3329-3336. DOI | PubMed
- Hussa EA, Darnell CL, Visick KL. RscS functions upstream of SypG to control the syp locus and biofilm formation in Vibrio fischeri. Journal of Bacteriology. 2008; 190:4576-4583. DOI | PubMed
- Jacob F. Evolution and tinkering. Science. 1977; 196:1161-1166. DOI | PubMed
- Jansen G, Crummenerl LL, Gilbert F, Mohr T, Pfefferkorn R, Thänert R, Rosenstiel P, Schulenburg H. evolutionary transition from pathogenicity to commensalism: global regulator mutations mediate fitness gains through virulence attenuation. Molecular Biology and Evolution. 2015; 32:2883-2896. DOI | PubMed
- Jones BW, Nishiguchi MK. Counterillumination in the hawaiian bobtail squid, Euprymna scolopes Berry (Mollusca: cephalopoda). Marine Biology. 2004; 144:1151-1155. DOI
- Jones SE, Paynich ML, Kearns DB, Knight KL. Protection from intestinal inflammation by bacterial exopolysaccharides. The Journal of Immunology. 2014; 192:4813-4820. DOI | PubMed
- Katoh K, Misawa K, Kuma K, Miyata T. MAFFT: a novel method for rapid multiple sequence alignment based on fast fourier transform. Nucleic Acids Research. 2002; 30:3059-3066. DOI | PubMed
- Kawecki TJ, Lenski RE, Ebert D, Hollis B, Olivieri I, Whitlock MC. Experimental evolution. Trends in Ecology & Evolution. 2012; 27:547-560. DOI | PubMed
- Kimbrough JH, Stabb EV. Antisocial luxO mutants provide a stationary-phase survival advantage in Vibrio fischeri ES114. Journal of Bacteriology. 2015; 198:673-687. DOI | PubMed
- Koch EJ, Miyashiro T, McFall-Ngai MJ, Ruby EG. Features governing symbiont persistence in the squid-Vibrio association. Molecular Ecology. 2014; 23:1624-1634. DOI | PubMed
- Kolter R, Helinski DR. Construction of plasmid R6K derivatives in vitro: characterization of the R6K replication region. Plasmid. 1978; 1:571-580. DOI | PubMed
- Koropatkin NM, Cameron EA, Martens EC. How glycan metabolism shapes the human gut microbiota. Nature Reviews Microbiology. 2012; 10:323-335. DOI | PubMed
- Koropatnick TA, Engle JT, Apicella MA, Stabb EV, Goldman WE, McFall-Ngai MJ. Microbial factor-mediated development in a host-bacterial mutualism. Science. 2004; 306:1186-1188. DOI | PubMed
- Koropatnick TA, Kimbell JR, McFall-Ngai MJ. Responses of host hemocytes during the initiation of the squid-Vibrio symbiosis. The Biological Bulletin. 2007; 212:29-39. DOI | PubMed
- Kuo A, Callahan SM, Dunlap PV. Modulation of luminescence operon expression by N-octanoyl-L-homoserine lactone in ainS mutants of Vibrio fischeri. Journal of Bacteriology. 1996; 178:971-976. DOI | PubMed
- Kwong WK, Moran NA. Evolution of host specialization in gut microbes: the bee gut as a model. Gut Microbes. 2015; 6:214-220. DOI | PubMed
- Langmead B, Salzberg SL. Fast gapped-read alignment with bowtie 2. Nature Methods. 2012; 9:357-359. DOI | PubMed
- Lee KH, Ruby EG. Detection of the light organ symbiont, Vibrio fischeri, in Hawaiian seawaters by using lux gene probes. Applied and Environmental Microbiology. 1992; 58:942-947. PubMed
- Lee KH, Ruby EG. Competition between Vibrio fischeri strains during initiation and maintenance of a light organ symbiosis. Journal of Bacteriology. 1994a; 176:1985-1991. DOI | PubMed
- Lee KH, Ruby EG. Effect of the squid host on the abundance and distribution of symbiotic Vibrio fischeri in nature. Applied and Environmental Microbiology. 1994b; 60PubMed
- Lee KH. Ecology of Vibrio Fischeri, the Light Organ Symbiont of the Hawaiian Sepiolid Squid Euprymna Scolopes. University of Southern California; 1994.
- Lee CE, Gelembiuk GW. Evolutionary origins of invasive populations. Evolutionary Applications. 2008; 1:427-448. DOI | PubMed
- Lenski RE, Rose MR, Simpson SC, Tadler SC. Long-Term experimental evolution in Escherichia coli. I. adaptation and divergence during 2,000 generations. The American Naturalist. 1991; 138:1315-1341. DOI
- Lenski RE, Travisano M. Dynamics of adaptation and diversification: a 10,000-generation experiment with bacterial populations. PNAS. 1994; 91:6808-6814. DOI | PubMed
- Li B, Dewey CN. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 2011; 12DOI | PubMed
- Lupp C, Urbanowski M, Greenberg EP, Ruby EG. The Vibrio fischeri quorum-sensing systems Ain and Lux sequentially induce luminescence gene expression and are important for persistence in the squid host. Molecular Microbiology. 2003; 50:319-331. DOI | PubMed
- Lupp C, Ruby EG. Vibrio fischeri LuxS and AinS: comparative study of two signal synthases. Journal of Bacteriology. 2004; 186:3873-3881. DOI | PubMed
- Lyell NL, Dunn AK, Bose JL, Vescovi SL, Stabb EV. Effective mutagenesis of Vibrio fischeri by using hyperactive mini-Tn5 derivatives. Applied and Environmental Microbiology. 2008; 74:7059-7063. DOI | PubMed
- Macmanes MD. On the optimal trimming of high-throughput mRNA sequence data. Frontiers in Genetics. 2014; 5DOI | PubMed
- Maindonald JH, Braun WJ. DAAG: data analysis and graphics data and functions. 2015. Publisher Full Text
- Mandel MJ, Wollenberg MS, Stabb EV, Visick KL, Ruby EG. A single regulatory gene is sufficient to alter bacterial host range. Nature. 2009; 458:215-218. DOI | PubMed
- Mandel MJ, Schaefer AL, Brennan CA, Heath-Heckman EA, Deloney-Marino CR, McFall-Ngai MJ, Ruby EG. Squid-derived chitin oligosaccharides are a chemotactic signal during colonization by Vibrio fischeri. Applied and Environmental Microbiology. 2012; 78:4620-4626. DOI | PubMed
- Marchetti M, Capela D, Glew M, Cruveiller S, Chane-Woon-Ming B, Gris C, Timmers T, Poinsot V, Gilbert LB, Heeb P, Médigue C, Batut J, Masson-Boivin C. Experimental evolution of a plant pathogen into a legume symbiont. PLoS Biology. 2010; 8DOI | PubMed
- Martín-Mora D, Ortega A, Reyes-Darias JA, García V, López-Farfán D, Matilla MA, Krell T. Identification of a chemoreceptor in Pseudomonas aeruginosa that specifically mediates chemotaxis toward α-Ketoglutarate. Frontiers in Microbiology. 2016; 7DOI | PubMed
- Mazmanian SK, Round JL, Kasper DL. A microbial symbiosis factor prevents intestinal inflammatory disease. Nature. 2008; 453:620-625. DOI | PubMed
- Miller JH. Experiments in Molecular Genetics. Cold Spring Harbor Laboratory: Cold Spring Harbor. New York: NY; 1972.
- Mitrophanov AY, Groisman EA. Signal integration in bacterial two-component regulatory systems. Genes & Development. 2008; 22:2601-2611. DOI | PubMed
- Miyashiro T, Wollenberg MS, Cao X, Oehlert D, Ruby EG. A single qrr gene is necessary and sufficient for LuxO-mediated regulation in Vibrio fischeri. Molecular Microbiology. 2010; 77:1556-1567. DOI | PubMed
- Miyashiro T, Klein W, Oehlert D, Cao X, Schwartzman J, Ruby EG. The N-acetyl-D-glucosamine repressor NagC of Vibrio fischeri facilitates colonization of Euprymna scolopes. Molecular Microbiology. 2011; 82:894-903. DOI | PubMed
- Miyashiro T, Ruby EG. Shedding light on bioluminescence regulation in Vibrio fischeri. Molecular Microbiology. 2012; 84:795-806. DOI | PubMed
- Miyashiro T, Oehlert D, Ray VA, Visick KL, Ruby EG. The putative oligosaccharide translocase SypK connects biofilm formation with quorum signaling in Vibrio fischeri. MicrobiologyOpen. 2014; 3:836-848. DOI | PubMed
- Morley VJ, Mendiola SY, Turner PE. Rate of novel host invasion affects adaptability of evolving RNA virus lineages. Proceedings of the Royal Society B: Biological Sciences. 2015; 282:20150801-20150807. DOI | PubMed
- Morris AR, Visick KL. The response regulator SypE controls biofilm formation and colonization through phosphorylation of the syp-encoded regulator SypA in Vibrio fischeri. Molecular Microbiology. 2013; 87:509-525. DOI | PubMed
- Nguyen LT, Schmidt HA, von Haeseler A, Minh BQ. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Molecular Biology and Evolution. 2015; 32:268-274. DOI | PubMed
- Nishiguchi MK, Ruby EG, McFall-Ngai MJ. Competitive dominance among strains of luminous bacteria provides an unusual form of evidence for parallel evolution in sepiolid squid-Vibrio symbioses. Applied and Environmental Microbiology. 1998; 64PubMed
- Nishiguchi MK. Host-symbiont recognition in the environmentally transmitted sepiolid squid-Vibrio mutualism. Microbial Ecology. 2002; 44:10-18. DOI | PubMed
- Nizet V, Esko JD. Essentials of Glycobiology. Cold Spring Harbor Laboratory Press: New York; 2009.
- Nyholm SV, McFall-Ngai MJ. Sampling the light-organ microenvironment of Euprymna scolopes: description of a population of host cells in association with the bacterial symbiont Vibrio fischeri. The Biological Bulletin. 1998; 195:89-97. DOI | PubMed
- Nyholm SV, Stabb EV, Ruby EG, McFall-Ngai MJ. Establishment of an animal-bacterial association: recruiting symbiotic vibrios from the environment. PNAS. 2000; 97:10231-10235. DOI | PubMed
- Nyholm SV, McFall-Ngai MJ. Dominance of Vibrio fischeri in secreted mucus outside the light organ of Euprymna scolopes: the first site of symbiont specificity. Applied and Environmental Microbiology. 2003; 69:3932-3937. DOI | PubMed
- Nyholm SV, McFall-Ngai MJ. The winnowing: establishing the squid-vibrio symbiosis. Nature Reviews Microbiology. 2004; 2:632-642. DOI | PubMed
- Nyholm SV, Stewart JJ, Ruby EG, McFall-Ngai MJ. Recognition between symbiotic Vibrio fischeri and the haemocytes of Euprymna scolopes. Environmental Microbiology. 2009; 11:483-493. DOI | PubMed
- O’Toole GA. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. 2011. DOI | PubMed
- Ochman H, Moran NA. Genes lost and genes found: evolution of bacterial pathogenesis and symbiosis. Science. 2001; 292:1096-1099. DOI | PubMed
- Orr HA. The rate of adaptation in asexuals. Genetics. 2000; 155:961-968. PubMed
- Orr HA. The distribution of fitness effects among beneficial mutations. Genetics. 2003; 163:1519-1526. PubMed
- Pan M, Schwartzman JA, Dunn AK, Lu Z, Ruby EG. A single host-derived glycan impacts key regulatory nodes of symbiont metabolism in a coevolved mutualism. mBio. 2015; 6DOI | PubMed
- Pasek S, Risler JL, Brézellec P. Gene fusion/fission is a Major contributor to evolution of multi-domain bacterial proteins. Bioinformatics. 2006; 22:1418-1423. DOI | PubMed
- Payne SM. Detection, isolation, and characterization of siderophores. Methods in Enzymology. 1994a; 235:329-344. DOI | PubMed
- Payne SM. Bacterial Pathogenesis Part A: Identification and Regulation of Virulence Factors. Elsevier; 1994b.
- Pearson JP, Gray KM, Passador L, Tucker KD, Eberhard A, Iglewski BH, Greenberg EP. Structure of the autoinducer required for expression of Pseudomonas aeruginosa virulence genes. PNAS. 1994; 91:197-201. DOI | PubMed
- Perry LA. Experimental Evolution of Vibrio Fischeri to Squid Symbiosis. University of New Hampshire; 2009.
- Ray VA, Driks A, Visick KL. Identification of a novel matrix protein that promotes biofilm maturation in Vibrio fischeri. Journal of Bacteriology. 2015; 197:518-528. DOI | PubMed
- Reuter S, Connor TR, Barquist L, Walker D, Feltwell T, Harris SR, Fookes M, Hall ME, Petty NK, Fuchs TM, Corander J, Dufour M, Ringwood T, Savin C, Bouchier C, Martin L, Miettinen M, Shubin M, Riehm JM, Laukkanen-Ninios R, Sihvonen LM, Siitonen A, Skurnik M, Falcão JP, Fukushima H, Scholz HC, Prentice MB, Wren BW, Parkhill J, Carniel E, Achtman M, McNally A, Thomson NR. Parallel independent evolution of pathogenicity within the genus Yersinia. PNAS. 2014; 111:6768-6773. DOI | PubMed
- Robinson MD, McCarthy DJ, Smyth GK. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 2010; 26:139-140. DOI | PubMed
- Rowland MA, Deeds EJ. Crosstalk and the evolution of specificity in two-component signaling. PNAS. 2014; 111:5550-5555. DOI | PubMed
- Ruby EG, Nealson KH. Pyruvate production and excretion by the luminous marine bacteria. Applied and Environmental Microbiology. 1977; 34:164-169. PubMed
- Ruby EG, Asato LM. Growth and flagellation of Vibrio fischeri during initiation of the sepiolid squid light organ symbiosis. Archives of Microbiology. 1993; 159:160-167. DOI | PubMed
- Ruby EG, Lee KH. The Vibrio fischeri-Euprymna scolopes light organ association: current ecological paradigms. Applied and Environmental Microbiology. 1998; 64:805-812. PubMed
- Ruby EG, McFall-Ngai MJ. Oxygen-utilizing reactions and symbiotic colonization of the squid light organ by Vibrio fischeri. Trends in Microbiology. 1999; 7:414-420. DOI | PubMed
- Saccheri I, Hanski I. Natural selection and population dynamics. Trends in Ecology & Evolution. 2006; 21:341-347. DOI | PubMed
- Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. NY: Cold Spring Harbor Laboratory Press: Cold Spring Harbor; 1989.
- Schaefer AL, Hanzelka BL, Eberhard A, Greenberg EP. Quorum sensing in Vibrio fischeri: probing autoinducer-LuxR interactions with autoinducer analogs. Journal of Bacteriology. 1996; 178:2897-2901. DOI | PubMed
- Schaefer AL, Hanzelka BL, Parsek MR, Greenberg EP, Detection GEP. Detection, purification, and structural elucidation of the acylhomoserine lactone inducer of Vibrio fischeri luminescence and other related molecules. Methods in Enzymology. 2000; 305:288-301. DOI | PubMed
- Schuster BM, Perry LA, Cooper VS, Whistler CA. Breaking the language barrier: experimental evolution of non-native Vibrio fischeri in squid tailors luminescence to the host. Symbiosis. 2010; 51:85-96. DOI
- Schuster M, Sexton DJ, Diggle SP, Greenberg EP, Joseph Sexton D, Peter Greenberg E. Acyl-homoserine lactone quorum sensing: from evolution to application. Annual Review of Microbiology. 2013; 67:43-63. DOI | PubMed
- Schwartzman JA, Koch E, Heath-Heckman EA, Zhou L, Kremer N, McFall-Ngai MJ, Ruby EG. The chemistry of negotiation: rhythmic, glycan-driven acidification in a symbiotic conversation. PNAS. 2015; 112:566-571. DOI | PubMed
- Seemann T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 2014; 30:2068-2069. DOI | PubMed
- Sengupta R, Altermann E, Anderson RC, McNabb WC, Moughan PJ, Roy NC. The role of cell surface architecture of lactobacilli in host-microbe interactions in the gastrointestinal tract. Mediators of Inflammation. 2013; 2013:1-16. DOI | PubMed
- Septer AN, Wang Y, Ruby EG, Stabb EV, Dunn AK. The haem-uptake gene cluster in Vibrio fischeri is regulated by Fur and contributes to symbiotic colonization. Environmental Microbiology. 2011; 13:2855-2864. DOI | PubMed
- Septer AN, Lyell NL, Stabb EV. The iron-dependent regulator Fur controls pheromone signaling systems and luminescence in the squid symbiont Vibrio fischeri ES114. Applied and Environmental Microbiology. 2013; 79:1826-1834. DOI | PubMed
- Shibata S, Yip ES, Quirke KP, Ondrey JM, Visick KL. Roles of the structural symbiosis polysaccharide (syp) genes in host colonization, biofilm formation, and polysaccharide biosynthesis in Vibrio fischeri. Journal of Bacteriology. 2012; 194:6736-6747. DOI | PubMed
- Small AL, McFall-Ngai MJ. Halide peroxidase in tissues that interact with bacteria in the host squid Euprymna scolopes. Journal of Cellular Biochemistry. 1999; 72:445-457. DOI | PubMed
- Soto W, Rivera FM, Nishiguchi MK. Ecological diversification of Vibrio fischeri serially passaged for 500 generations in novel squid host Euprymna tasmanica. Microbial Ecology. 2014; 67:700-721. DOI | PubMed
- Soto W, Nishiguchi MK. Microbial experimental evolution as a novel research approach in the Vibrionaceae and squid-Vibrio symbiosis. Frontiers in Microbiology. 2014; 5DOI | PubMed
- Stabb EV, Ruby EG. RP4-based plasmids for conjugation between Escherichia coli and members of the Vibrionaceae. Methods in Enzymology. 2002; 358:413-426. DOI | PubMed
- Stamatakis A. RAxML-VI-HPC: maximum likelihood-based phylogenetic analyses with thousands of taxa and mixed models. Bioinformatics. 2006; 22:2688-2690. DOI | PubMed
- Stewart V, Chen LL. The S Helix mediates signal transmission as a HAMP domain coiled-coil extension in the NarX nitrate sensor from Escherichia coli K-12. Journal of Bacteriology. 2010; 192:734-745. DOI | PubMed
- Takemura AF, Chien DM, Polz MF. Associations and dynamics of Vibrionaceae in the environment, from the genus to the population level. Frontiers in Microbiology. 2014; 5DOI | PubMed
- Taylor TB, Mulley G, Dills AH, Alsohim AS, McGuffin LJ, Studholme DJ, Silby MW, Brockhurst MA, Johnson LJ, Jackson RW. Evolution. evolutionary resurrection of flagellar motility via rewiring of the nitrogen regulation system. Science. 2015; 347:1014-1017. DOI | PubMed
- Thurman TJ, Barrett RD. The genetic consequences of selection in natural populations. Molecular Ecology. 2016; 25:1429-1448. DOI | PubMed
- Travisano M, Shaw RG. Lost in the map. Evolution. 2013; 67:305-314. DOI | PubMed
- Verma SC, Miyashiro T. Quorum sensing in the squid-Vibrio symbiosis. International Journal of Molecular Sciences. 2013; 14:16386-16401. DOI | PubMed
- Visick KL, Ruby EG. The periplasmic, group III catalase of Vibrio fischeri is required for normal symbiotic competence and is induced both by oxidative stress and by approach to stationary phase. Journal of Bacteriology. 1998; 180:2087-2092. PubMed
- Visick KL, McFall-Ngai MJ. An exclusive contract: specificity in the Vibrio fischeri-Euprymna scolopes partnership. Journal of Bacteriology. 2000; 182:1779-1787. DOI | PubMed
- Visick KL, Foster J, Doino J, McFall-Ngai M, Ruby EG. Vibrio fischeri lux genes play an important role in colonization and development of the host light organ. Journal of Bacteriology. 2000; 182:4578-4586. DOI | PubMed
- Visick KL. An intricate network of regulators controls biofilm formation and colonization by Vibrio fischeri. Molecular Microbiology. 2009; 74:782-789. DOI | PubMed
- Vogel C, Bashton M, Kerrison ND, Chothia C, Teichmann SA, Structure TSA. Structure, function and evolution of multidomain proteins. Current Opinion in Structural Biology. 2004; 14:208-216. DOI | PubMed
- Vuong C, Voyich JM, Fischer ER, Braughton KR, Whitney AR, DeLeo FR, Otto M. Polysaccharide intercellular adhesin (PIA) protects Staphylococcus epidermidis against major components of the human innate immune system. Cellular Microbiology. 2004; 6:269-275. DOI | PubMed
- Wahl LM, Gerrish PJ. The probability that beneficial mutations are lost in populations with periodic bottlenecks. Evolution. 2001; 55:2606-2610. DOI | PubMed
- Wang Y, Diaz Arenas C, Stoebel DM, Flynn K, Knapp E, Dillon MM, Wünsche A, Hatcher PJ, Moore FB, Cooper VS, Cooper TF. Benefit of transferred mutations is better predicted by the fitness of recipients than by their ecological or genetic relatedness. PNAS. 2016; 113:5047-5052. DOI | PubMed
- Waters CM, Bassler BL. Quorum sensing: cell-to-cell communication in bacteria. Annual Review of Cell and Developmental Biology. 2005; 21:319-346. DOI | PubMed
- Weis VM, Small AL, McFall-Ngai MJ. A peroxidase related to the mammalian antimicrobial protein myeloperoxidase in the Euprymna-Vibrio mutualism. PNAS. 1996; 93:13683-13688. DOI | PubMed
- Whistler CA, Ruby EG. GacA regulates symbiotic colonization traits of Vibrio fischeri and facilitates a beneficial association with an animal host. Journal of Bacteriology. 2003; 185:7202-7212. DOI | PubMed
- Whistler CA, Koropatnick TA, Pollack A, McFall-Ngai MJ, Ruby EG. The GacA global regulator of Vibrio fischeri is required for normal host tissue responses that limit subsequent bacterial colonization. Cellular Microbiology. 2007; 9:766-778. DOI | PubMed
- Whitehead NA, Barnard AM, Slater H, Simpson NJ, Salmond GP. Quorum-sensing in Gram-negative bacteria. FEMS Microbiology Reviews. 2001; 25:365-404. DOI | PubMed
- Wielgoss S, Barrick JE, Tenaillon O, Wiser MJ, Dittmar WJ, Cruveiller S, Chane-Woon-Ming B, Médigue C, Lenski RE, Schneider D. Mutation rate dynamics in a bacterial population reflect tension between adaptation and genetic load. PNAS. 2013; 110:222-227. DOI | PubMed
- Wier AM, Nyholm SV, Mandel MJ, Massengo-Tiassé RP, Schaefer AL, Koroleva I, Splinter-Bondurant S, Brown B, Manzella L, Snir E, Almabrazi H, Scheetz TE, Bonaldo MF, Casavant TL, Soares MB, Cronan JE, Reed JL, Ruby EG, McFall-Ngai MJ. Transcriptional patterns in both host and bacterium underlie a daily rhythm of anatomical and metabolic change in a beneficial symbiosis. PNAS. 2010; 107:2259-2264. DOI | PubMed
- Williams DW, Jordan RP, Wei XQ, Alves CT, Wise MP, Wilson MJ, Lewis MA. Interactions of Candida albicans with host epithelial surfaces. Journal of Oral Microbiology. 2013; 5:22434-22438. DOI | PubMed
- Wilson K. Preparation of genomic DNA From bacteria. John Wiley & Sons, Inc: Hoboken, USA; 2001.
- Wiser MJ, Ribeck N, Lenski RE. Long-term dynamics of adaptation in asexual populations. Science. 2013; 342:1364-1367. DOI | PubMed
- Wiser MJ, Lenski RE. A comparison of methods to measure fitness in Escherichia coli. PLoS One. 2015; 10:e0126210-0126211. DOI | PubMed
- Wolfe AJ, Millikan DS, Campbell JM, Visick KL. Vibrio fischeri sigma54 controls motility, biofilm formation, luminescence, and colonization. Applied and Environmental Microbiology. 2004; 70:2520-2524. DOI | PubMed
- Wollenberg MS, Ruby EG. Population structure of Vibrio fischeri within the light organs of Euprymna scolopes squid from two Oahu (Hawaii) populations. Applied and Environmental Microbiology. 2009; 75:193-202. DOI | PubMed
- Ye L, Zheng X, Zheng H. Effect of sypQ gene on poly-N-acetylglucosamine biosynthesis in Vibrio parahaemolyticus and its role in infection process. Glycobiology. 2014; 24:351-358. DOI | PubMed
- Yildiz FH, Visick KL. Vibrio biofilms: so much the same yet so different. Trends in Microbiology. 2009; 17:109-118. DOI | PubMed
- Yip ES, Grublesky BT, Hussa EA, Visick KL, novel A. A novel, conserved cluster of genes promotes symbiotic colonization and sigma-dependent biofilm formation by Vibrio fischeri. Molecular Microbiology. 2005; 57:1485-1498. DOI | PubMed
- Yip ES, Geszvain K, DeLoney-Marino CR, Visick KL. The symbiosis regulator rscS controls the syp gene locus, biofilm formation and symbiotic aggregation by Vibrio fischeri. Molecular Microbiology. 2006; 62:1586-1600. DOI | PubMed
Fonte
Sabrina Pankey M, Foxall RL, Ster IM, Perry LA, Schuster BM, et al. () Host-selected mutations converging on a global regulator drive an adaptive leap towards symbiosis in bacteria. eLife 6e24414. https://doi.org/10.7554/eLife.24414





